用于液体活检工作流程的增强型甲基化检测
液体活检技术正在迅速改变早期癌症检测和监测的格局。通过提供一种微创方法,液体活检能够分析循环生物标志物,例如 cfDNA(无细胞脱氧核糖核酸),这些标志物携带有关癌症相关基因和表观遗传改变的宝贵信息。在这些生物标志物中,DNA 甲基化变化脱颖而出,成为识别和追踪各种癌症的一种颇具前景的途径,具有高特异性和灵敏度。
尽管具有前景,但由于其丰度有限且片段化模式独特,使用 cfDNA 面临独特的挑战。大多数 cfDNA 片段的长度约为 167 bp,反映了其核小体结合起点。这种片段化会导致:
- 较低的起始-终止多样性,产生更多独特的分子,称为重复测序片段
- 降低文库复杂性,会阻碍检测灵敏度
这些固有特性使传统的甲基化检测工作流程进行 cfDNA 分析时不太有效,因此需要定制的高级工具和技术来解决这些限制。
Twist 甲基化 UMI 接头通过唯一分子标签 (UMI) 实现精准的重复序列消除,并减少低多样性样本中的假重复序列识别,从而强化了 cfDNA 甲基化工作流程。设计可确保与 EM-Seq 方案的兼容性,保留了片段长度,最大限度地增加了可用数据,从而提高了靶标覆盖度和可重复性——使其成为高可信度甲基化研究的关键工具。
用于液体活检工作流程的增强型甲基化检测
液体活检技术正在迅速改变早期癌症检测和监测的格局。通过提供一种微创方法,液体活检能够分析循环生物标志物,例如 cfDNA(无细胞脱氧核糖核酸),这些标志物携带有关癌症相关基因和表观遗传改变的宝贵信息。在这些生物标志物中,DNA 甲基化变化脱颖而出,成为识别和追踪各种癌症的一种颇具前景的途径,具有高特异性和灵敏度。
尽管具有前景,但由于其丰度有限且片段化模式独特,使用 cfDNA 面临独特的挑战。大多数 cfDNA 片段的长度约为 167 bp,反映了其核小体结合起点。这种片段化会导致:
- 较低的起始-终止多样性,产生更多独特的分子,称为重复测序片段
- 降低文库复杂性,会阻碍检测灵敏度
这些固有特性使传统的甲基化检测工作流程进行 cfDNA 分析时不太有效,因此需要定制的高级工具和技术来解决这些限制。
Twist 甲基化 UMI 接头通过唯一分子标签 (UMI) 实现精准的重复序列消除,并减少低多样性样本中的假重复序列识别,从而强化了 cfDNA 甲基化工作流程。设计可确保与 EM-Seq 方案的兼容性,保留了片段长度,最大限度地增加了可用数据,从而提高了靶标覆盖度和可重复性——使其成为高可信度甲基化研究的关键工具。
将 UMI 纳入分析以提高重复序列分辨率
UMI 发现的重复识别将文库重复减少了 15% 以上,与标准位点重复识别相比,可增加文库复杂性 (25-35%) 和平均靶标覆盖度 (6-8%)。(图 1)
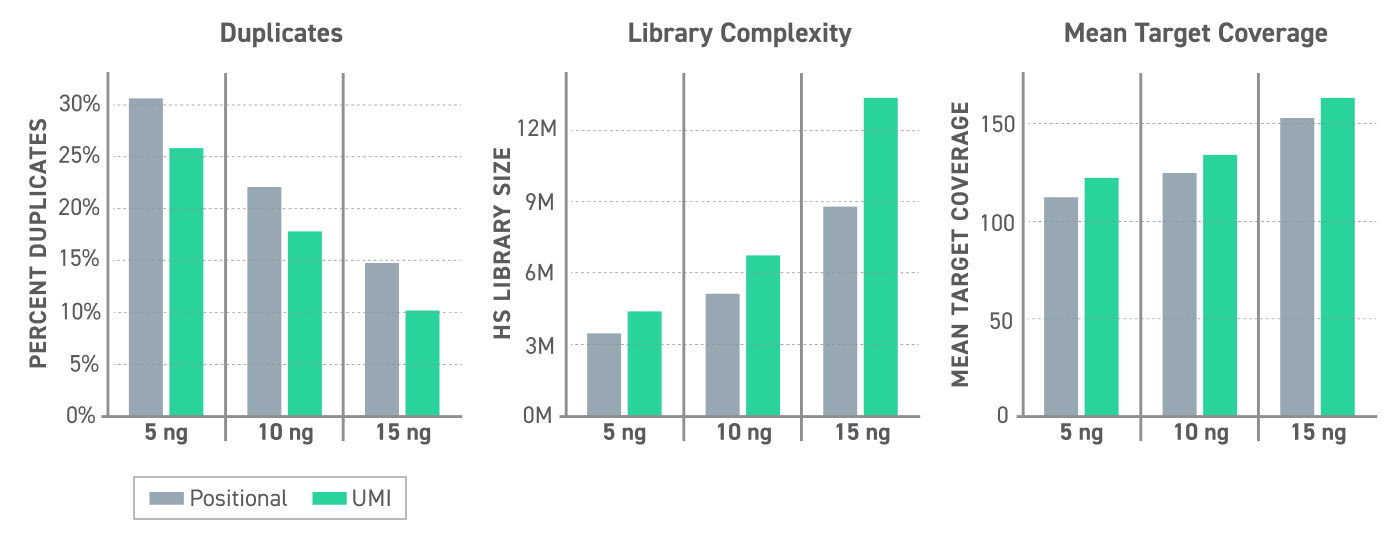
图 1. 在 NEBNext EM-Seq 文库制备后,使用可变 cfDNA 起始量( 5、10 或 15 ng cfDNA)的 UMI 性能生成带有 Twist 甲基化 UMI 接头的 EM-seq 转换文库。根据靶向甲基化测序方案,使用 Twist 联盟泛癌种甲基化组合 - 1.5 MB 捕获文库,并在 Illumina Nextseq 550上测序至 1000X 原始覆盖度。使用 GATK MarkDuplicates 工具(标准位点法)或使用带 GATK UmiAwareMarkDuplicatesWithMateCigar 工具的 UMI 识别重复。
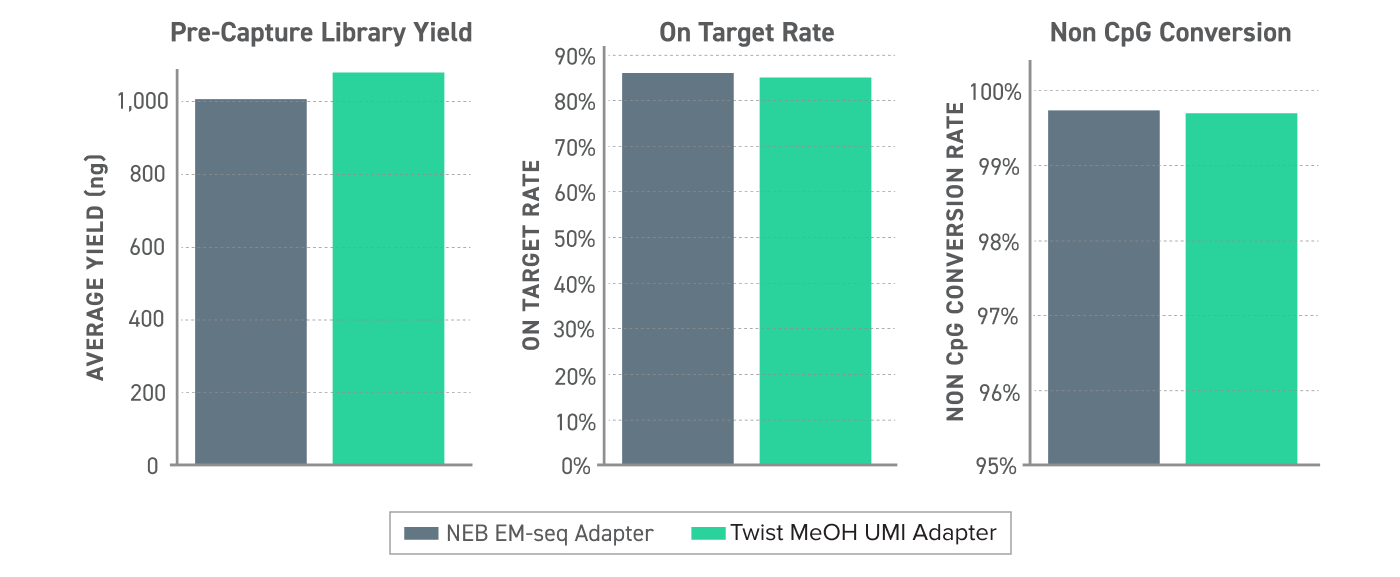
图 2. 在 Twist 甲基化检测工作流程中使用甲基化 UMI 性能( 10 ng 人 cfDNA)生成带有 NEB EMseq 接头或新 Twist 甲基化 UMI 接头的 EM-seq 转换文库。根据靶向甲基化测序方案,使用 Twist 联盟泛癌种甲基化组合 - 1.5 MB 捕获文库,并在 Illumina Nextseq 550上测序至 1000X 原始覆盖度。
生物信息学工作流程示例
UMI 信息可以整合到生物信息学流程中,以便在下游分析之前去除重复。

图 3. 处理 UMI 进行重复标记的分析工作流程汇总
处理原始片段,标记接头序列并提取 UMI 信息。然后使用 BWA-meth 将片段与参考基因组进行比对,并使用 UMI 信息进行重复标记。重复标记后,可以使用 GATK 收集 Picard 指标,并执行其他分析。
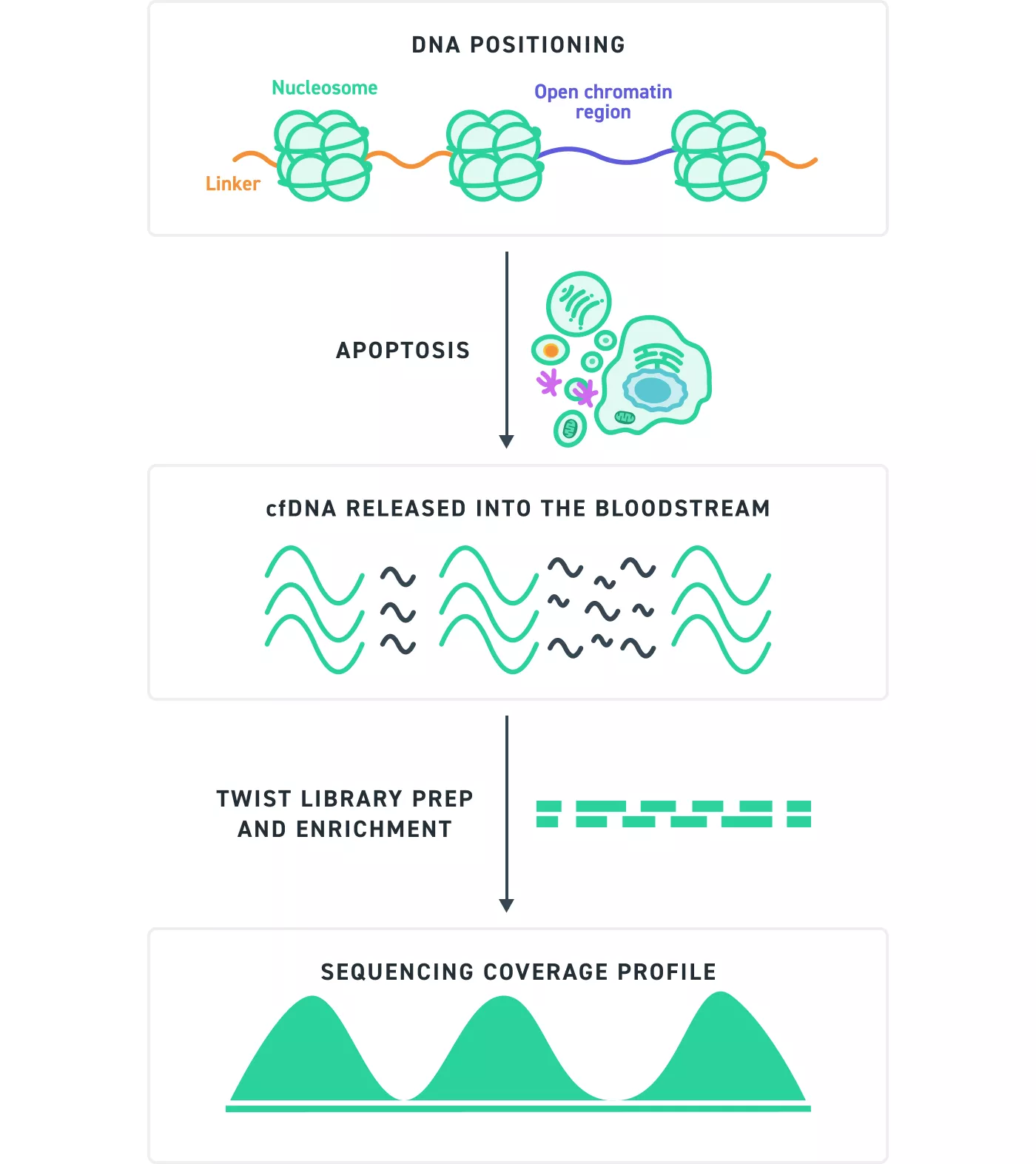
图 4. 细胞凋亡 cfDNA 片段至测序覆盖度示意图:
在细胞凋亡期间,无细胞 DNA (cfDNA) 被释放到血液中。虽然内切酶会随机地分割 DNA,但 DNA 被核糖体包裹的方式会引发位置偏差,导致有限的片段起始和终止位点的多样性。因此,不同的分子在测序数据中可能会出现重复。在文库制备过程中添加了唯一分子标识符 (UMI),以标记单个分子,从而准确区分真正的重复分子和单独的唯一分子。
将 UMI 纳入分析以提高重复序列分辨率
UMI 发现的重复识别将文库重复减少了 15% 以上,与标准位点重复识别相比,可增加文库复杂性 (25-35%) 和平均靶标覆盖度 (6-8%)。(图 1)

图 1. 在 NEBNext EM-Seq 文库制备后,使用可变 cfDNA 起始量( 5、10 或 15 ng cfDNA)的 UMI 性能生成带有 Twist 甲基化 UMI 接头的 EM-seq 转换文库。根据靶向甲基化测序方案,使用 Twist 联盟泛癌种甲基化组合 - 1.5 MB 捕获文库,并在 Illumina Nextseq 550上测序至 1000X 原始覆盖度。使用 GATK MarkDuplicates 工具(标准位点法)或使用带 GATK UmiAwareMarkDuplicatesWithMateCigar 工具的 UMI 识别重复。

图 2. 在 Twist 甲基化检测工作流程中使用甲基化 UMI 性能( 10 ng 人 cfDNA)生成带有 NEB EMseq 接头或新 Twist 甲基化 UMI 接头的 EM-seq 转换文库。根据靶向甲基化测序方案,使用 Twist 联盟泛癌种甲基化组合 - 1.5 MB 捕获文库,并在 Illumina Nextseq 550上测序至 1000X 原始覆盖度。
生物信息学工作流程示例
UMI 信息可以整合到生物信息学流程中,以便在下游分析之前去除重复。

图 3. 处理 UMI 进行重复标记的分析工作流程汇总
处理原始片段,标记接头序列并提取 UMI 信息。然后使用 BWA-meth 将片段与参考基因组进行比对,并使用 UMI 信息进行重复标记。重复标记后,可以使用 GATK 收集 Picard 指标,并执行其他分析。
图 4. 细胞凋亡 cfDNA 片段至测序覆盖度示意图:
在细胞凋亡期间,无细胞 DNA (cfDNA) 被释放到血液中。虽然内切酶会随机地分割 DNA,但 DNA 被核糖体包裹的方式会引发位置偏差,导致有限的片段起始和终止位点的多样性。因此,不同的分子在测序数据中可能会出现重复。在文库制备过程中添加了唯一分子标识符 (UMI),以标记单个分子,从而准确区分真正的重复分子和单独的唯一分子。

110830
产品:Twist 甲基化 UMI 接头 - TruSeq 兼容,96 个样本110830
产品:Twist 甲基化 UMI 接头 - TruSeq 兼容,96 个样本