解锁未开发的酶序列空间,实现最佳探索
我们的新型酶序列是从各种未培养微生物的单细胞基因组中确定的,并利用基于结构预测、基于结构的聚类和过滤的 ML 辅助模型,根据所需特性进行选择,这些序列在其他地方可能找不到。
转氨酶(TA)是一种催化胺基从胺基供体转移到酮或醛的酶。这种促进前手性酮立体选择性胺化的能力在制药生产中非常宝贵,因为手性胺作为活性药物成分(API)的构建模块的需求量很大。
我们已经确定并缩小了在各种极端条件下对多种底物保持活性的 TA 酶的子集,这些酶可用于制药生产过程。
我们的筛选试剂盒能够选择功能相关的序列,未来有可能将这些序列与下游酶工程资源相结合,为蛋白质工程提供更有效的途径。
筛选用酶的选择方法
bitBiome bit-GEM 数据库是可搜索且高度实用的微生物基因组序列的领先资源。bit-GEM 数据库利用自然界最强大的微生物资源,包括土壤、温泉、海水、矿场和其他极端环境中的样本,包含了通过 bitBiome 单细胞测序平台获得的众多基因序列。这种测序方法能够确保更深入的基因组覆盖,从而发现微生物“暗物质”。这些序列与公共领域中的序列相似度较低,从而赋予客户在该序列空间内自由操作的权限。



解锁未开发的酶序列空间,实现最佳探索
我们的新型酶序列是从各种未培养微生物的单细胞基因组中确定的,并利用基于结构预测、基于结构的聚类和过滤的 ML 辅助模型,根据所需特性进行选择,这些序列在其他地方可能找不到。
转氨酶(TA)是一种催化胺基从胺基供体转移到酮或醛的酶。这种促进前手性酮立体选择性胺化的能力在制药生产中非常宝贵,因为手性胺作为活性药物成分(API)的构建模块的需求量很大。
我们已经确定并缩小了在各种极端条件下对多种底物保持活性的 TA 酶的子集,这些酶可用于制药生产过程。
我们的筛选试剂盒能够选择功能相关的序列,未来有可能将这些序列与下游酶工程资源相结合,为蛋白质工程提供更有效的途径。
筛选用酶的选择方法
bitBiome bit-GEM 数据库是可搜索且高度实用的微生物基因组序列的领先资源。bit-GEM 数据库利用自然界最强大的微生物资源,包括土壤、温泉、海水、矿场和其他极端环境中的样本,包含了通过 bitBiome 单细胞测序平台获得的众多基因序列。这种测序方法能够确保更深入的基因组覆盖,从而发现微生物“暗物质”。这些序列与公共领域中的序列相似度较低,从而赋予客户在该序列空间内自由操作的权限。



筛选用酶的选择方法
bit-GEM 数据库利用大自然强大的微生物资源,包括来自土壤、温泉、海水、采矿场和其他极端条件等环境的样本,包含由 bitBiome 单细胞测序平台获取的大量基因序列。这种测序方法能够确保更深入的基因组覆盖,从而发现微生物“暗物质”。这些序列与公共领域中的序列相似度较低,因此在该序列空间内操作起来更加自由。该试剂盒中提供的 48 种酶的选择不仅得益于 Twist DNA 合成能力,还得益于生物信息学领域的合作,以优化功能序列空间。
我们的试剂盒中酶的选择方法
在 19 种底物中,共鉴定出 369 种活性转氨酶。其中 273 种酶在 10% 二甲基亚砜中具有活性,30 种酶在 60°C 下具有活性。最后,对酶的 pH 敏感性和选择性进行测试。
转氨酶筛选标准
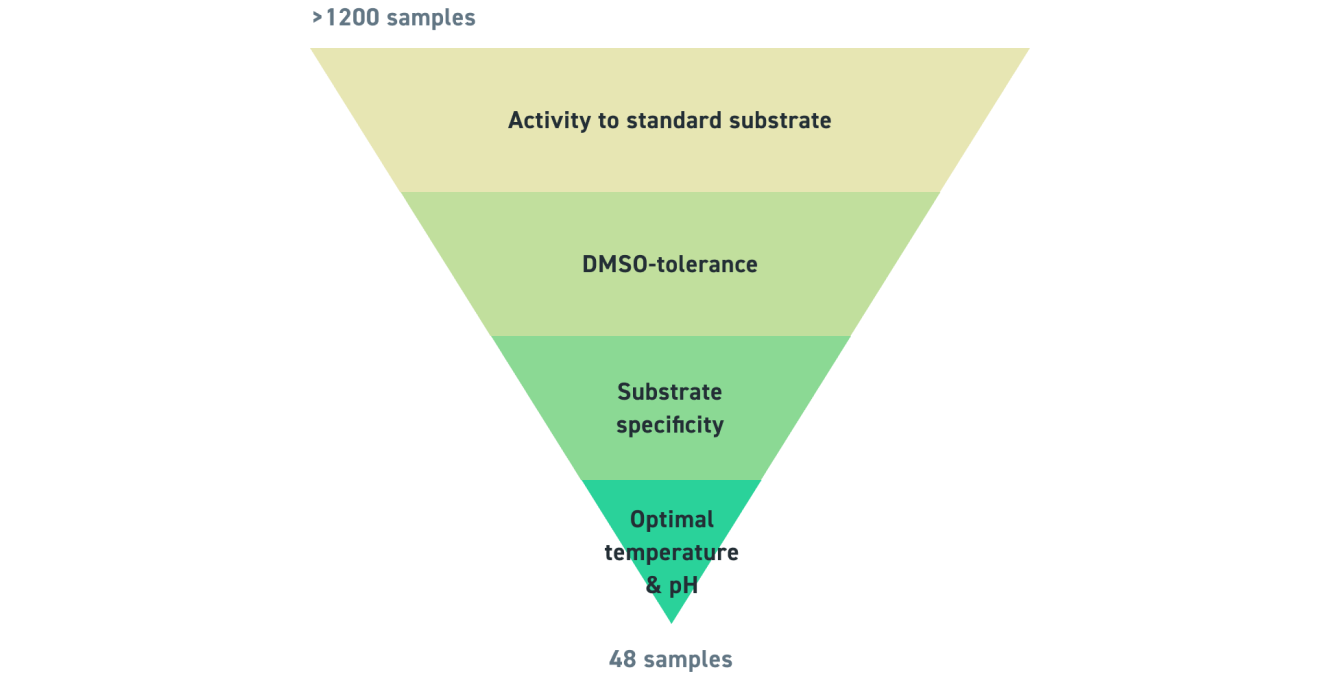
以下是针对上述不同条件(不同百分比的 DMSO、有机溶剂、温度和 pH 值)下各种底物所测试的酶的分解。
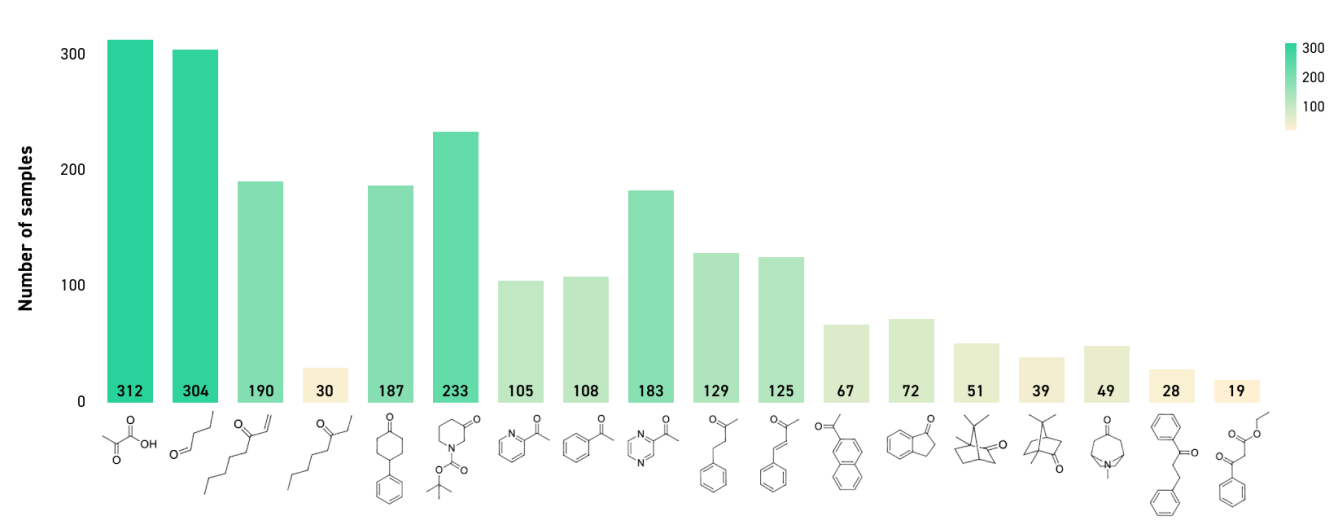
筛选用酶的选择方法
bit-GEM 数据库利用大自然强大的微生物资源,包括来自土壤、温泉、海水、采矿场和其他极端条件等环境的样本,包含由 bitBiome 单细胞测序平台获取的大量基因序列。这种测序方法能够确保更深入的基因组覆盖,从而发现微生物“暗物质”。这些序列与公共领域中的序列相似度较低,因此在该序列空间内操作起来更加自由。该试剂盒中提供的 48 种酶的选择不仅得益于 Twist DNA 合成能力,还得益于生物信息学领域的合作,以优化功能序列空间。
我们的试剂盒中酶的选择方法
在 19 种底物中,共鉴定出 369 种活性转氨酶。其中 273 种酶在 10% 二甲基亚砜中具有活性,30 种酶在 60°C 下具有活性。最后,对酶的 pH 敏感性和选择性进行测试。
转氨酶筛选标准

以下是针对上述不同条件(不同百分比的 DMSO、有机溶剂、温度和 pH 值)下各种底物所测试的酶的分解。

