Desvele los espacios de las secuencias enzimáticas no explotados para permitir una exploración óptima
Nuestras nuevas secuencias enzimáticas se han identificado a partir de diferentes genomas unicelulares microbianos no cultivados y se han seleccionado en función de unas propiedades deseables mediante modelos asistidos por aprendizaje automático de acuerdo con la predicción de la estructura, el agrupamiento basado en estructuras y el filtrado. Es probable que no encuentre estas secuencias en ningún otro lugar.
Las transaminasas (TA) son enzimas que catalizan la transferencia de un grupo amino del donante del grupo amino a una cetona o aldehído. Esta capacidad para facilitar la aminación estereoselectiva de cetonas proquirales es valiosa en la fabricación farmacéutica, en la que la demanda de aminos quirales como bloques de construcción para ingredientes farmacéuticos activos (IFA) es alta.
Hemos identificado y estrechado un subconjunto de enzimas TA que mantienen su actividad en diferentes condiciones extremas en numerosos sustratos, lo que es útil para el proceso de fabricación farmacéutica.
Nuestro kit de cribado permite la selección de secuencias funcionalmente pertinentes con la posibilidad futura de combinar estos recursos de ingeniería enzimática posteriores para proporcionar una vía más eficiente de ingeniería de proteínas.
Cómo se seleccionan las enzimas para el cribado
La base de datos bitBiome bit-GEM es el recurso líder para secuencias genómicas de microbios muy utilizables y que se pueden buscar. Basada en los recursos microbianos más potentes de la naturaleza, incluidas las muestras de entornos como el suelo, las fuentes termales, el agua marina, las minas y otras condiciones extremas, la base de datos bit-GEM contiene una impresionante matriz de secuencias genéticas adquiridas mediante la plataforma de secuenciación unicelular bitBiome. Este método de secuenciación garantiza una cobertura genómica más exhaustiva, lo que permite el descubrimiento de la “materia oscura” microbiana. Estas secuencias se parecen poco a las del dominio público, lo que da al cliente la libertad de trabajar en ese espacio de la secuencia.



Desvele los espacios de las secuencias enzimáticas no explotados para permitir una exploración óptima
Nuestras nuevas secuencias enzimáticas se han identificado a partir de diferentes genomas unicelulares microbianos no cultivados y se han seleccionado en función de unas propiedades deseables mediante modelos asistidos por aprendizaje automático de acuerdo con la predicción de la estructura, el agrupamiento basado en estructuras y el filtrado. Es probable que no encuentre estas secuencias en ningún otro lugar.
Las transaminasas (TA) son enzimas que catalizan la transferencia de un grupo amino del donante del grupo amino a una cetona o aldehído. Esta capacidad para facilitar la aminación estereoselectiva de cetonas proquirales es valiosa en la fabricación farmacéutica, en la que la demanda de aminos quirales como bloques de construcción para ingredientes farmacéuticos activos (IFA) es alta.
Hemos identificado y estrechado un subconjunto de enzimas TA que mantienen su actividad en diferentes condiciones extremas en numerosos sustratos, lo que es útil para el proceso de fabricación farmacéutica.
Nuestro kit de cribado permite la selección de secuencias funcionalmente pertinentes con la posibilidad futura de combinar estos recursos de ingeniería enzimática posteriores para proporcionar una vía más eficiente de ingeniería de proteínas.
Cómo se seleccionan las enzimas para el cribado
La base de datos bitBiome bit-GEM es el recurso líder para secuencias genómicas de microbios muy utilizables y que se pueden buscar. Basada en los recursos microbianos más potentes de la naturaleza, incluidas las muestras de entornos como el suelo, las fuentes termales, el agua marina, las minas y otras condiciones extremas, la base de datos bit-GEM contiene una impresionante matriz de secuencias genéticas adquiridas mediante la plataforma de secuenciación unicelular bitBiome. Este método de secuenciación garantiza una cobertura genómica más exhaustiva, lo que permite el descubrimiento de la “materia oscura” microbiana. Estas secuencias se parecen poco a las del dominio público, lo que da al cliente la libertad de trabajar en ese espacio de la secuencia.



Cómo se seleccionan las enzimas para el cribado
Basada en los potentes recursos microbianos de la naturaleza, incluidas las muestras de entornos como el suelo, las fuentes termales, el agua marina, las minas y otras condiciones extremas, la base de datos bit-GEM contiene una impresionante matriz de secuencias genéticas adquiridas mediante la plataforma de secuenciación unicelular bitBiome. Este método de secuenciación garantiza una cobertura genómica más exhaustiva, lo que permite el descubrimiento de la “materia oscura” microbiana. Estas secuencias se parecen poco a las del dominio público, lo que proporciona mayor libertad de trabajar en ese espacio de la secuencia. La selección de las 48 enzimas proporcionadas en este kit estuvo potenciada por las capacidades de síntesis de ADN Twist y por un esfuerzo bioinformático colaborativo para refinar el espacio funcional de la secuencia.
Cómo se seleccionaron las enzimas para nuestro kit
Se identificaron un total de 369 transaminasas activas con 19 sustratos. Se determinó que 273 enzimas eran activas en DMSO al 10 % con 30 activas a 60 °C. Por último, las enzimas se probaron en cuanto a sensibilidad al pH y selectividad.
Criterios para el cribado de transaminasas
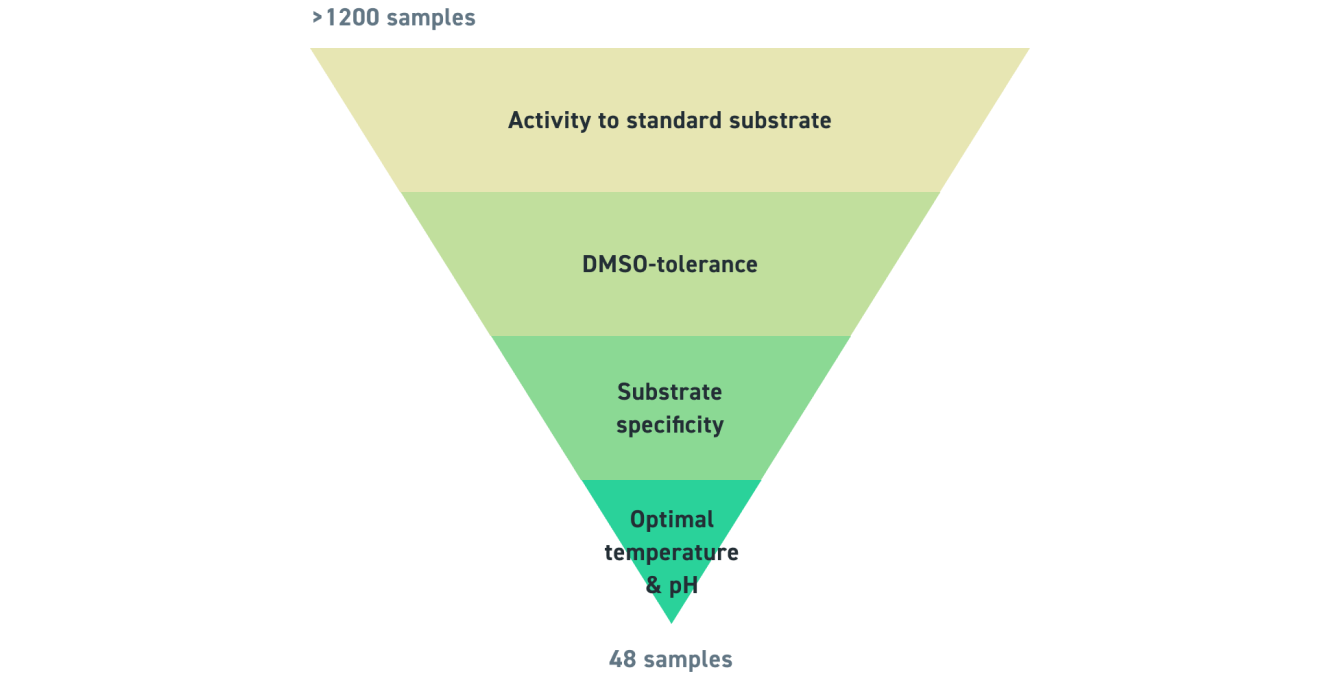
A continuación, se proporciona un resumen de las enzimas probadas frente a varios sustratos en las diferentes condiciones anteriormente descritas (diferentes porcentajes de DMSO, disolventes orgánicos, temperaturas y pH).
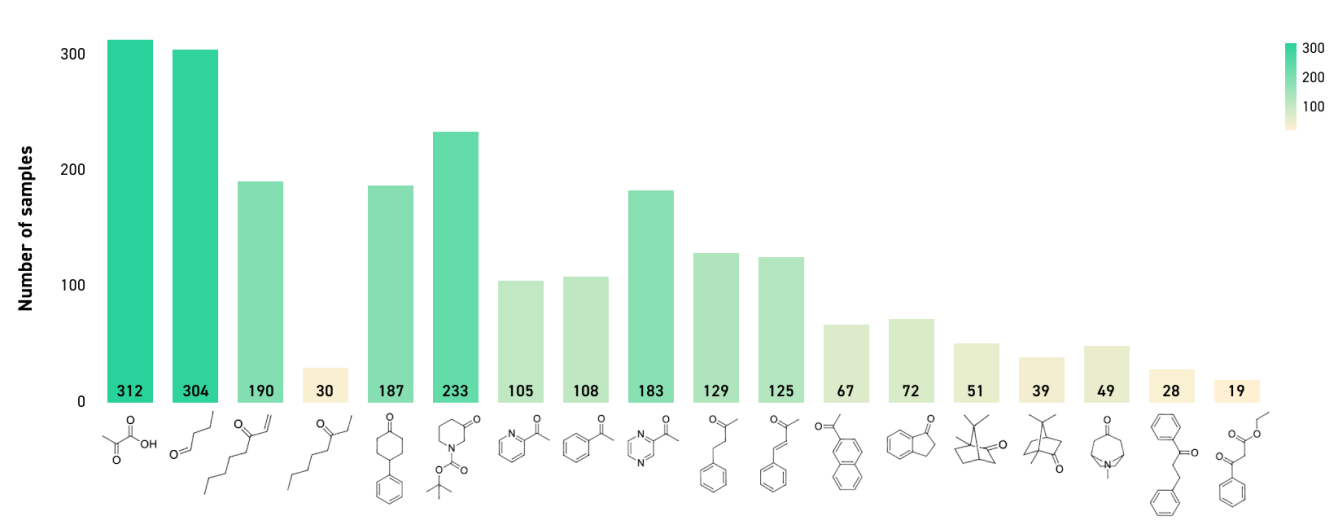
Cómo se seleccionan las enzimas para el cribado
Basada en los potentes recursos microbianos de la naturaleza, incluidas las muestras de entornos como el suelo, las fuentes termales, el agua marina, las minas y otras condiciones extremas, la base de datos bit-GEM contiene una impresionante matriz de secuencias genéticas adquiridas mediante la plataforma de secuenciación unicelular bitBiome. Este método de secuenciación garantiza una cobertura genómica más exhaustiva, lo que permite el descubrimiento de la “materia oscura” microbiana. Estas secuencias se parecen poco a las del dominio público, lo que proporciona mayor libertad de trabajar en ese espacio de la secuencia. La selección de las 48 enzimas proporcionadas en este kit estuvo potenciada por las capacidades de síntesis de ADN Twist y por un esfuerzo bioinformático colaborativo para refinar el espacio funcional de la secuencia.
Cómo se seleccionaron las enzimas para nuestro kit
Se identificaron un total de 369 transaminasas activas con 19 sustratos. Se determinó que 273 enzimas eran activas en DMSO al 10 % con 30 activas a 60 °C. Por último, las enzimas se probaron en cuanto a sensibilidad al pH y selectividad.
Criterios para el cribado de transaminasas

A continuación, se proporciona un resumen de las enzimas probadas frente a varios sustratos en las diferentes condiciones anteriormente descritas (diferentes porcentajes de DMSO, disolventes orgánicos, temperaturas y pH).

