提高农作物产量。消除携带疟疾的蚊子。纠正既往无法治愈的遗传疾病。CRISPR-Cas9 作为基因组剪刀首次亮相已有七年,“Cas9 可以做什么?”已经变成一个很难回答的问题。毕竟,什么是 Cas9 不能做的?取消其核酸酶活性,Cas9 成为用于转录控制、表观遗传修改、基因组标记和碱基编辑等应用的模块化平台。Cas9 在生物技术上的巨大成功已经努力将 CRISPR 工具箱扩展到包括其他 CRISPR 核酸酶,从而为下一代 CRISPR 应用提供支持。
CRISPR 相关 (Cas) 酶存在于大多数古细菌和许多细菌中,是它们的适应性免疫的组成部分。2015 年,在 Cas9 首次被重新用作基因编辑器仅几年之后,Cas 系统根据其独特特征被分为 2 个类别、5 种类型和 16 种亚型。如今,分类系统包括 2 个类别、6 个类型和 33 个子类型(Makarova 等人,2020 年)。这种变化在一定程度上是由对新的 2 类系统的追捧所推动的,该系统可用于 Cas9 基因组工程。其中包括 DNA 编辑器 Cas12aa (Cpf1),和以 Cas13 为基础的 RNA 编辑器。
Argonautes 是一组与 CRISPR-Cas 系统截然不同的核酸引导蛋白,在 2016 年遭到了负面报道,当时几个独立团体未能复制其所谓的基因编辑能力的说法后,一篇引人注目的论文被撤回(Gao 等人,2016 年)。尽管如此,Argonautes 还是值得在这里被提及,仅仅是希望指出它们的一个应用比 CRISPR 系统更适合。
我们在下面重点介绍了 Twist Bioscience 专家编辑的 CRISPR(和 Argonaute)技术中一些最具变革性的开发。
Cas12 简化了组合筛选
许多生物学过程(例如肿瘤转移)涉及复杂的遗传相互作用,而使用单一淘汰法是无法完全解决这些相互作用的。尽管 Cas9 是单一淘汰法筛选应用的金标准,但由于其次优的复用能力,Cas9 在组合筛选中的应用受到了阻碍。多个独立团体最近对 V 型 CRISPR 系统 Cas12aa (Cpf1) 进行了优化,以提供 Cas9 的改进替代方案,以进行大规模组合基因编辑(DeWeirdt 等人,2020 年; Gier 等人,2020 年)。
是什么让 Cas12a 在组合筛选中比 Cas9 更好?Cas12a 可以处理不带 RNase III 和反式激活 RNA (tracrRNA) 的 CRISPR RNA (crRNA) 阵列,这是 Cas9 完成相同任务所需的组件。由于 Cas9 本身无法处理 crRNA 阵列,因此 Cas9 引导通常必须在单独的表达盒中表达,每个表达盒一个。多重基因编辑的 Cas9 引导中的重复元素使其在慢病毒递送,PCR 和深度测序过程中易于重组和解偶联。这些因素会使文库设计、克隆和分析复杂化。
使用 Cas12a,可以合成包含多个 crRNA 的寡核苷酸,并将其轻松克隆到慢病毒载体中,以进行组合筛选。此外,多重基因编辑的 Cas12a 引导载体中的重复元素相对较短,且对序列修改的耐受性更高,使多重基因编辑的 Cas12a 引导在整个筛选过程中不易重组和解偶联(DeWeirdt 等人,2020 年;Gier 等人,2020 年)。Twist Bioscience 的 300mer 寡核苷酸可同时编码 3-4 个引导。
尽管 Cas12a 是出色的多重基因编辑器,但与 Cas9 相比,它的某些局限性包括限制性更强的前间区序列邻近基序 (PAM) 特异性 (5’-TTTV) 及更低的突变效率。这些参数已通过氨基酸球菌属(AsCas12a;Kleinstiver 等人,2019 年)的 Cas12a 的结构引导蛋白质工程改造得到了改善,最终产生了一种名为增强型 AsCas12a (enAsCas12a) 的修改酶,该酶在前间区序列邻近基序识别重要的残基中包含多个点突变。除了扩展其 PAM 特异性(现在包含 TTYN、VTTV、和 TRTV 等)之外,偶然间发现 enAsCas12a 的编辑效率比 AsCas12a 平均提高了两倍。通过添加多个核定位序列,并阐明靶内和靶外 crRNA 规则,AsCas12a 的效率得到了进一步提高(DeWeirdt 等人,2020 年;Gier 等人,2020 年)。
VI 型 CRISPR 系统将目光投向 RNA
使用短夹的 RNA (shRNA) 的 RNA 干扰 (RNAi) 是一种完善的基因抑制策略,它选择了一种内源性 RNA 处理通路来降解细胞信使 RNA。但是,RNAi 受脱靶效应和抑制效率不一的困扰。特异性问题已通过 CRISPR 干扰解决,该干扰是一种抑制方法,使用了与转录阻遏域 KRAB 融合的核酸酶失效的 Cas9。即使如此,在基础基因表达高或 shRNA 夹处理不良的情况下,CRISPR 干扰和 RNAi 有时仍无法稳健地抑制其靶标。
输入 RfxCas13d (CasRx),这是一种 RNA 引导,针对 RNA 的 CRISPR 核酸酶。CasRx 可能不是第一个发现的 RNA 编辑器,但与其他 Cas13 系统(LwaCas13a 和 PspCas13b)和现有方法并排比较表明,CasRx 可以诱导许多基因中最实质性的抑制,并具有很高的启动特异性。而且,核酸酶失效的变体 dCasRx 可以靶向前 mRNA 的顺式元素,以调节其拼接。为了突出此功能,Konermann 等人(2018 年)将 dCasRx 定位到内含接头的消声器中,以在额颞叶痴呆神经元模型中纠正 Tau 编码 mRNA 的病理性拼接。
由于它们偏好单链 RNA,因此一般而言,Cas13 系统的靶向可能会受到 RNA 二级结构的阻碍(Abudayyeh 等人,2017 年)。然而,它们通过靶向基因组上的 RNA 引起瞬时变化的能力,以及它们缺少序列限制,使得它们成为用于实验和治疗应用的有吸引力的转录组编辑工具。
1 类 CRISPR-Cas 系统使 CRISPR 工具套件多元化
随着 2 类 CRISPR-Cas 系统(例如 Cas9、Cas12s 和 Cas13s)等继续在基因组工程领域占据主导地位,1 类 CRISPR-Cas 系统因其独特的裂解特性和多蛋白组成而开始受到瞩目。其中最常见的是 I 型系统,该系统将 Cas3 解旋酶与辅助 Cas 蛋白复合物(称为 CRISPR 相关复合物)相结合,用于抗病毒防御或“Cascade”。
如果 Cas9 和 Cas12a 是基因组剪刀,Cas3 则是基因组打碎机。Cas3 并非产生像 Cas9 这样的小插入缺失,而是在目标基因组位点的上游引起一系列大的基因组缺失(长度介于 0.5 到 100 kb 之间(Dolan 等人,2019 年)。此功能有望实现新颖的高通量筛选应用,由此,与 2 类 CRISPR 系统相比,可以使用较少的引导生成缺失突变体的文库。
The Cascade complex alone also provides a modular platform for RNA-guided transcriptional activation and repression. Site-directed repression can be achieved using crRNAs in cas3-deleted E. coli (Luo et al., 2015; Rath et al., 2015) or in mammalian cells expressing exogenous Cascade proteins (Pickar-Oliver et al., 2019; Chen et al., 2020).
For gene activation, transcriptional activator domains need to be fused to one or more members of Cascade. Chen et al. (2020) obtained the best results when the synthetic VPR (VP64-p65-Rta) activator domain was fused to Csy3, the backbone component of Pseudomonas aeruginosa Cascade (PaeCascade) that interacts with the crRNA. Because multiple Csy3 proteins participate in the formation of the PaeCascade complex, multiple transcriptional effector domains can be recruited to a single Cascade complex. This strategy does not necessarily work for other class 1 systems, however, as the fusion of transcriptional effectors to Cas7 of EcoCascade (its Csy3 equivalent) failed to elevate transcription at targeted loci (Pickar-Oliver et al., 2019).
Cascade 的多蛋白性质可在为转录控制模块化设备中派上用场,但是这也意味着多种效应蛋白必须被共同表达。这可能会妨碍在某些应用中 1 类系统的使用。
奇怪的 CRISPR 基因座揭示了 CRISPR 相关的转座酶
Most CRISPR loci contain genes for site-directed nucleases. For example, CRISPR loci encoding type I systems typically contain cas3, which encodes the endonuclease effector of these Cas systems. Recent bioinformatic analyses, however, revealed CRISPR loci containing Tn7-like transposon genes instead of Cas endonuclease genes. From these loci, two CRISPR-associated transposon systems have been characterized: the class 2 type V-K effector Cas12k (Strecker et al., 2019) and the class 1 type I-F Cascade complex (Halpin-Healy et al., 2020). Instead of cleaving DNA, these Cas systems can insert large segments of DNA into the genome in a site-directed and nuclease-independent fashion.
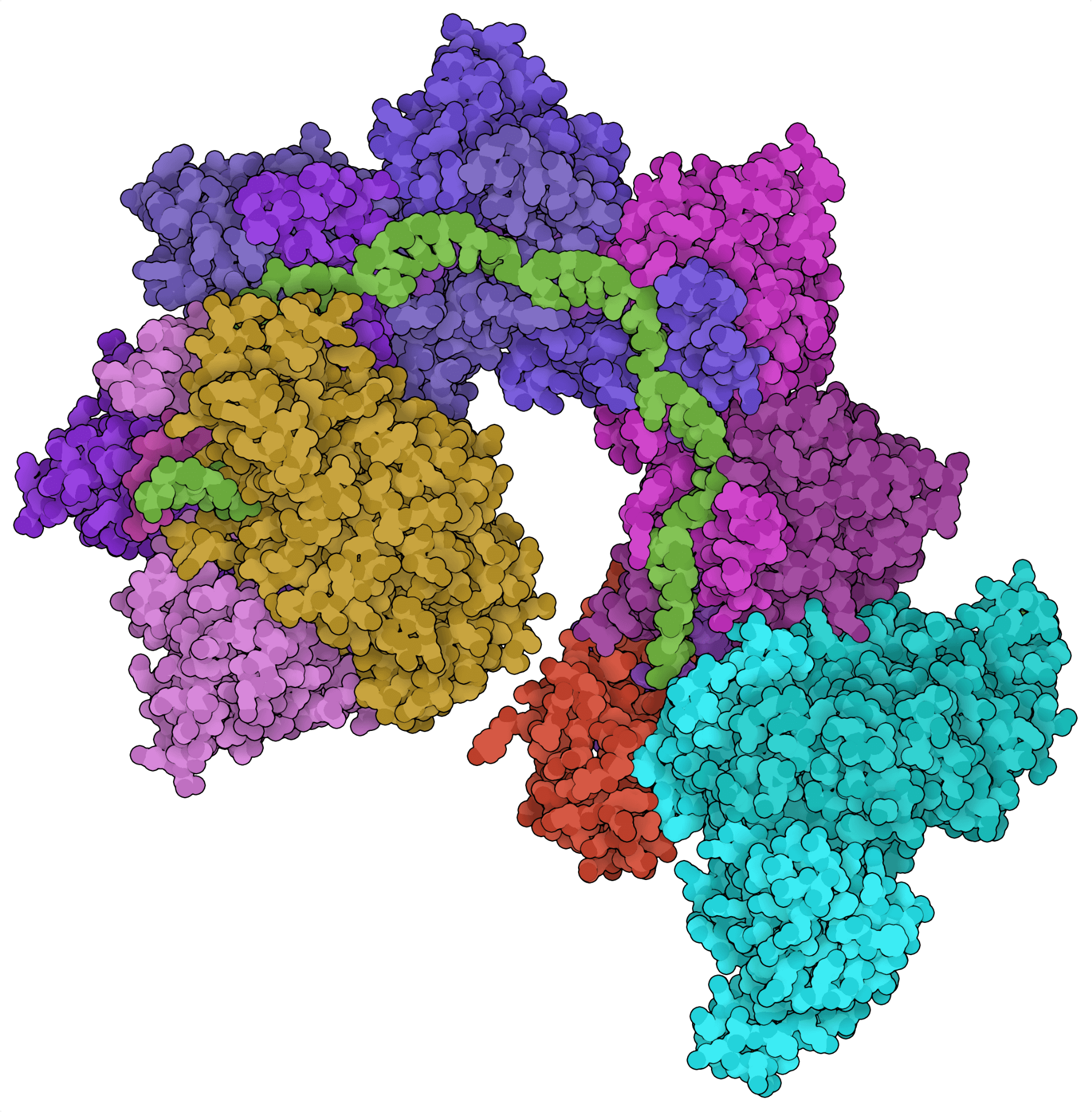
传统的以 CRISPR 为基础的基因敲入方法依赖于定点 DNA 断裂后同源性修复,这是一个非常低效的过程。新识别的 CRISPR 相关转座子通过允许大 DNA 片段的转座,提供了一种可能更有效的解决方法。CRISPR 相关的转座酶可用于替代有缺陷的外显子或产生嵌合抗原受体 (CAR) T 细胞。虽然前景尚佳,但这些系统的转座酶活性尚未在真核细胞中得到证实。
Argonautes 促进 DNA 数据存储
Sometimes the best alternative to Cas9 is not a CRISPR system at all. This is true for nick-based DNA data storage, which requires a nicking enzyme to encode information in native DNA. DNA has been proposed as an alternative to current silicon-based storage media because of its relatively high durability and enormous storage density. To further push the limits of the information storage density of DNA, researchers have recently developed a nick-based approach (Tabatabaei et al., 2020).
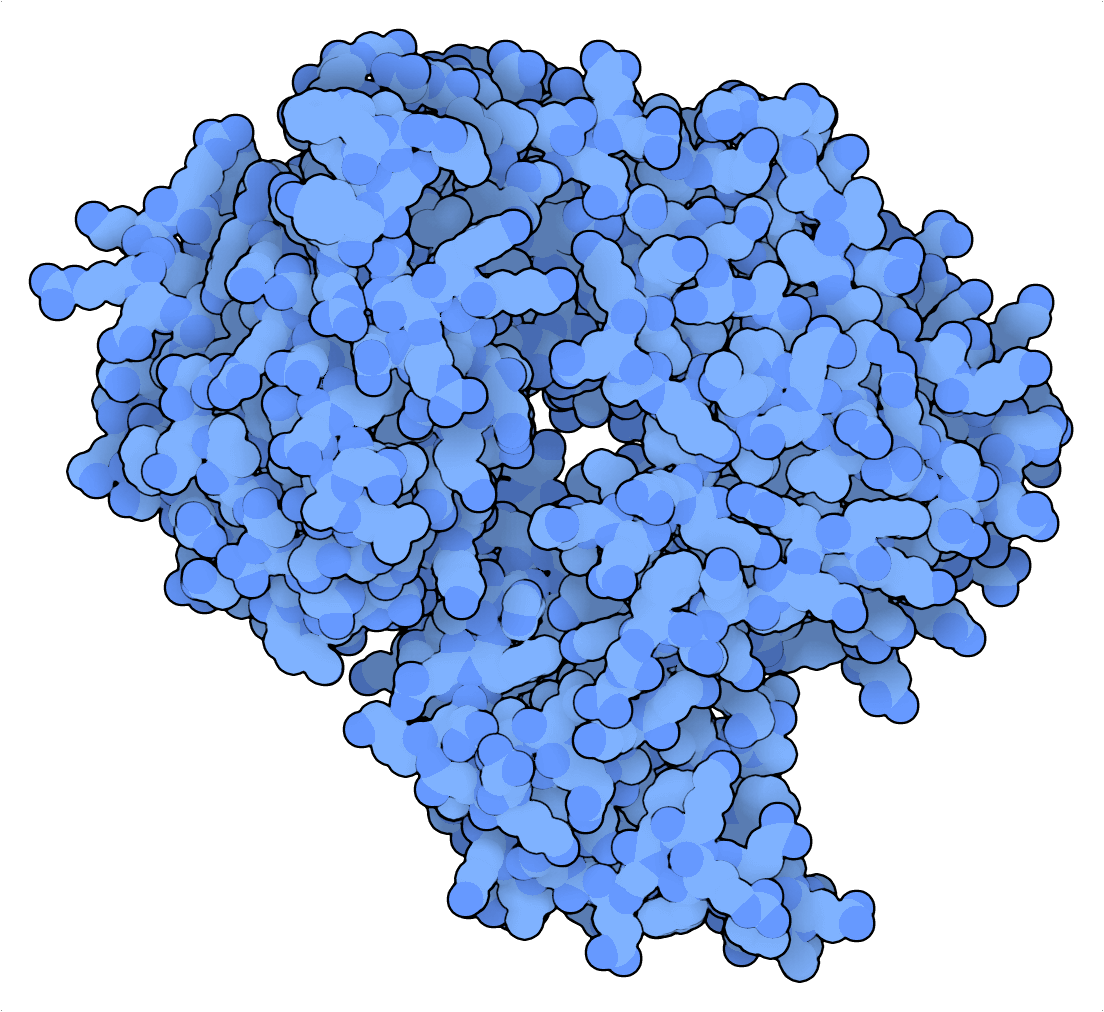
Tabatabaei 等人(2020 年)利用 PfArgo 的有利切割特性,创建了一种将 dsDNA 链变成“打孔卡”的 DNA 写入设备。在这里,二进制信息以切割的形式在定位上编码,可以通过使 DNA 单链片段变性来读取。然后可以通过下一代测序或纳米孔测序对这些片段进行测序,并反映到参考打孔卡以检索编码信息。Tabatabaei 等人(2020 年)突出了此策略出色的存储密度,并将林肯纪念堂(Lincoln Memorial)的 329 千字节(KB)JPEG 文件压缩为14 KB 的 DNA 文件!
对于以切割为基础的 DNA 数据存储,相比较焦磷酸焦球菌 (PfAgo),Cas9 切割酶的切割性质较弱。前者是一种单周转酶,依靠不稳定且受限于 PAM 的 RNA-DNA 相互作用进行靶向,而后者可以使用不受任何特定靶向序列限制的短 DNA 引导,在每种酶中诱导数百个切割。
这些只是 CRISPR 和 Argonaute 系列中核酸引导的蛋白质使之成为可能的其中一些应用。要了解有关如何扩展您的 CRISPR 工具箱的更多信息,请与团队成员联系。
Protein illustrations were generated from PDB structures using Illustrate, a non-photorealistic biomolecular illustration tool (Goodsell et al., 2019).
参考文献
Abudayyeh OO, Gootenberg JS, Essletzbichler P, Han S, Joung J, Belanto JJ, Verdine V, Cox DBT, Kellner MJ, Regev A, Lander ES, Voytas DF, Ting AY, Zhang F (2017) RNA targeting with CRISPR-Cas13. Nature 550:280-284.
Chen Y, Liu J, Zhi S, Zheng Q, Ma W, Huang J, Liu Y, Liu D, Liang P, Songyang Z (2020) Repurposing type I-F CRISPR-Cas system as a transcriptional activation tool in human cells. Nat Commun 11:3136.
DeWeirdt, PC, Sangree, AK, Hanna, RE et al. (2020) Genetic screens in isogenic mammalian cell lines without single cell cloning. Nat Commun 11, 752.
Dolan AE, Hou Z, Xiao Y, Gramelspacher MJ, Heo J, Howden SE, Freddolino PL, Ke A, Zhang Y (2019) Introducing a Spectrum of Long-Range Genomic Deletions in Human Embryonic Stem Cells Using Type I CRISPR-Cas. Mol Cell 74:936-950 e935.
Gao F, Shen XZ, Jiang F, Wu Y, Han C (2016) DNA-guided genome editing using the Natronobacterium gregoryi Argonaute. Nat Biotechnol 34:768-773.
Gier, RA, Budinich, KA, Evitt, NH et al. (2020) High-performance CRISPR-Cas12a genome editing for combinatorial genetic screening. Nat Commun 11, 3455
Goodsell, DS, Autin, L, Olson, AJ (2019) Illustrate: Software for Biomolecular Illustration. Structure 27:1716-1720.e1
Halpin-Healy TS, Klompe SE, Sternberg SH, Fernandez IS (2020) Structural basis of DNA targeting by a transposon-encoded CRISPR-Cas system. Nature 577:271-274.
Kleinstiver BP, Sousa AA, Walton RT, Tak YE, Hsu JY, Clement K, Welch MM, Horng JE, Malagon-Lopez J, Scarfo I, Maus MV, Pinello L, Aryee MJ, Joung JK (2019) Engineered CRISPR-Cas12a variants with increased activities and improved targeting ranges for gene, epigenetic and base editing. Nat Biotechnol 37:276-282.
Konermann S, Lotfy P, Brideau NJ, Oki J, Shokhirev MN, Hsu PD (2018) Transcriptome Engineering with RNA-Targeting Type VI-D CRISPR Effectors. Cell 173:665-676 e614.
Luo ML, Mullis AS, Leenay RT, Beisel CL (2015) Repurposing endogenous type I CRISPR-Cas systems for programmable gene repression. Nucleic Acids Res 43:674-681.
Makarova KS et al. (2020) Evolutionary classification of CRISPR-Cas systems: a burst of class 2 and derived variants. Nat Rev Microbiol 18:67-83.
Pickar-Oliver A, Black JB, Lewis MM, Mutchnick KJ, Klann TS, Gilcrest KA, Sitton MJ, Nelson CE, Barrera A, Bartelt LC, Reddy TE, Beisel CL, Barrangou R, Gersbach CA (2019) Targeted transcriptional modulation with type I CRISPR-Cas systems in human cells. Nat Biotechnol 37:1493-1501.
Rath D, Amlinger L, Hoekzema M, Devulapally PR, Lundgren M (2015) Efficient programmable gene silencing by Cascade. Nucleic Acids Res 43:237-246.
Strecker J, Ladha A, Gardner Z, Schmid-Burgk JL, Makarova KS, Koonin EV, Zhang F (2019) RNA-guided DNA insertion with CRISPR-associated transposases. Science 365:48-53.
Tabatabaei SK, Wang B, Athreya NBM, Enghiad B, Hernandez AG, Fields CJ, Leburton JP, Soloveichik D, Zhao H, Milenkovic O (2020) DNA punch cards for storing data on native DNA sequences via enzymatic nicking. Nat Commun 11:1742.
您怎么认为?
喜欢
不喜欢
喜爱
惊讶
有兴趣
