酵素による断片化の強化
Twist Library Preparation Enzymatic Fragmentation (EF) Kit 2.0 は、より効率的なシーケンス解析パイプラインを実現し、より正確な次世代シーケンシング (NGS) の結果を得るために構築されています。従来の Twist Library Preparation EF Kit 1.0 と比較して、この最新キットにはいくつかの利点があります。新たな断片化用・ライゲーション用モジュールに加えて、この新しいキットには Equinox Library Prep Amp Mix が含まれています。この酵素によるホットスタート技術を用いることで、エラー率が低く*、サンプル量が少ない場合でも効率良く多くの収量が得られるため、FFPE のような高い効率と収量が求められるサンプルに最適です。
このキットの合理的なワークフローによって、ライブラリ調製ステップが一つのチューブ内で完結し、低キメラ率および Twist の高均一性ターゲットエンリッチメントワークフローとあわせて、より高品質なライブラリ調製とシーケンス解析結果につながります。
* Twist Enzymatic Fragmentation 1.0 との比較
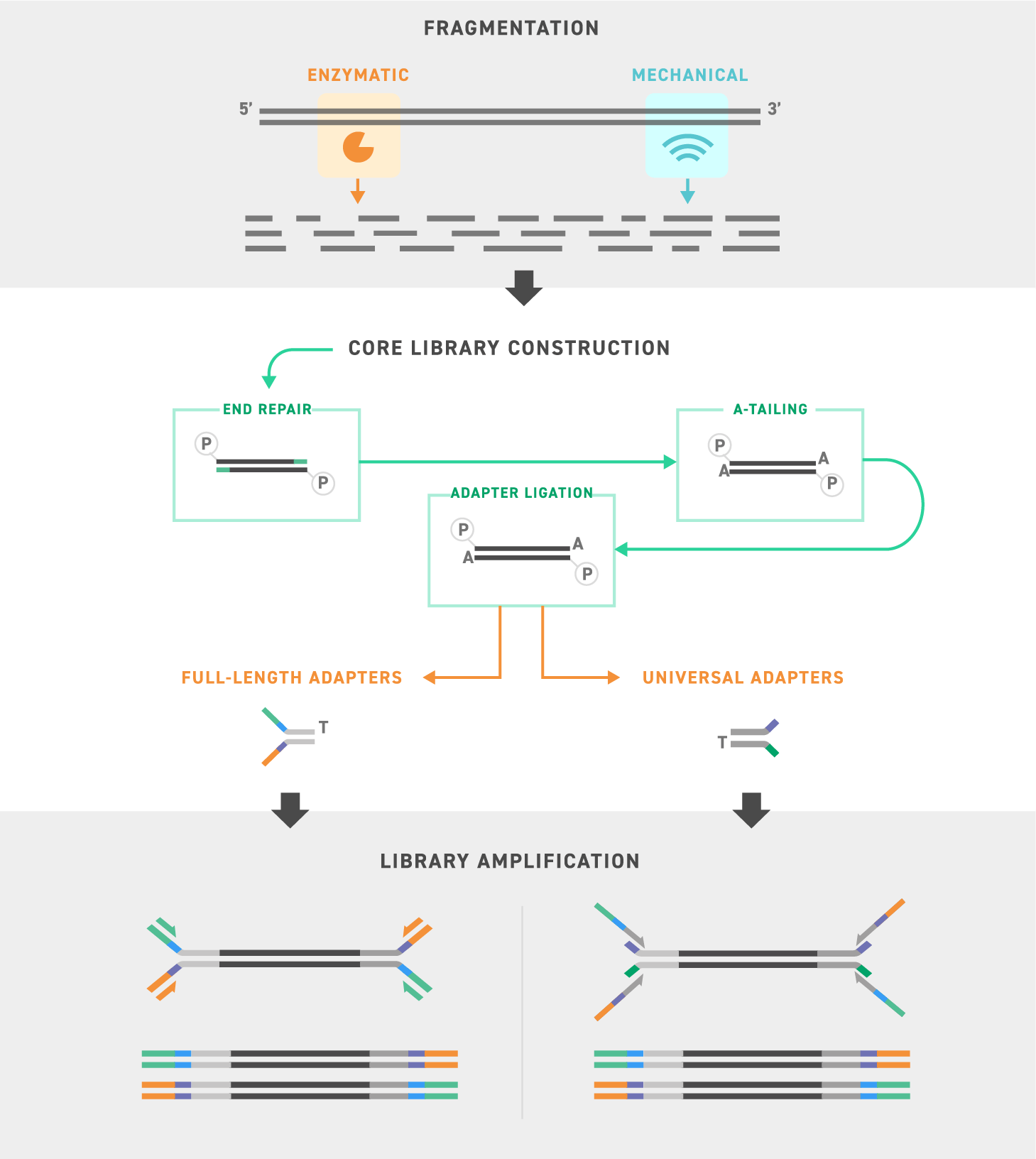
ライブラリ調製ワークフロー
Twist Library Preparation EF Kit 2.0 には、末端修復、dA-テーリング、アダプターライゲーションおよびライブラリ増幅に必要な試薬が含まれています。また、ゲノム DNAサンプル断片化用の酵素も含まれており、インサートサイズを調節することができます。コアライブラリの構築後は、お客様のアプリケーションのニーズに合わせて全長アダプターまたはユニバーサルアダプターをお使いいただけます。
全血由来サンプルを追跡することで、全エクソームシーケンスの精度を確保します。Twist Human Sample ID キットは、確立された Twist ライブラリ調製およびマルチプレックス PCR を利用してサンプルを識別・追跡し、サンプルの取り違えや不正確なデータ取得を防止します。
難易度の高いサンプルから優れた NGS データを引き出すには、シーケンス解析パイプライン全体で最適な試薬を用いる必要があります。
こちらのブログ記事では、優れた NGS データにつながる優れたライブラリ品質を確保するために Twist Library Preparation Enzymatic Fragmentation Kit 2.0 がどのような試薬で構成されているかをご紹介しています。

FFPE のような劣化した難しいサンプルを取り扱う場合、物理的断片化が有効である可能性があります。また、酵素による断片化と同様に、高品質な試薬が高品質な結果をもたらす鍵となります。Mechanical Fragmentation Library Preparation Kit の詳細は、こちらをご覧ください。
酵素による断片化の強化
Twist Library Preparation Enzymatic Fragmentation (EF) Kit 2.0 は、より効率的なシーケンス解析パイプラインを実現し、より正確な次世代シーケンシング (NGS) の結果を得るために構築されています。従来の Twist Library Preparation EF Kit 1.0 と比較して、この最新キットにはいくつかの利点があります。新たな断片化用・ライゲーション用モジュールに加えて、この新しいキットには Equinox Library Prep Amp Mix が含まれています。この酵素によるホットスタート技術を用いることで、エラー率が低く*、サンプル量が少ない場合でも効率良く多くの収量が得られるため、FFPE のような高い効率と収量が求められるサンプルに最適です。
このキットの合理的なワークフローによって、ライブラリ調製ステップが一つのチューブ内で完結し、低キメラ率および Twist の高均一性ターゲットエンリッチメントワークフローとあわせて、より高品質なライブラリ調製とシーケンス解析結果につながります。
* Twist Enzymatic Fragmentation 1.0 との比較
ライブラリ調製ワークフロー
Twist Library Preparation EF Kit 2.0 には、末端修復、dA-テーリング、アダプターライゲーションおよびライブラリ増幅に必要な試薬が含まれています。また、ゲノム DNAサンプル断片化用の酵素も含まれており、インサートサイズを調節することができます。コアライブラリの構築後は、お客様のアプリケーションのニーズに合わせて全長アダプターまたはユニバーサルアダプターをお使いいただけます。

全血由来サンプルを追跡することで、全エクソームシーケンスの精度を確保します。Twist Human Sample ID キットは、確立された Twist ライブラリ調製およびマルチプレックス PCR を利用してサンプルを識別・追跡し、サンプルの取り違えや不正確なデータ取得を防止します。
難易度の高いサンプルから優れた NGS データを引き出すには、シーケンス解析パイプライン全体で最適な試薬を用いる必要があります。
こちらのブログ記事では、優れた NGS データにつながる優れたライブラリ品質を確保するために Twist Library Preparation Enzymatic Fragmentation Kit 2.0 がどのような試薬で構成されているかをご紹介しています。

FFPE のような劣化した難しいサンプルを取り扱う場合、物理的断片化が有効である可能性があります。また、酵素による断片化と同様に、高品質な試薬が高品質な結果をもたらす鍵となります。Mechanical Fragmentation Library Preparation Kit の詳細は、こちらをご覧ください。
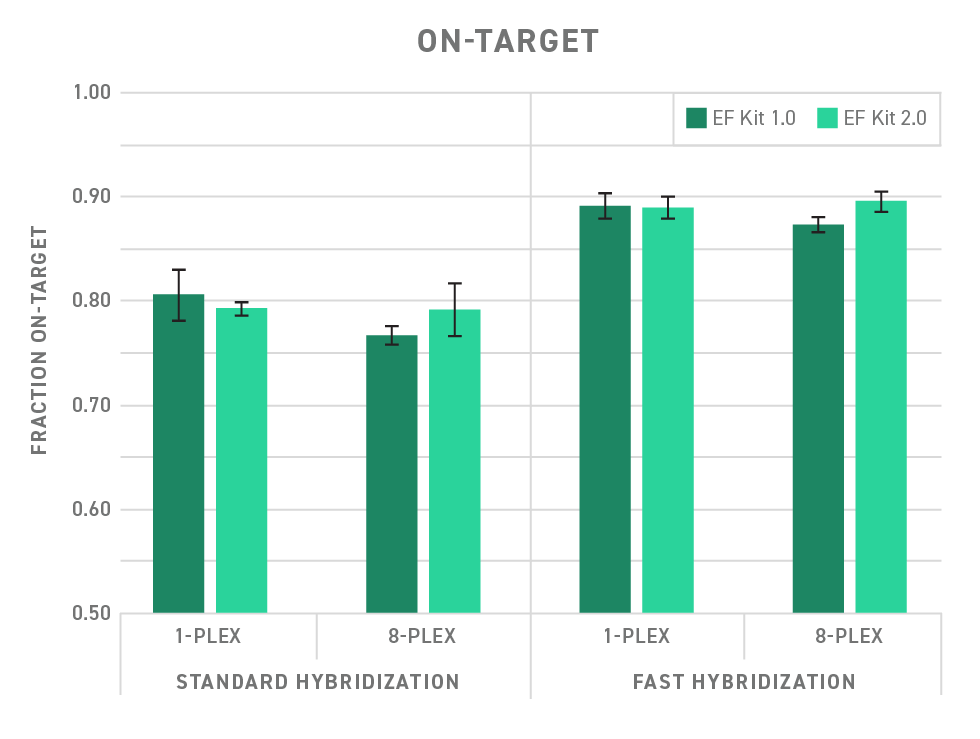
Twist Library Preparation EF Kit 2.0 のオンターゲット率は、 Twist Library Preparation EF Kit 1.0 で達成されたオンターゲット率と同等です。図では、異なるハイブリダイゼーション条件におけるオンターゲット率を示しています。
ターゲットエンリッチメントは、 Twist Core Exome Panel および Twist Universal Adapter System を用いて実施しました。シーケンシングは NextSeq® 550 または 2000 プラットフォームで行いました。データをターゲットサイズの 150 倍にダウンサンプリングし、Picard Metrics を用いて解析しました。エラーバーは反復して実施した際の標準偏差を表しています。
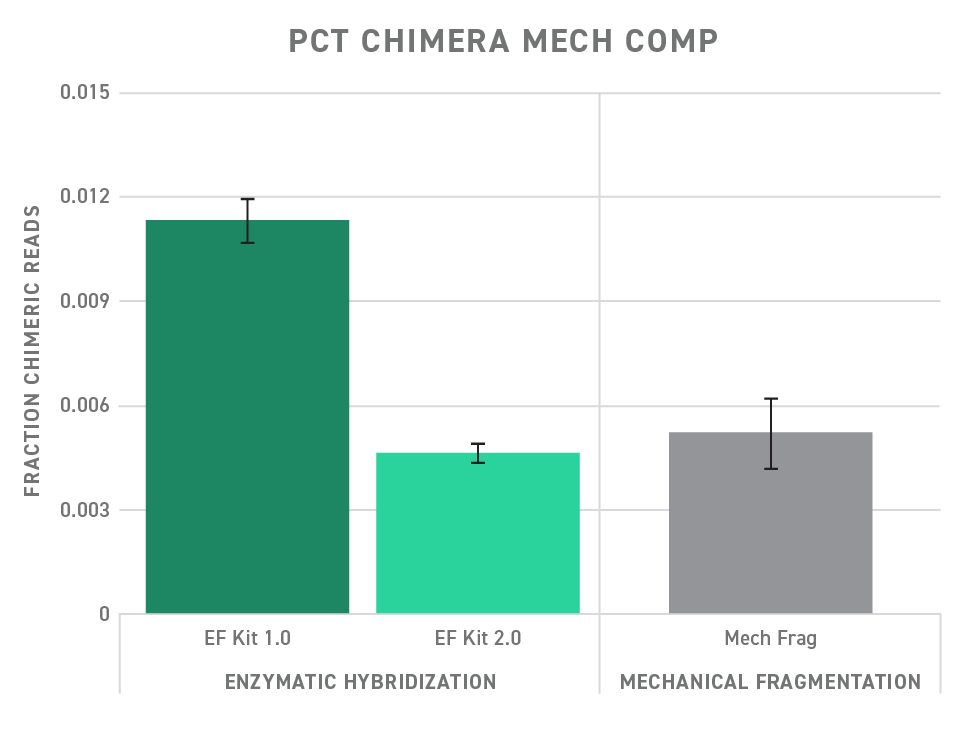
ライブラリ調製中、不適切な DNA ライゲーションや組換えを最小限に抑えることで、キメラ由来のシーケンス解析エラーができる限り起きないようにします。Twist Library Preparation EF Kit 2.0 では、 Twist Library Preparation EF Kit 1.0 と比較して、キメラリードの割合が大幅に減少していることが図から分かります。また、物理的断片化を用いたライブラリ調製と同等のキメラ率を示しています。
図において NGS ライブラリは酵素による断片化(Enzymatic Fragmentation)1.0 もしくは 2.0、または物理的断片化キットそれぞれの Twist ライブラリ調整キットを用いて作製しました。ターゲットエンリッチメントは、 Twist Core Exome Panel および Twist Universal Adapter System を用いて実施しました。シーケンシングは NextSeq® 2000 プラットフォームで行いました。データはターゲットサイズの 150 倍までダウンサンプリングし、Picard Metrics の PCT_CHIMERAS の値を得ました。エラーバーは反復して実施した際の標準偏差を表しています。
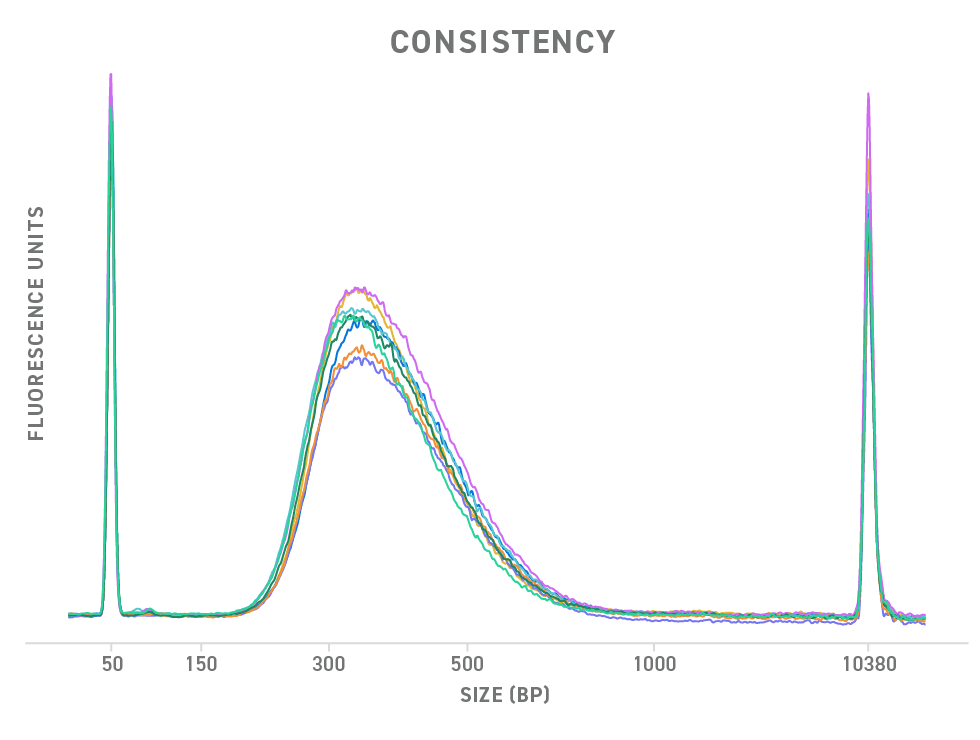
再現可能なパフォーマンスにより、結果の信頼性が保証されます。実験条件を一定にすると、 Twist Library Preparation EF Kit 2.0 を用いることで、堅牢で一貫した DNA ライブラリの断片サイズが常に得られます。こちらの図は、本キットで調製された8種類の NGS ライブラリの電気泳動図です。蛍光曲線が重なっていることから、一貫したライブラリ調製が可能であることが分かります。
この図において、 Twist Library Preparation EF Kit 2.0 と Twist Universal Adapter System を用いて NGS ライブラリを作製しました。50 ng の高品質ゲノム DNA を 37 ℃で 20 分間断片化しました。増幅には PCR を6サイクル行いました。サンプルは Agilent DNA 7500 アッセイで解析し、Expert 2100 ソフトウェアで結果を重ね合わせました。
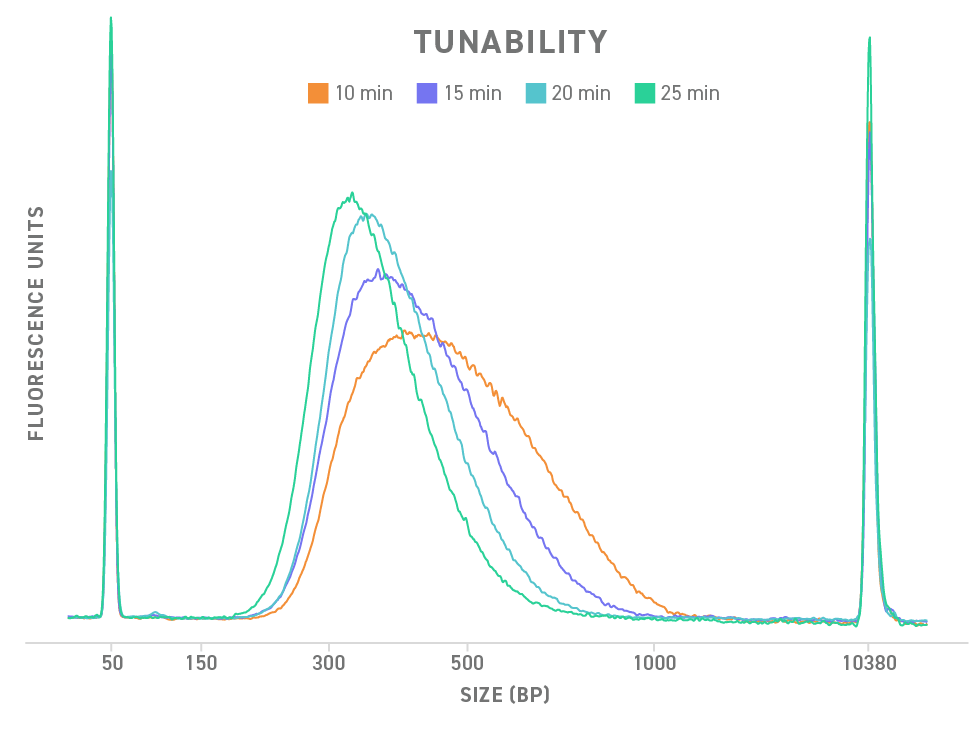
Twist Library Preparation EF Kit 2.0 では、断片化時間を調整することで、 DNA ライブラリの断片サイズを特定のニーズに合わせて調節できます。こちらの図は、断片化時間を変えて作製した4つの NGS ライブラリの電気泳動図です。
NGS ライブラリは、 Twist Library Preparation EF Kit 2.0 と Twist Universal Adapter System を用いて作製しました。50 ng の高品質ゲノム DNA を 37 ℃においてさまざまな時間で断片化しました。増幅には PCR を6サイクル行いました。サンプルは Agilent DNA 7500 アッセイで解析し、Expert 2100 ソフトウェアで結果を重ね合わせました。
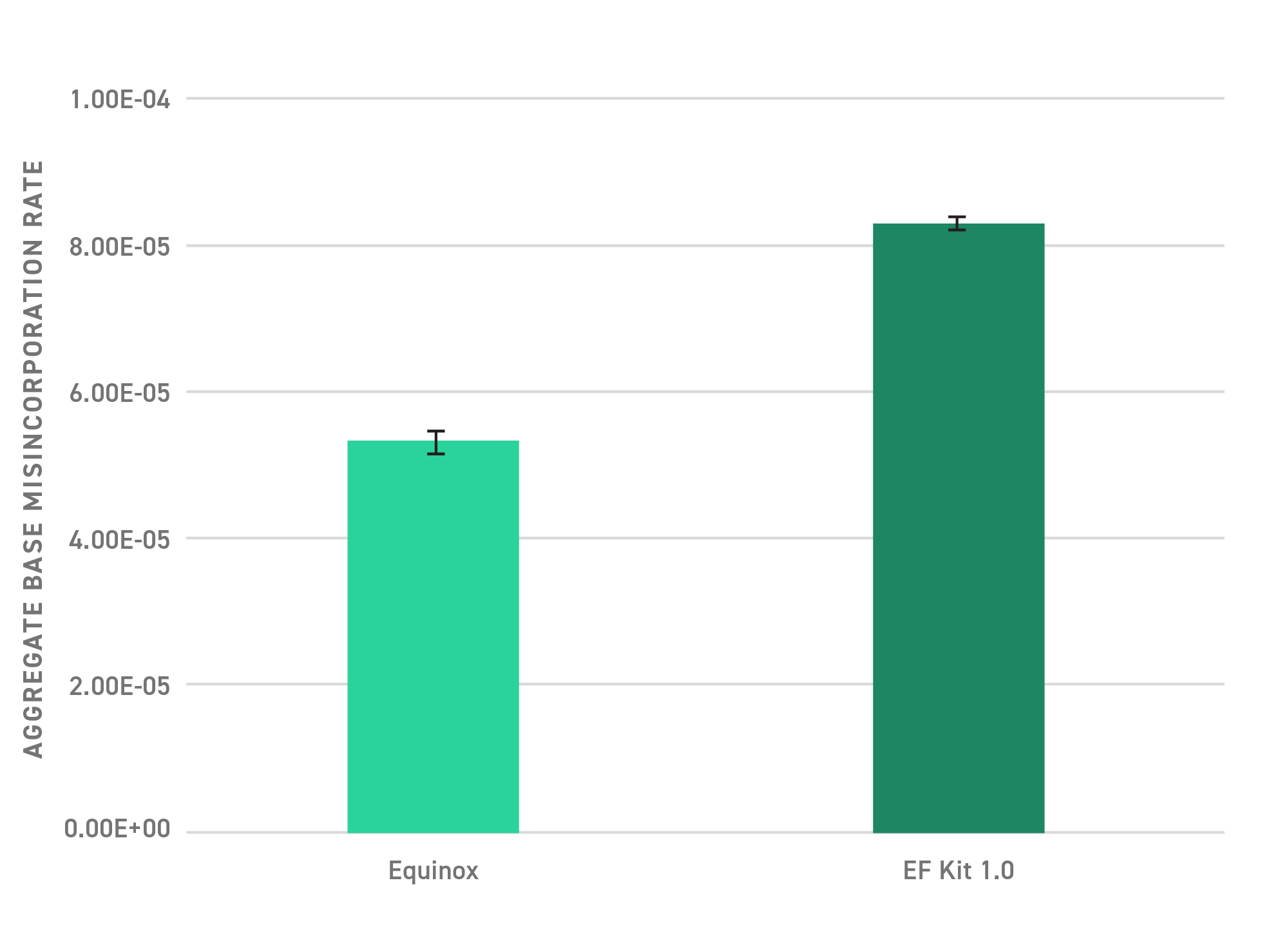
増幅時のエラーは避けられませんが、できれば減らしたいものです。Twist Library Preparation EF Kit 2.0 には、高忠実度のホットスタート酵素を特徴とした Equinox Master Mix が含まれています。これまで推奨されてきた一般的なポリメラーゼの使用と比較して、Equinox Master Mix は誤って取り込まれる塩基が少なく、低エラー率であることが実証されています。つまり、より正確な増幅が可能であり、結果として信頼性の高いシーケンス解析データを得ることができます。
NGS を用いた独自のアッセイにより、多数のミスマッチ塩基対にわたって見られる誤った塩基の取り込みを測定した結果を右図で示しています。表示されている値は、すべての誤取込イベントの平均エラー率を表しています。
Twist Library Preparation EF Kit 2.0 のオンターゲット率は、 Twist Library Preparation EF Kit 1.0 で達成されたオンターゲット率と同等です。図では、異なるハイブリダイゼーション条件におけるオンターゲット率を示しています。
ターゲットエンリッチメントは、 Twist Core Exome Panel および Twist Universal Adapter System を用いて実施しました。シーケンシングは NextSeq® 550 または 2000 プラットフォームで行いました。データをターゲットサイズの 150 倍にダウンサンプリングし、Picard Metrics を用いて解析しました。エラーバーは反復して実施した際の標準偏差を表しています。

ライブラリ調製中、不適切な DNA ライゲーションや組換えを最小限に抑えることで、キメラ由来のシーケンス解析エラーができる限り起きないようにします。Twist Library Preparation EF Kit 2.0 では、 Twist Library Preparation EF Kit 1.0 と比較して、キメラリードの割合が大幅に減少していることが図から分かります。また、物理的断片化を用いたライブラリ調製と同等のキメラ率を示しています。
図において NGS ライブラリは酵素による断片化(Enzymatic Fragmentation)1.0 もしくは 2.0、または物理的断片化キットそれぞれの Twist ライブラリ調整キットを用いて作製しました。ターゲットエンリッチメントは、 Twist Core Exome Panel および Twist Universal Adapter System を用いて実施しました。シーケンシングは NextSeq® 2000 プラットフォームで行いました。データはターゲットサイズの 150 倍までダウンサンプリングし、Picard Metrics の PCT_CHIMERAS の値を得ました。エラーバーは反復して実施した際の標準偏差を表しています。

再現可能なパフォーマンスにより、結果の信頼性が保証されます。実験条件を一定にすると、 Twist Library Preparation EF Kit 2.0 を用いることで、堅牢で一貫した DNA ライブラリの断片サイズが常に得られます。こちらの図は、本キットで調製された8種類の NGS ライブラリの電気泳動図です。蛍光曲線が重なっていることから、一貫したライブラリ調製が可能であることが分かります。
この図において、 Twist Library Preparation EF Kit 2.0 と Twist Universal Adapter System を用いて NGS ライブラリを作製しました。50 ng の高品質ゲノム DNA を 37 ℃で 20 分間断片化しました。増幅には PCR を6サイクル行いました。サンプルは Agilent DNA 7500 アッセイで解析し、Expert 2100 ソフトウェアで結果を重ね合わせました。

Twist Library Preparation EF Kit 2.0 では、断片化時間を調整することで、 DNA ライブラリの断片サイズを特定のニーズに合わせて調節できます。こちらの図は、断片化時間を変えて作製した4つの NGS ライブラリの電気泳動図です。
NGS ライブラリは、 Twist Library Preparation EF Kit 2.0 と Twist Universal Adapter System を用いて作製しました。50 ng の高品質ゲノム DNA を 37 ℃においてさまざまな時間で断片化しました。増幅には PCR を6サイクル行いました。サンプルは Agilent DNA 7500 アッセイで解析し、Expert 2100 ソフトウェアで結果を重ね合わせました。

増幅時のエラーは避けられませんが、できれば減らしたいものです。Twist Library Preparation EF Kit 2.0 には、高忠実度のホットスタート酵素を特徴とした Equinox Master Mix が含まれています。これまで推奨されてきた一般的なポリメラーゼの使用と比較して、Equinox Master Mix は誤って取り込まれる塩基が少なく、低エラー率であることが実証されています。つまり、より正確な増幅が可能であり、結果として信頼性の高いシーケンス解析データを得ることができます。
NGS を用いた独自のアッセイにより、多数のミスマッチ塩基対にわたって見られる誤った塩基の取り込みを測定した結果を右図で示しています。表示されている値は、すべての誤取込イベントの平均エラー率を表しています。

104206
Twist Library Preparation EF Kit 2.0, 16 Samples104207
Twist Library Preparation EF Kit 2.0, 96 Samplesゲノム DNA の酵素による断片化、ライブラリの構築と増幅に必要な試薬、酵素、精製ビーズが含まれます。
104206
Twist Library Preparation EF Kit 2.0, 16 Samples104207
Twist Library Preparation EF Kit 2.0, 96 Samplesゲノム DNA の酵素による断片化、ライブラリの構築と増幅に必要な試薬、酵素、精製ビーズが含まれます。
プロトコル
Library Preparation EF 2.0 with Enzymatic Fragmentation and Combinatorial Dual Indices
プロトコル
Library Preparation EF 2.0 with Enzymatic Fragmentation and Twist Universal Adapter System