Verbesserte enzymatische Fragmentierung
Das Library Preparation Enzymatic Fragmentation Kit 2.0 von Twist wurde entwickelt, um Ihnen dabei zu helfen, eine effizientere Sequenzierungspipeline und akkuratere Ergebnisse beim Next-Generation Sequencing (NGS) zu erreichen. Im Vergleich zum ursprünglichen Library Preparation EF Kit 1.0 von Twist bietet dieses aktualisierte Kit mehrere Vorteile. Zusätzlich zu neuen Fragmentierungs- und Ligationsmodulen umfasst das aktualisierte Kit jetzt auch den Equinox Library Prep Amp Mix. Diese Hot-Start-Enzymformulierung weist eine niedrigere Fehlerrate* und höhere Effizienz bei geringen Input-Volumen auf, wodurch es sich ideal für Proben wie FFPE eignet, bei denen eine hohe Effizienz und Ausbeute erforderlich sind.
Der optimierte Workflow des Kits kombiniert Schritte zur Libraryvorbereitung in einer Reaktion in nur einem Röhrchen. Neben der geringen Chimärenrate und dem sehr einheitlichen Target Enrichment-Workflow von Twist verbessert dies die Qualität der Libraryvorbereitung und der Sequenzierungsergebnisse.
*Im Vergleich zu Twist Enzymatic Fragmentation 1.0
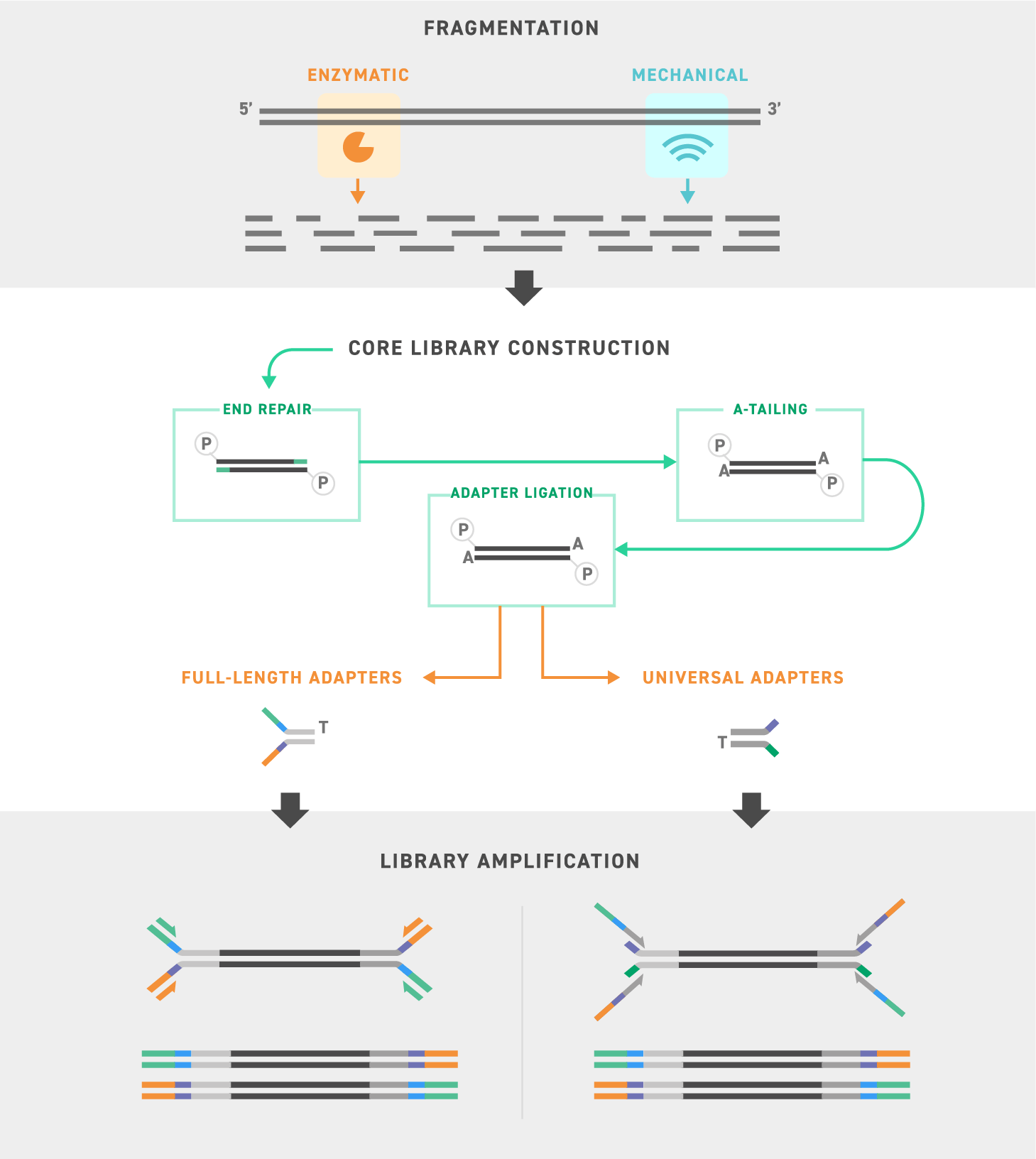
Libraryvorbereitung-Workflow
Das Library Preparation EF Kit 2.0 von Twist umfasst die Reagenzien, die für End-Repair, dA-tailing, Adapter-Ligation und Library-Amplifikation erforderlich sind. Dieses Kit enthält auch die Enzyme für die Fragmentierung von gDNA-Proben und ermöglicht eine Anpassung der Fragmentgröße. Nach der Erstellung der Core-Library kann entweder ein Universal Adapter oder ein Adapter in voller Länge verwendet werden, um den Anforderungen Ihrer Anwendung gerecht zu werden.
Sorgen Sie für Präzision bei der gesamten Exom-Sequenzierung durch die Verfolgung von Vollblutproben. Die Humanproben-ID von Twist verwendet bewährte Twist Library Prep-Kits und Muliplex-PCR zur Identifizierung und Verfolgung von Proben, um Verwechslungen von Proben und fehlerhafte Daten zu vermeiden.
Um hervorragende NGS-Daten selbst aus den schwersten Proben zu erhalten, müssen die besten Reagenzien in der gesamten Sequenzierungs-Pipeline verwendet werden.
Unseren Blog lesen, um zu erfahren, wie das Twist Library Preparation Enzymatic Fragmentation Kit 2.0 formuliert ist, um eine herausragende Library-Qualität zu gewährleisten, die zu herausragenden NGS-Daten führt.

Wenn mit schwierigen, degradierten Proben wie FFPE gearbeitet wird, könnte die mechanische Fragmentierung eine gute Option sein. Und wie bei der enzymatischen Fragmentierung sind qualitativ hochwertige Reagenzien wesentlich, um hochwertige Ergebnisse zu liefern. Mehr über unser Mechanical Fragmentation Library Preparation Kit.
Verbesserte enzymatische Fragmentierung
Das Library Preparation Enzymatic Fragmentation Kit 2.0 von Twist wurde entwickelt, um Ihnen dabei zu helfen, eine effizientere Sequenzierungspipeline und akkuratere Ergebnisse beim Next-Generation Sequencing (NGS) zu erreichen. Im Vergleich zum ursprünglichen Library Preparation EF Kit 1.0 von Twist bietet dieses aktualisierte Kit mehrere Vorteile. Zusätzlich zu neuen Fragmentierungs- und Ligationsmodulen umfasst das aktualisierte Kit jetzt auch den Equinox Library Prep Amp Mix. Diese Hot-Start-Enzymformulierung weist eine niedrigere Fehlerrate* und höhere Effizienz bei geringen Input-Volumen auf, wodurch es sich ideal für Proben wie FFPE eignet, bei denen eine hohe Effizienz und Ausbeute erforderlich sind.
Der optimierte Workflow des Kits kombiniert Schritte zur Libraryvorbereitung in einer Reaktion in nur einem Röhrchen. Neben der geringen Chimärenrate und dem sehr einheitlichen Target Enrichment-Workflow von Twist verbessert dies die Qualität der Libraryvorbereitung und der Sequenzierungsergebnisse.
*Im Vergleich zu Twist Enzymatic Fragmentation 1.0
Libraryvorbereitung-Workflow
Das Library Preparation EF Kit 2.0 von Twist umfasst die Reagenzien, die für End-Repair, dA-tailing, Adapter-Ligation und Library-Amplifikation erforderlich sind. Dieses Kit enthält auch die Enzyme für die Fragmentierung von gDNA-Proben und ermöglicht eine Anpassung der Fragmentgröße. Nach der Erstellung der Core-Library kann entweder ein Universal Adapter oder ein Adapter in voller Länge verwendet werden, um den Anforderungen Ihrer Anwendung gerecht zu werden.

Sorgen Sie für Präzision bei der gesamten Exom-Sequenzierung durch die Verfolgung von Vollblutproben. Die Humanproben-ID von Twist verwendet bewährte Twist Library Prep-Kits und Muliplex-PCR zur Identifizierung und Verfolgung von Proben, um Verwechslungen von Proben und fehlerhafte Daten zu vermeiden.
Um hervorragende NGS-Daten selbst aus den schwersten Proben zu erhalten, müssen die besten Reagenzien in der gesamten Sequenzierungs-Pipeline verwendet werden.
Unseren Blog lesen, um zu erfahren, wie das Twist Library Preparation Enzymatic Fragmentation Kit 2.0 formuliert ist, um eine herausragende Library-Qualität zu gewährleisten, die zu herausragenden NGS-Daten führt.

Wenn mit schwierigen, degradierten Proben wie FFPE gearbeitet wird, könnte die mechanische Fragmentierung eine gute Option sein. Und wie bei der enzymatischen Fragmentierung sind qualitativ hochwertige Reagenzien wesentlich, um hochwertige Ergebnisse zu liefern. Mehr über unser Mechanical Fragmentation Library Preparation Kit.
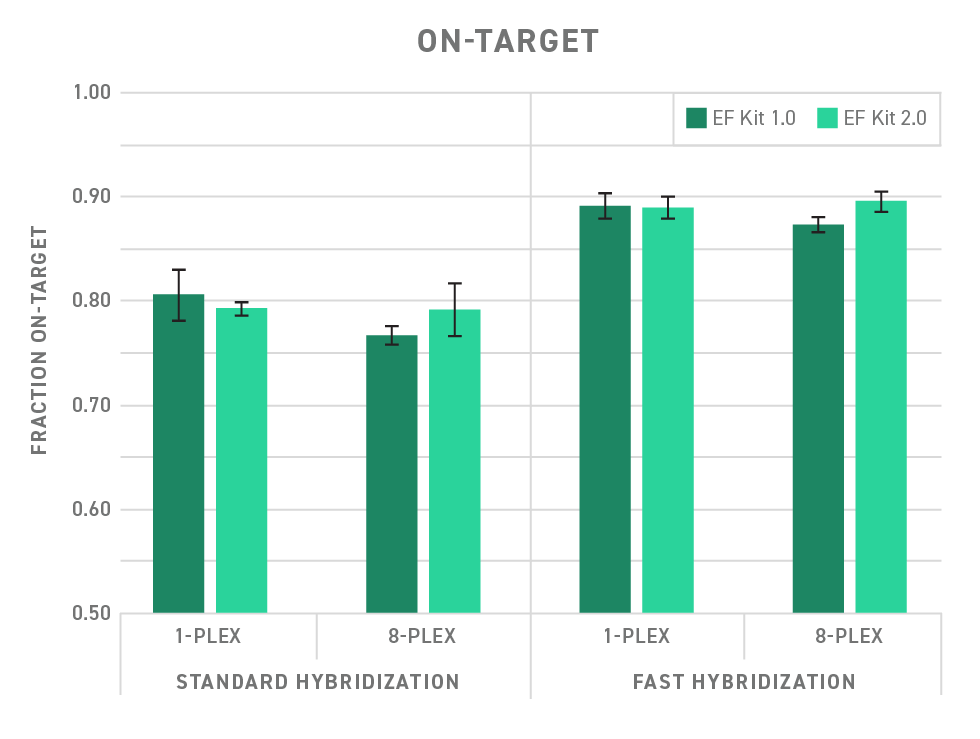
Die On-Target-Rate für das Twist Library Preparation EF Kit 2.0 erreicht eine äquivalente On-Target-Rate wie das Twist Library Preparation EF Kit 1.0. Hier wird ein Vergleich der Fraktion eines On-Target-Aufrufs unter verschiedenen Hybridisierungsbedingungen dargestellt.
Das Target Enrichment wurde mit dem Twist Core Exome Panel und dem Twist Universal Adapter System durchgeführt. Die Sequenzierung wurde mit den NextSeq® 550 oder 2000 Plattformen durchgeführt. Die Daten wurden auf das 150-fache der Zielgröße herunterreduziert und mit Picard Metrics analysiert. Die Fehlerbalken stellen die Standardabweichung zwischen den Replikaten dar.
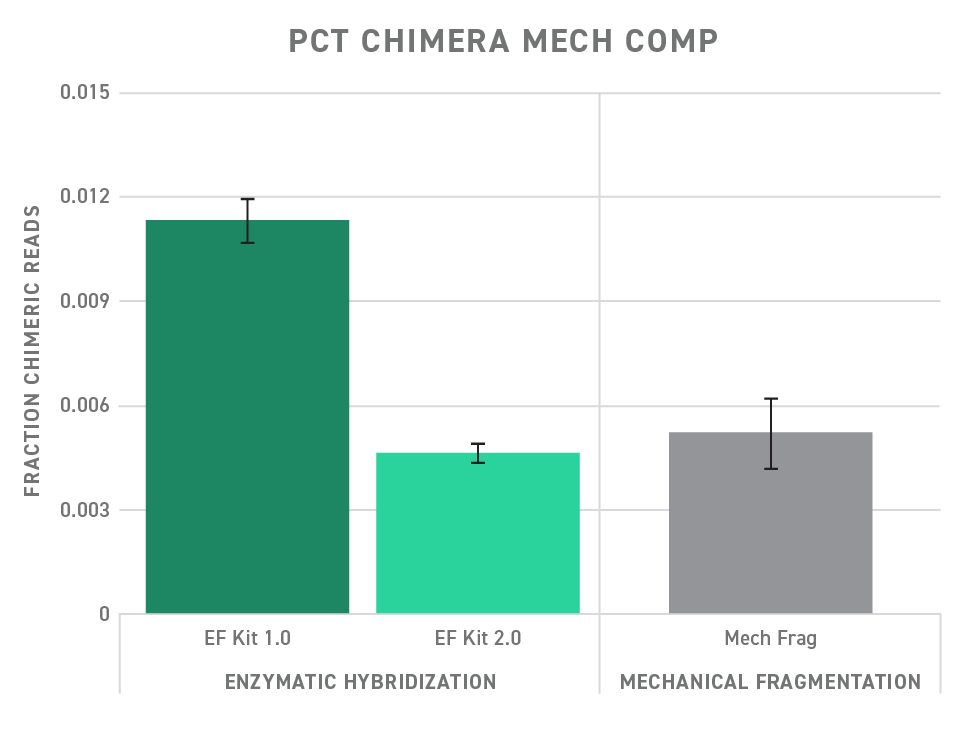
Während der Libraryvorbereitung werden durch die Minimierung ungeeigneter DNA-Ligationen oder -Rekombinationen die durch Chimären verursachten Sequenzierungsfehler minimiert. Das aktualisierte Twist Library Preparation EF Kit 2.0 liefert eine signifikant reduzierte Fraktion der Chimär-Auslesung, hier im Vergleich mit dem Twist Library Preparation EF Kit 1.0 dargestellt. Die Chimärenrate entspricht auch derjenigen bei der Libraryvorbereitung mit mechanischer Fragmentierung.
Die NGS-Libraries wurden mit dem Twist Library Preparation Kit with Enzymatic Fragmentation 1.0, Twist Enzymatic Kit with Enzymatic Fragmentation 2.0 oder Twist Library Preparation with Mechanical Fragmentation Kit erstellt. Das Target Enrichment wurde mit dem Twist Core Exome Panel und dem Twist Universal Adapter System durchgeführt. Die Sequenzierung wurde mit der NextSeq® 2000 Plattform durchgeführt. Die Daten wurden auf das 150-Fache der Zielgröße herunterreduziert und mit der Picard-Metrik PCT_CHIMERAS angegeben. Die Fehlerbalken stellen die Standardabweichung zwischen den Replikaten dar.
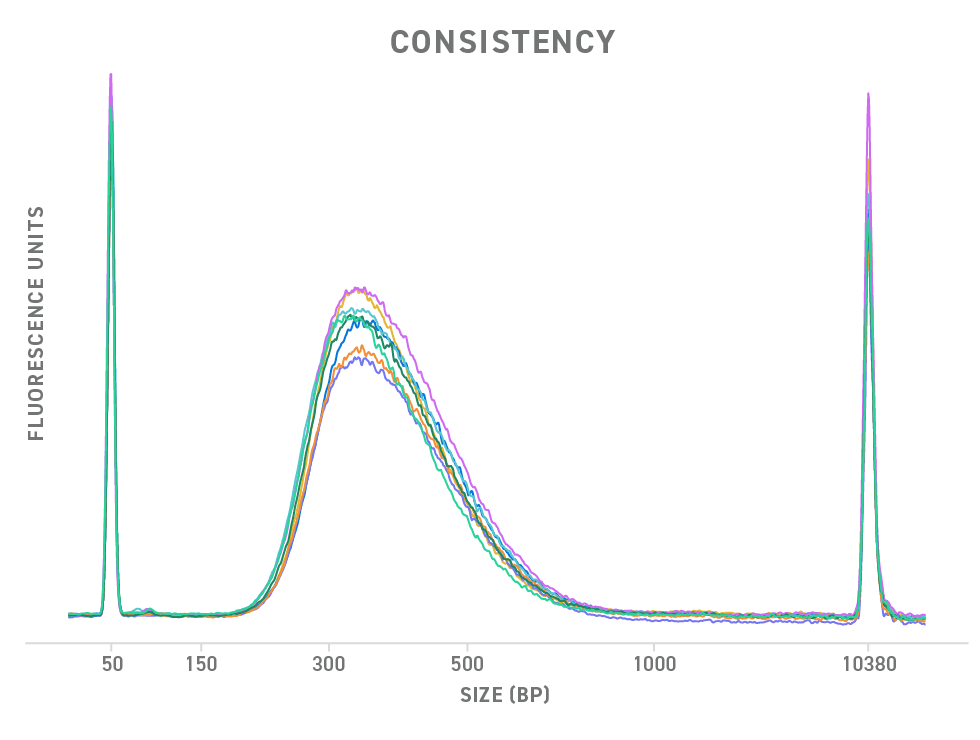
Wiederholbare Performance gewährleistet die Zuversicht bei den Ergebnissen. Wenn die Bedingungen bei den Experimenten gleich bleiben, liefert das Twist Library Preparation EF Kit 2.0 eine robuste, gleichbleibende DNA-Library-Abschnittsgröße. Hier werden 8 verschiedene Elektropherogramme von NGS-Libraries gezeigt, die mit dem Kit erzeugt wurden. Die Überlappung der Fluoreszenzkurven zeigen die Einheitlichkeit der Libraryvorbereitung, die bei jedem Lauf erreicht werden kann.
Die NGS-Libraries wurden mit dem Twist Library Preparation EF Kit 2.0 und dem Twist Universal Adapter System erzeugt. 50 ng hochqualitative gDNA wurden 20 Minuten lang bei 37 °C fragmentiert. 6 PCR-Zyklen wurden zur Amplifikation verwendet. Die Proben wurden mit einem Agilent DNA 7500 Assay analysiert und die Ergebnisse wurden mit der Expert 2100-Software überlagert.
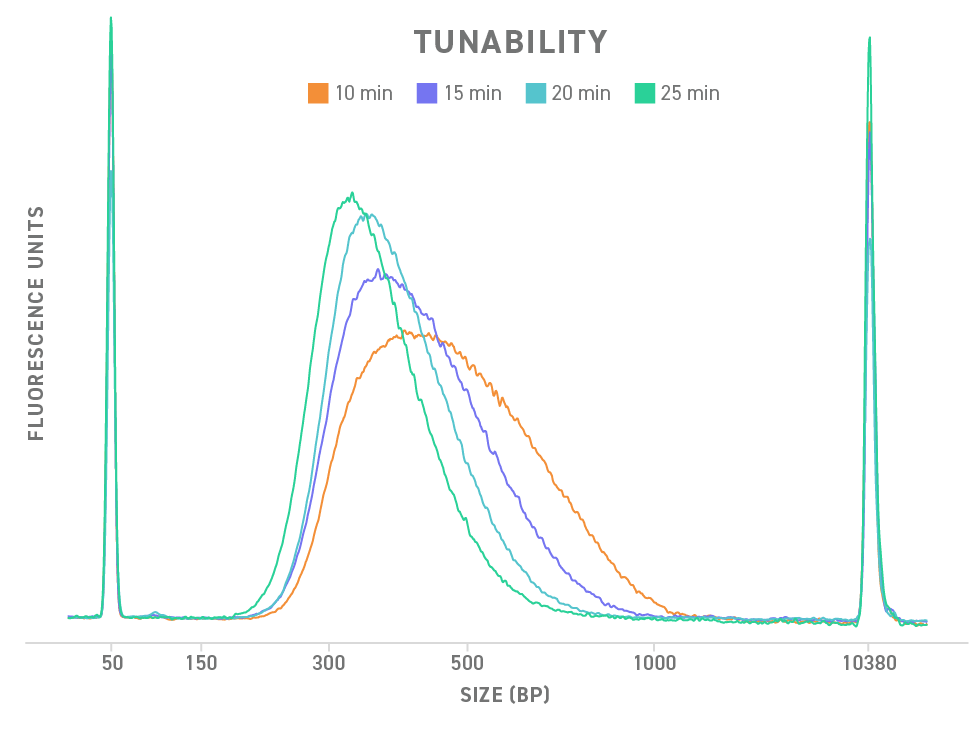
Mit dem Twist Library Preparation EF Kit 2.0 kann die DNA-Library-Fragmentgröße durch das einfache Adjustieren der Fragmentierungszeit auf Ihre besonderen Anforderungen angepasst werden. Hier werden vier Elektropherogramme von NGS-Libraries dargestellt, die mit verschiedenen Fragmentierungszeiten generiert wurden.
Die NGS-Libraries wurden mit dem Twist Library Preparation EF Kit 2.0 und dem Twist Universal Adapter System erzeugt. 50 ng hochqualitative gDNA wurden verschieden lang bei 37 °C fragmentiert. 6 PCR-Zyklen wurden zur Amplifikation verwendet. Die Proben wurden mit einem Agilent DNA 7500 Assay analysiert und die Ergebnisse wurden mit der Expert 2100-Software überlagert.
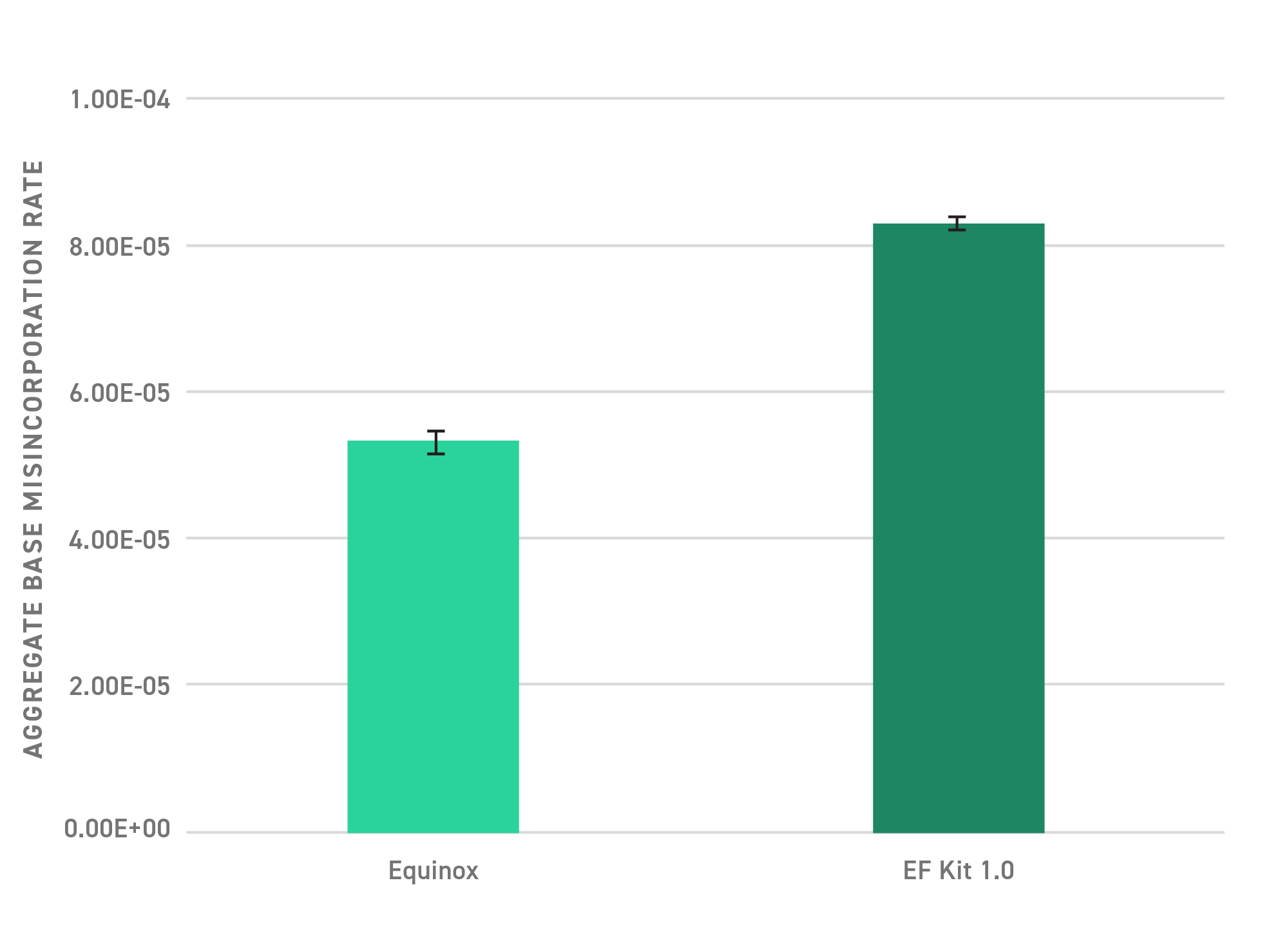
Fehler während der Amplifikation sind unvermeidbar, müssen aber nicht gängig sein. Das aktualisierte Twist Library Preparation EF Kit 2.0 umfasst den Equinox Master Mix mit einem sehr zuverlässigen Hot-Start-Enzym. Im Vergleich zu der zuvor empfohlenen und viel verwendeten Polymerase weist der Equinox Master Mix eine geringere Fehlerrate mit weniger falsch eingefügten Basen auf. Das bedeutet eine akkuratere Amplifikation und letztendlich Sequenzierungsdaten, denen Sie vertrauen können.
Mit einem proprietären NGS-basierten Assay wurde das Auftreten von Basen-Fehlpaarungen über zahlreiche Paare von nicht übereinstimmenden Basen gemessen. Die dargestellten Werte repräsentieren die durchschnittliche Fehlerrate aller Fehlpaarungen.
Die On-Target-Rate für das Twist Library Preparation EF Kit 2.0 erreicht eine äquivalente On-Target-Rate wie das Twist Library Preparation EF Kit 1.0. Hier wird ein Vergleich der Fraktion eines On-Target-Aufrufs unter verschiedenen Hybridisierungsbedingungen dargestellt.
Das Target Enrichment wurde mit dem Twist Core Exome Panel und dem Twist Universal Adapter System durchgeführt. Die Sequenzierung wurde mit den NextSeq® 550 oder 2000 Plattformen durchgeführt. Die Daten wurden auf das 150-fache der Zielgröße herunterreduziert und mit Picard Metrics analysiert. Die Fehlerbalken stellen die Standardabweichung zwischen den Replikaten dar.

Während der Libraryvorbereitung werden durch die Minimierung ungeeigneter DNA-Ligationen oder -Rekombinationen die durch Chimären verursachten Sequenzierungsfehler minimiert. Das aktualisierte Twist Library Preparation EF Kit 2.0 liefert eine signifikant reduzierte Fraktion der Chimär-Auslesung, hier im Vergleich mit dem Twist Library Preparation EF Kit 1.0 dargestellt. Die Chimärenrate entspricht auch derjenigen bei der Libraryvorbereitung mit mechanischer Fragmentierung.
Die NGS-Libraries wurden mit dem Twist Library Preparation Kit with Enzymatic Fragmentation 1.0, Twist Enzymatic Kit with Enzymatic Fragmentation 2.0 oder Twist Library Preparation with Mechanical Fragmentation Kit erstellt. Das Target Enrichment wurde mit dem Twist Core Exome Panel und dem Twist Universal Adapter System durchgeführt. Die Sequenzierung wurde mit der NextSeq® 2000 Plattform durchgeführt. Die Daten wurden auf das 150-Fache der Zielgröße herunterreduziert und mit der Picard-Metrik PCT_CHIMERAS angegeben. Die Fehlerbalken stellen die Standardabweichung zwischen den Replikaten dar.

Wiederholbare Performance gewährleistet die Zuversicht bei den Ergebnissen. Wenn die Bedingungen bei den Experimenten gleich bleiben, liefert das Twist Library Preparation EF Kit 2.0 eine robuste, gleichbleibende DNA-Library-Abschnittsgröße. Hier werden 8 verschiedene Elektropherogramme von NGS-Libraries gezeigt, die mit dem Kit erzeugt wurden. Die Überlappung der Fluoreszenzkurven zeigen die Einheitlichkeit der Libraryvorbereitung, die bei jedem Lauf erreicht werden kann.
Die NGS-Libraries wurden mit dem Twist Library Preparation EF Kit 2.0 und dem Twist Universal Adapter System erzeugt. 50 ng hochqualitative gDNA wurden 20 Minuten lang bei 37 °C fragmentiert. 6 PCR-Zyklen wurden zur Amplifikation verwendet. Die Proben wurden mit einem Agilent DNA 7500 Assay analysiert und die Ergebnisse wurden mit der Expert 2100-Software überlagert.

Mit dem Twist Library Preparation EF Kit 2.0 kann die DNA-Library-Fragmentgröße durch das einfache Adjustieren der Fragmentierungszeit auf Ihre besonderen Anforderungen angepasst werden. Hier werden vier Elektropherogramme von NGS-Libraries dargestellt, die mit verschiedenen Fragmentierungszeiten generiert wurden.
Die NGS-Libraries wurden mit dem Twist Library Preparation EF Kit 2.0 und dem Twist Universal Adapter System erzeugt. 50 ng hochqualitative gDNA wurden verschieden lang bei 37 °C fragmentiert. 6 PCR-Zyklen wurden zur Amplifikation verwendet. Die Proben wurden mit einem Agilent DNA 7500 Assay analysiert und die Ergebnisse wurden mit der Expert 2100-Software überlagert.

Fehler während der Amplifikation sind unvermeidbar, müssen aber nicht gängig sein. Das aktualisierte Twist Library Preparation EF Kit 2.0 umfasst den Equinox Master Mix mit einem sehr zuverlässigen Hot-Start-Enzym. Im Vergleich zu der zuvor empfohlenen und viel verwendeten Polymerase weist der Equinox Master Mix eine geringere Fehlerrate mit weniger falsch eingefügten Basen auf. Das bedeutet eine akkuratere Amplifikation und letztendlich Sequenzierungsdaten, denen Sie vertrauen können.
Mit einem proprietären NGS-basierten Assay wurde das Auftreten von Basen-Fehlpaarungen über zahlreiche Paare von nicht übereinstimmenden Basen gemessen. Die dargestellten Werte repräsentieren die durchschnittliche Fehlerrate aller Fehlpaarungen.

104206
Twist Library Preparation EF Kit 2.0, 16 Proben104207
Twist Library Preparation EF Kit 2.0, 96 ProbenReagenzien, Enzyme und Aufreinigungs-Beads erforderlich für die enzymatische Fragmentierung von gDNA, Library-Konstruktion und Amplifikation
104206
Twist Library Preparation EF Kit 2.0, 16 Proben104207
Twist Library Preparation EF Kit 2.0, 96 ProbenReagenzien, Enzyme und Aufreinigungs-Beads erforderlich für die enzymatische Fragmentierung von gDNA, Library-Konstruktion und Amplifikation
Protokoll
Library Preparation EF 2.0 mit enzymatischer Fragmentierung und kombinatorischen dualen Indizes
Protokoll
Library Preparation EF 2.0 mit enzymatischer Fragmentierung und Twist Universal Adapter System
Videos
Fehlerbehebung mit Twist: EF 2.0 Fragmentierung-Mastermix-Herstellung und Zugabe zur Probe
Protokoll
Library Preparation EF 2.0 mit enzymatischer Fragmentierung und kombinatorischen dualen Indizes
Protokoll
Library Preparation EF 2.0 mit enzymatischer Fragmentierung und Twist Universal Adapter System
Videos
Fehlerbehebung mit Twist: EF 2.0 Fragmentierung-Mastermix-Herstellung und Zugabe zur Probe
