未開拓の酵素配列空間を開放し、最適な探索を可能に
当社の新規酵素配列は、多様な未培養微生物単細胞ゲノムから同定され、構造予測、構造に基づくクラスタリング及びフィルタリングを基にした機械学習支援モデルを用いて、望ましい特性を持つよう選択されています。このような配列は他のどこにも見つからない可能性が高いです。
トランスアミナーゼ(TA)は、アミンドナーからケトンまたはアルデヒドへのアミン基の転移を触媒する酵素です。プロキラルケトンの立体選択的アミノ化を促進するこの能力は、医薬品製造において有用であり、医薬品有効成分(API)の構成成分としてキラルアミンの需要が高い製薬業界では貴重な存在です。
当社は、医薬品製造プロセスに有用な、多種多様な基質にわたって様々な極限条件下で活性を維持する TA 酵素のサブセットを同定し、絞り込みました。
当社のスクリーニングキットは、機能面で重要な配列の選択や、これらを下流の酵素工学のリソースと組み合わせることを可能にし、より効率的なタンパク質工学の経路を提供する可能性を秘めています。
スクリーニング用酵素の選定方法
bitBiome bit-GEM データベースは、検索可能で有用性の高い微生物ゲノム配列の主要なリソースです。bit-GEM データベースには、土壌、温泉、海水、鉱山、極限環境等の自然環境から採取したサンプルなど、自然界で最も強力な微生物資源を利用し、bitBiome シングルセルシーケンシングプラットフォームによって取得された膨大な遺伝子配列が含まれています。このシーケンシング法は、より深いゲノムカバレッジを保証し、未培養微生物の発見を可能にします。これらの配列は、公知の領域にある配列との類似性が低いため、その配列空間内で自由に利用することができます。



未開拓の酵素配列空間を開放し、最適な探索を可能に
当社の新規酵素配列は、多様な未培養微生物単細胞ゲノムから同定され、構造予測、構造に基づくクラスタリング及びフィルタリングを基にした機械学習支援モデルを用いて、望ましい特性を持つよう選択されています。このような配列は他のどこにも見つからない可能性が高いです。
トランスアミナーゼ(TA)は、アミンドナーからケトンまたはアルデヒドへのアミン基の転移を触媒する酵素です。プロキラルケトンの立体選択的アミノ化を促進するこの能力は、医薬品製造において有用であり、医薬品有効成分(API)の構成成分としてキラルアミンの需要が高い製薬業界では貴重な存在です。
当社は、医薬品製造プロセスに有用な、多種多様な基質にわたって様々な極限条件下で活性を維持する TA 酵素のサブセットを同定し、絞り込みました。
当社のスクリーニングキットは、機能面で重要な配列の選択や、これらを下流の酵素工学のリソースと組み合わせることを可能にし、より効率的なタンパク質工学の経路を提供する可能性を秘めています。
スクリーニング用酵素の選定方法
bitBiome bit-GEM データベースは、検索可能で有用性の高い微生物ゲノム配列の主要なリソースです。bit-GEM データベースには、土壌、温泉、海水、鉱山、極限環境等の自然環境から採取したサンプルなど、自然界で最も強力な微生物資源を利用し、bitBiome シングルセルシーケンシングプラットフォームによって取得された膨大な遺伝子配列が含まれています。このシーケンシング法は、より深いゲノムカバレッジを保証し、未培養微生物の発見を可能にします。これらの配列は、公知の領域にある配列との類似性が低いため、その配列空間内で自由に利用することができます。



スクリーニング用酵素の選定方法
bit-GEM データベースには、土壌、温泉、海水、鉱山、極限環境等の自然環境から採取したサンプルなど、自然界で最も強力な微生物資源を利用し、bitBiome シングルセルシーケンシングプラットフォームによって取得された膨大な遺伝子配列が含まれています。このシーケンシング法は、より深いゲノムカバレッジを保証し、未培養微生物の発見を可能にします。これらの配列は、これらの配列は、公知の領域にある配列との類似性が低いため、その配列空間内でより自由に操作することができます。このキットで提供される 48 種類の酵素の選定は、Twist の DNA 合成能力だけでなく、機能的配列空間を洗練させるための共同バイオインフォマティクスの取り組みにより実現しました。
キットに含まれる酵素の選定方法
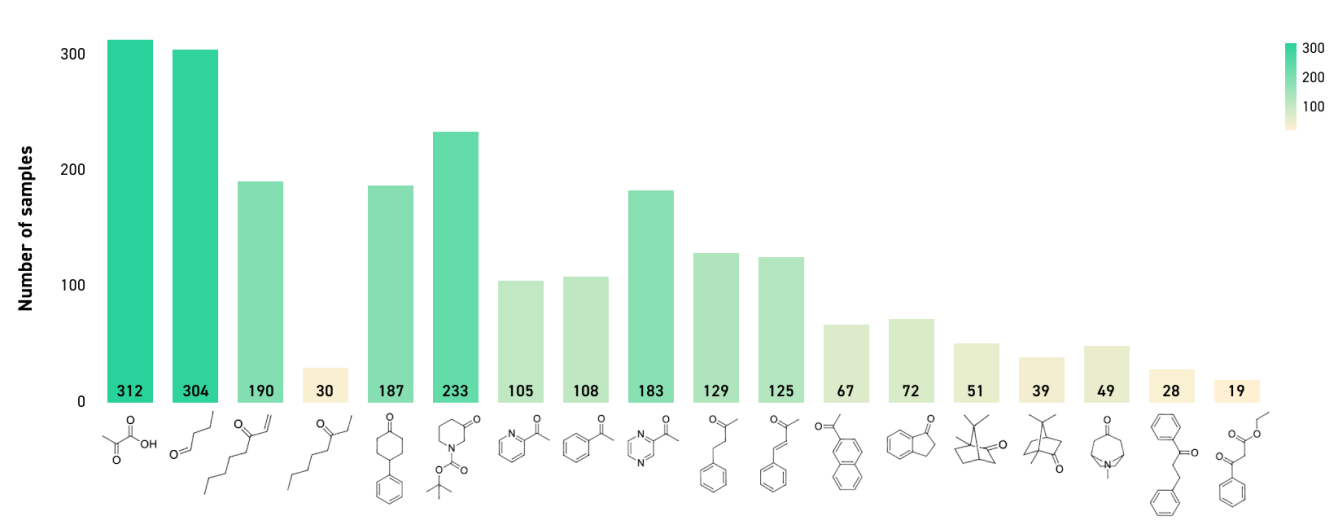
19 種類の基質を用いて、活性を持つ計 369 種類のトランスアミナーゼを同定しました。そのうち 273 種類の酵素は 10% DMSO 中で活性を示し、30 種類の酵素は 60 ℃ で活性を示しました。最後に、酵素の pH 感受性と選択性について試験を行いました。
トランスアミナーゼスクリーニングの基準
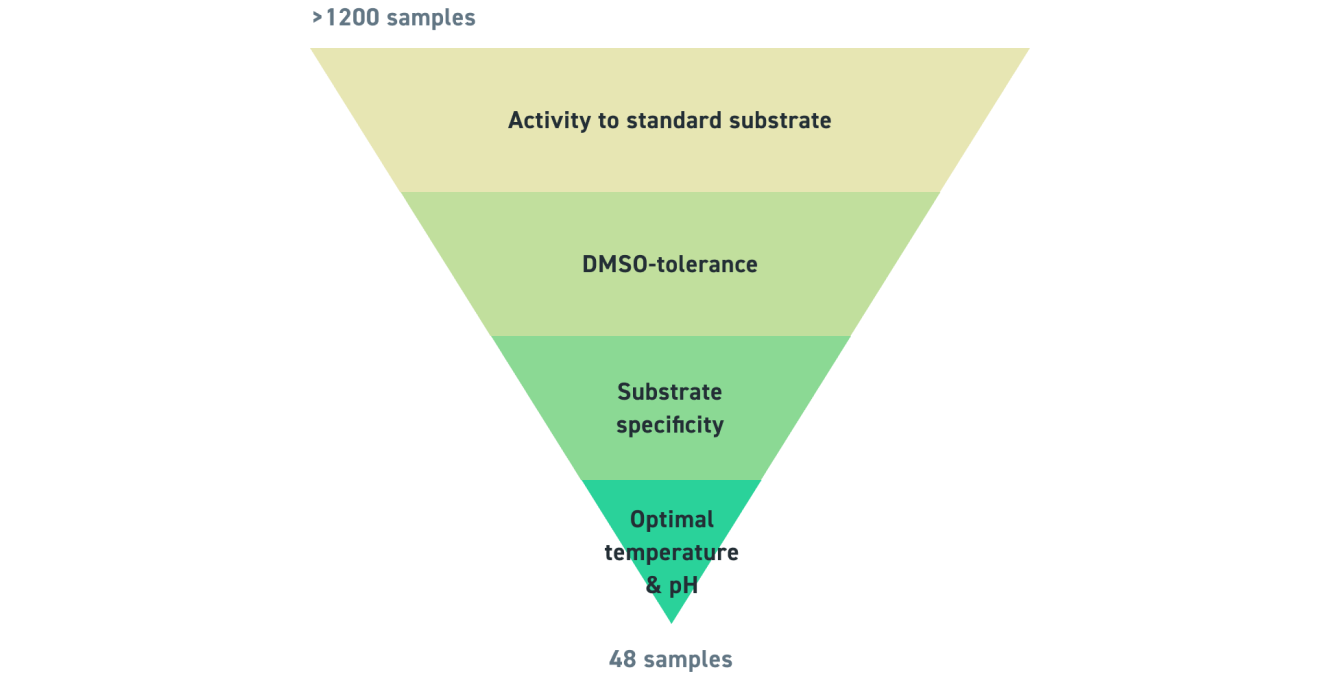
以下は、前述のさまざまな条件(DMSO の種々の割合、有機溶媒、温度、pH レベル)で、さまざまな基質に対して試験を行った酵素の分析結果です。
スクリーニング用酵素の選定方法
bit-GEM データベースには、土壌、温泉、海水、鉱山、極限環境等の自然環境から採取したサンプルなど、自然界で最も強力な微生物資源を利用し、bitBiome シングルセルシーケンシングプラットフォームによって取得された膨大な遺伝子配列が含まれています。このシーケンシング法は、より深いゲノムカバレッジを保証し、未培養微生物の発見を可能にします。これらの配列は、これらの配列は、公知の領域にある配列との類似性が低いため、その配列空間内でより自由に操作することができます。このキットで提供される 48 種類の酵素の選定は、Twist の DNA 合成能力だけでなく、機能的配列空間を洗練させるための共同バイオインフォマティクスの取り組みにより実現しました。
キットに含まれる酵素の選定方法
19 種類の基質を用いて、活性を持つ計 369 種類のトランスアミナーゼを同定しました。そのうち 273 種類の酵素は 10% DMSO 中で活性を示し、30 種類の酵素は 60 ℃ で活性を示しました。最後に、酵素の pH 感受性と選択性について試験を行いました。
トランスアミナーゼスクリーニングの基準

以下は、前述のさまざまな条件(DMSO の種々の割合、有機溶媒、温度、pH レベル)で、さまざまな基質に対して試験を行った酵素の分析結果です。