Das Twist Human Methylome Panel zielt auf 3,98 Millionen CpG-Stellen durch 123 MB genomischen Inhalts ab, um biologisch relevante Methylierungsmarker anzuvisieren. Umfangreicher Inhalt macht dieses Panel zur idealen Wahl für Wissenschaftler bei der Erforschung der Methylierungsfraktion in einer Vielzahl von Anwendungen von Krebsmetastasen, menschlicher Entwicklung und funktioneller Genomik.
Das Panel ist für die Verwendung mit dem Twist NGS Methylation Detection System für einen vollständigen durchgängigen Workflow mit branchenführender Leistung optimiert und validiert. Eine hohe Erfassungseffizienz erhöht die Erkennungsempfindlichkeit im gesamten Epigenom und senkt gleichzeitig die Sequenzierungskosten. Das Panel ist ideal für das Screening von Kohortenproben und die Identifizierung unterschiedlich methylierter Regionen.
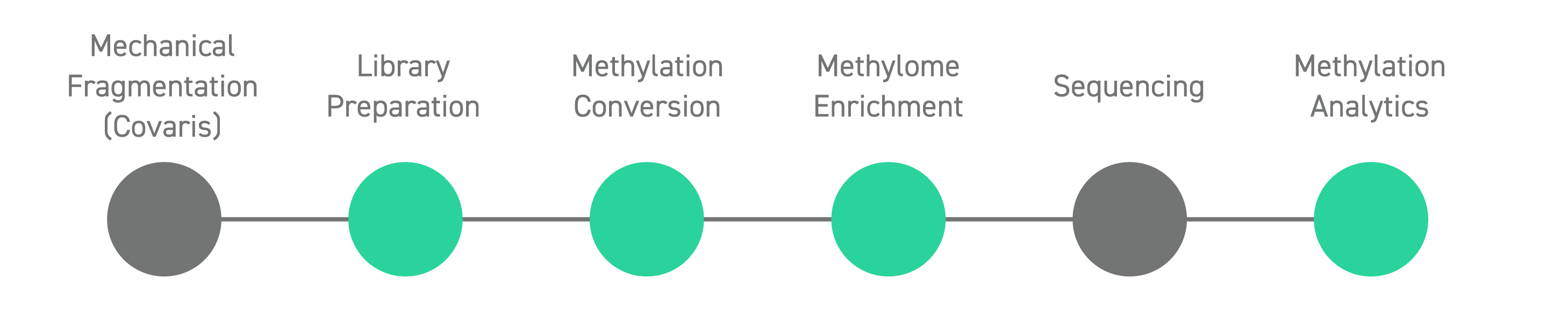
Eine Komplettlösung, die eine hochkomplexe und einheitliche Sequenzierungsauslesung für die Methylierungsanalyse erzeugt. Das durchgängige Protokoll erreicht dies durch die Kombination eines innovativen, enzymatischen Konvertierungsprozesses, eines optimierten Target Enrichment-Workflows und eines hochentwickelten Panel-Design-Prozesses.
Das Twist Human Methylome Panel zielt auf 3,98 Millionen CpG-Stellen durch 123 MB genomischen Inhalts ab, um biologisch relevante Methylierungsmarker anzuvisieren. Umfangreicher Inhalt macht dieses Panel zur idealen Wahl für Wissenschaftler bei der Erforschung der Methylierungsfraktion in einer Vielzahl von Anwendungen von Krebsmetastasen, menschlicher Entwicklung und funktioneller Genomik.
Das Panel ist für die Verwendung mit dem Twist NGS Methylation Detection System für einen vollständigen durchgängigen Workflow mit branchenführender Leistung optimiert und validiert. Eine hohe Erfassungseffizienz erhöht die Erkennungsempfindlichkeit im gesamten Epigenom und senkt gleichzeitig die Sequenzierungskosten. Das Panel ist ideal für das Screening von Kohortenproben und die Identifizierung unterschiedlich methylierter Regionen.
Eine Komplettlösung, die eine hochkomplexe und einheitliche Sequenzierungsauslesung für die Methylierungsanalyse erzeugt. Das durchgängige Protokoll erreicht dies durch die Kombination eines innovativen, enzymatischen Konvertierungsprozesses, eines optimierten Target Enrichment-Workflows und eines hochentwickelten Panel-Design-Prozesses.

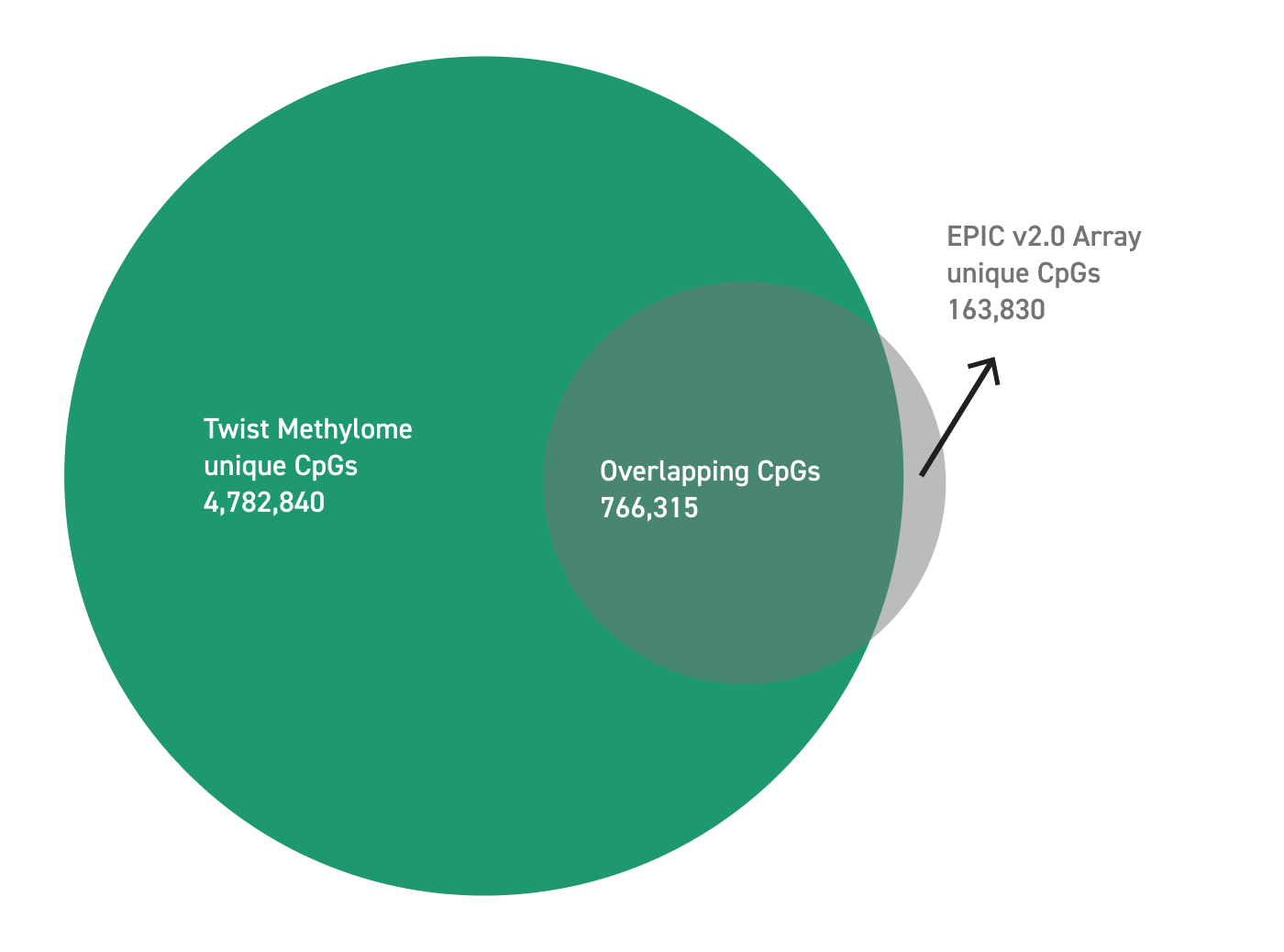
Twist Human Methylome enthält 3,98 Millionen CpG-Stellen in 123 MB genomischen Inhalts, um biologisch relevante Methylierungsmarker anzuvisieren. Das Panel ist hochgradig spezifisch bei der Erfassung und Erkennung der aktuellen, am stärksten annotierten und relevantesten CpG-Methylierungsregionen des Genoms. 84 % (17.915.988) der CpG-Inseln im Genom werden mithilfe dieses Panels durch Anreicherung identifiziert. Weitere 105.288.339 Basen nachfolgender Shores, Shelves und Open-Sea-CpGs und Basenpaare werden abgedeckt. Abdeckungsüberlappung, bestimmt durch interne experimentelle Abdeckung von Daten, einschließlich Shadow-Abdeckung im Vergleich zu Array-Abdeckungsdatendateien.
Ein Microarray ist mit statischem Inhalt konzipiert, der die Identifizierung neuer epigenetischer Targets von Interesse behindern kann. Aktuelle Arrays auf dem Markt weisen auch eine begrenzte Abdeckung des Methyloms über das Epigenom hinweg auf, wodurch ein großer Teil der methylierten CpG-Stellen ungemessen bleibt. Der Twist Human Methylome Target Enrichment-Ansatz behebt diese Einschränkungen mit Hybrid Capture-Panels, die erweiterte Inhalte und Einzelbasenauflösung über den gesamten von NGS-Plattformen bereitgestellten Sequenzabschnitt ermöglichen.
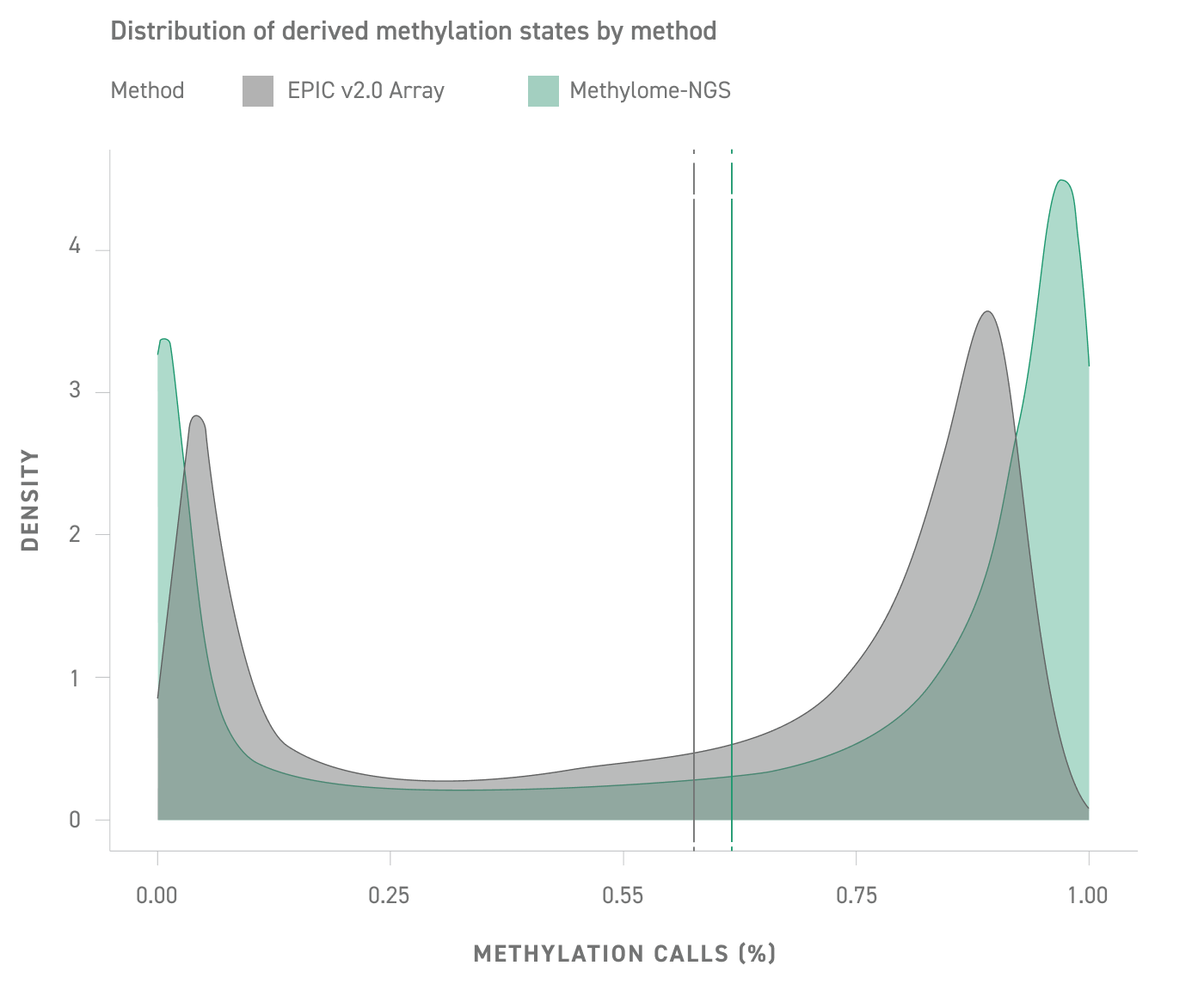
Microarrays haben inhärente Einschränkungen an extremen Enden der Methylierungserkennung, die auf starkes Hintergrundrauschen am unteren Ende und Signalsättigung am oberen Ende zurückzuführen sind. Aufgrund des höheren Dynamikbereichs von NGS kann das Twist Human Methylome Panel eine genauere Bestimmung von differenziell methylierten Regionen (DMR) ermöglichen, insbesondere sowohl am unteren als auch am oberen Ende der Methylierungsfraktion.
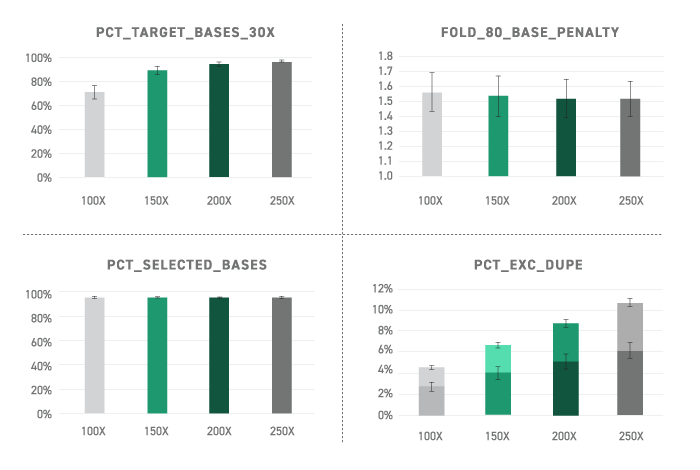
Das Twist Human Methylome Panel wurde entwickelt und im Nasslabor optimiert, um eine genaue, empfindliche und leistungsstarke Hybrid Capture-Effizienz zu bieten. Das Panel erreicht eine Abdeckungstiefe von 90 % der Basen bei 30-facher Abdeckung mit hohen Sondenspezifitäten von 95 % On-Target-Raten. Das Panel erreicht auch eine Fold 80 von 1,54. Dies stellt eine hohe Homogenität über die Zielregion hinweg bereit, indem das Delta zwischen den Spitzen und Tälern der Base-Call-Anhäufungen verringert wird. Die Library-Komplexität wurde umgekehrt durch eine Duplizierungsrate von < 4 % bei einer hohen Sequenzierungstiefe von 150-facher Rohabdeckung dargestellt. Die allgemeinen Erfassungseffizienz-Metriken des Panels bieten ein hohes Maß an Vertrauen in die Erkennung über die Methylierungsfraktion hinweg bei minimierten Sequenzierungskosten.
Twist Human Methylome enthält 3,98 Millionen CpG-Stellen in 123 MB genomischen Inhalts, um biologisch relevante Methylierungsmarker anzuvisieren. Das Panel ist hochgradig spezifisch bei der Erfassung und Erkennung der aktuellen, am stärksten annotierten und relevantesten CpG-Methylierungsregionen des Genoms. 84 % (17.915.988) der CpG-Inseln im Genom werden mithilfe dieses Panels durch Anreicherung identifiziert. Weitere 105.288.339 Basen nachfolgender Shores, Shelves und Open-Sea-CpGs und Basenpaare werden abgedeckt. Abdeckungsüberlappung, bestimmt durch interne experimentelle Abdeckung von Daten, einschließlich Shadow-Abdeckung im Vergleich zu Array-Abdeckungsdatendateien.

Ein Microarray ist mit statischem Inhalt konzipiert, der die Identifizierung neuer epigenetischer Targets von Interesse behindern kann. Aktuelle Arrays auf dem Markt weisen auch eine begrenzte Abdeckung des Methyloms über das Epigenom hinweg auf, wodurch ein großer Teil der methylierten CpG-Stellen ungemessen bleibt. Der Twist Human Methylome Target Enrichment-Ansatz behebt diese Einschränkungen mit Hybrid Capture-Panels, die erweiterte Inhalte und Einzelbasenauflösung über den gesamten von NGS-Plattformen bereitgestellten Sequenzabschnitt ermöglichen.
Microarrays haben inhärente Einschränkungen an extremen Enden der Methylierungserkennung, die auf starkes Hintergrundrauschen am unteren Ende und Signalsättigung am oberen Ende zurückzuführen sind. Aufgrund des höheren Dynamikbereichs von NGS kann das Twist Human Methylome Panel eine genauere Bestimmung von differenziell methylierten Regionen (DMR) ermöglichen, insbesondere sowohl am unteren als auch am oberen Ende der Methylierungsfraktion.

Das Twist Human Methylome Panel wurde entwickelt und im Nasslabor optimiert, um eine genaue, empfindliche und leistungsstarke Hybrid Capture-Effizienz zu bieten. Das Panel erreicht eine Abdeckungstiefe von 90 % der Basen bei 30-facher Abdeckung mit hohen Sondenspezifitäten von 95 % On-Target-Raten. Das Panel erreicht auch eine Fold 80 von 1,54. Dies stellt eine hohe Homogenität über die Zielregion hinweg bereit, indem das Delta zwischen den Spitzen und Tälern der Base-Call-Anhäufungen verringert wird. Die Library-Komplexität wurde umgekehrt durch eine Duplizierungsrate von < 4 % bei einer hohen Sequenzierungstiefe von 150-facher Rohabdeckung dargestellt. Die allgemeinen Erfassungseffizienz-Metriken des Panels bieten ein hohes Maß an Vertrauen in die Erkennung über die Methylierungsfraktion hinweg bei minimierten Sequenzierungskosten.

105517
Twist Human Methylome Panel, 2 Reaktionen105520
Twist Human Methylome Panel, 12 Reaktionen105521
Twist Human Methylome Panel, 96 Reaktionen105517
Twist Human Methylome Panel, 2 Reaktionen105520
Twist Human Methylome Panel, 12 Reaktionen105521
Twist Human Methylome Panel, 96 Reaktionen
Technischer Hinweis
Analyse der Twist EM-seq Targeted Methylation, Sequencing-Daten mithilfe des Twist Human Methylome Panel
Technischer Hinweis
Analyse der Twist EM-seq Targeted Methylation, Sequencing-Daten mithilfe des Twist Human Methylome Panel