Einblicke in die Sequenzierung viraler Erreger, die mit akuten Atemwegsinfektionen in Verbindung stehen
In den Atemwegen können je nach Wirtsumgebung viele verschiedene Arten von kommensalen und pathogenen Organismen vorkommen. Der Nachweis einer Spezies allein bietet möglicherweise nicht den vollständigsten Überblick über die biologische Analyse einer Probe. Das Twist Respiratory Virus Research Panel ermöglicht die Anreicherung und Sequenzierung viraler Erreger von Atemwegserkrankungen, so dass Forscher die Spezies nachweisen und Sequenzinformationen über die viralen Genome erhalten können.
Außerdem können sich die Symptome einer akuten Atemwegsinfektion mit denen anderer Atemwegsviren überschneiden. Das Twist Respiratory Virus Research Panel kann Forschern helfen, die Infektionsursache zu ermitteln und Koinfektionen festzustellen.
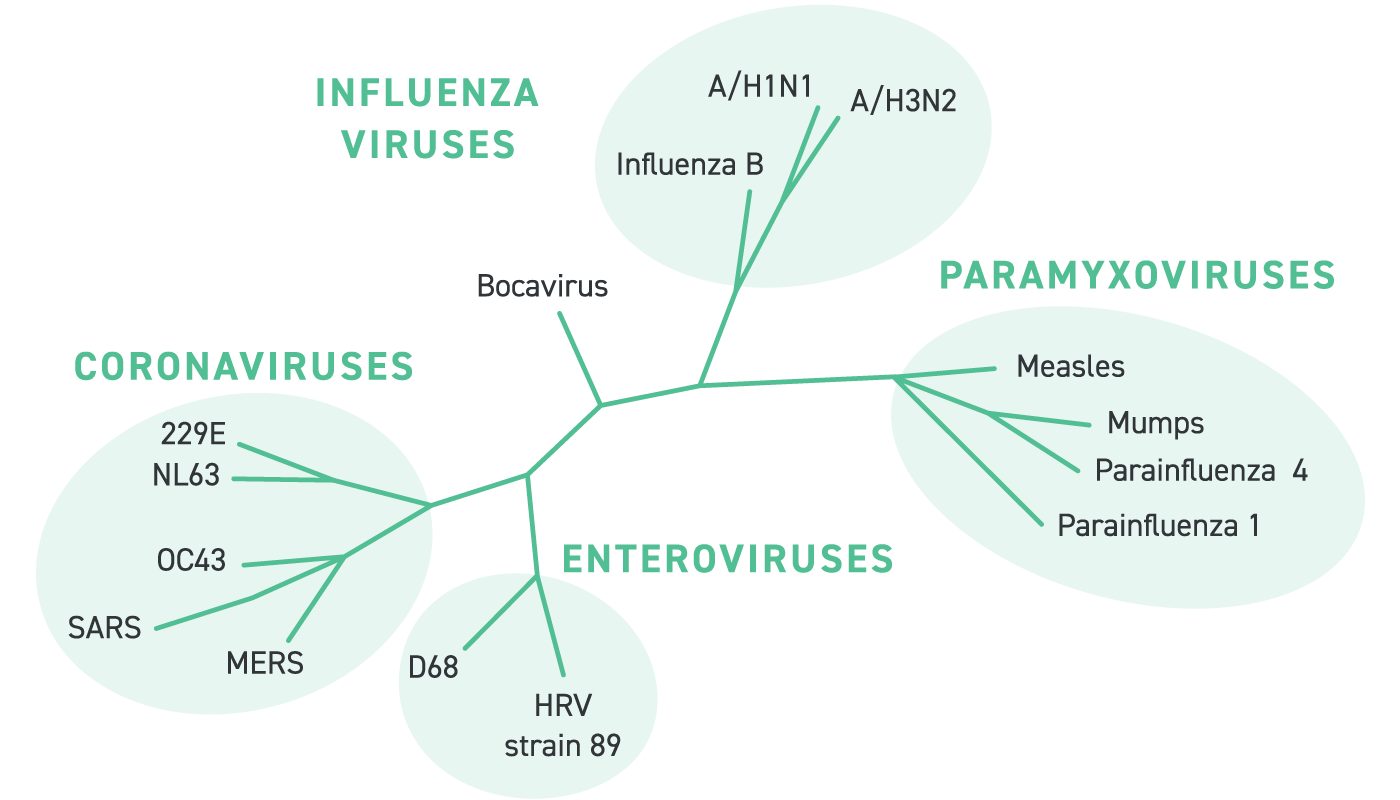
Das Design dieses Panels beinhaltet die Verwendung des CATCH-Algorithmus (Compact Aggregation of Targets for Comprehensive Hybridization). Weitere Einzelheiten zu CATCH finden Sie unter Metsky, H.C. und Siddle, K.J. et al., „Capturing sequence diversity in metagenomes with comprehensive and scalable probe design“. Nature Biotechnology“, 37(2), 160–168 (2019). DOI: 10,1038/ s41587-018-0006-x
Einblicke in die Sequenzierung viraler Erreger, die mit akuten Atemwegsinfektionen in Verbindung stehen
In den Atemwegen können je nach Wirtsumgebung viele verschiedene Arten von kommensalen und pathogenen Organismen vorkommen. Der Nachweis einer Spezies allein bietet möglicherweise nicht den vollständigsten Überblick über die biologische Analyse einer Probe. Das Twist Respiratory Virus Research Panel ermöglicht die Anreicherung und Sequenzierung viraler Erreger von Atemwegserkrankungen, so dass Forscher die Spezies nachweisen und Sequenzinformationen über die viralen Genome erhalten können.
Außerdem können sich die Symptome einer akuten Atemwegsinfektion mit denen anderer Atemwegsviren überschneiden. Das Twist Respiratory Virus Research Panel kann Forschern helfen, die Infektionsursache zu ermitteln und Koinfektionen festzustellen.

Das Design dieses Panels beinhaltet die Verwendung des CATCH-Algorithmus (Compact Aggregation of Targets for Comprehensive Hybridization). Weitere Einzelheiten zu CATCH finden Sie unter Metsky, H.C. und Siddle, K.J. et al., „Capturing sequence diversity in metagenomes with comprehensive and scalable probe design“. Nature Biotechnology“, 37(2), 160–168 (2019). DOI: 10,1038/ s41587-018-0006-x
Die 41.047 Sonden in diesem Panel, die aus der GenBank-Datenbank zusammengestellt wurden, werden es Forschern ermöglichen, COVID-19-Symptome von denen anderer Atemwegserkrankungen, sowohl Influenza- als auch Nicht-Influenza-bezogen, zu trennen. Das Twist Respiratory Panel ermöglicht die Anreicherung dieser viralen Sequenzen zur hoch aufgelösten NGS. Dies führt zu einem hochempfindlichen Nachweis von nur 100 Kopien viralen Materials mit einer mehr als 5000-fachen Anreicherung, selbst bei schwierigen Proben, und einer Abdeckung von > 99,9 % des Genoms bei 1X oder mehr nach der Anreicherung.
Jede synthetische Viruskontrolle von Twist wurde vor der cDNA-Synthese zu 50 ng der humanen Carrier-RNA bei 1.000.000 Kopien hinzugefügt. Für jeden Standard zeigen wir den prozentualen Anteil der viralen On-Target-Reads, das Fold Enrichment vor der Eingabe, den Median der Abdeckung über das Genom, den prozentualen Anteil der Basen mit mindestens 30-facher Abdeckung und die Vorlagenlänge. Virusgenome sind nach Vorlagenlänge (kleinstes bis längstes) unter allen Genomsegmenten angeordnet. In den meisten Fällen fanden wir heraus, dass über 70 Prozent der Reads von Virusgenomen in diesen Libraries stammten, was eine mindestens 2500-fache Anreicherung gegenüber dem zugefügten Inhalt darstellt.
Ein Vergleich des Prozentsatzes der viralen On-Target-Reads, die durch eine synthetische Viruskontrolle von Twist bei verschiedenen Virustitern erfasst wurden, unter Verwendung einer Multiplex- und Singleplex-Hybridisierungsreaktion. Die erste Grafik zeigt Vergleiche zwischen einer Multiplex- und Singleplex-Hybridisierungsreaktion unter Verwendung von 1.000.000 Kopien pro Library. Die zweite Grafik zeigt einen ähnlichen Vergleich zwischen 100, 10.000 und 1.000.000 Kopien pro Library. Jede Viruskontrolle wurde mit dem Twist Standard Target Enrichment-Workflow und dem Twist Respiratory Virus Research Panel erfasst. Diese Daten zeigen, dass die 8-Plex-Erfassung bei Verwendung des Twist Respiratory Virus Research Panel eine vergleichbar effiziente Anreicherung wie eine Einzelplex-Erfassung ermöglicht.
Die 41.047 Sonden in diesem Panel, die aus der GenBank-Datenbank zusammengestellt wurden, werden es Forschern ermöglichen, COVID-19-Symptome von denen anderer Atemwegserkrankungen, sowohl Influenza- als auch Nicht-Influenza-bezogen, zu trennen. Das Twist Respiratory Panel ermöglicht die Anreicherung dieser viralen Sequenzen zur hoch aufgelösten NGS. Dies führt zu einem hochempfindlichen Nachweis von nur 100 Kopien viralen Materials mit einer mehr als 5000-fachen Anreicherung, selbst bei schwierigen Proben, und einer Abdeckung von > 99,9 % des Genoms bei 1X oder mehr nach der Anreicherung.
Jede synthetische Viruskontrolle von Twist wurde vor der cDNA-Synthese zu 50 ng der humanen Carrier-RNA bei 1.000.000 Kopien hinzugefügt. Für jeden Standard zeigen wir den prozentualen Anteil der viralen On-Target-Reads, das Fold Enrichment vor der Eingabe, den Median der Abdeckung über das Genom, den prozentualen Anteil der Basen mit mindestens 30-facher Abdeckung und die Vorlagenlänge. Virusgenome sind nach Vorlagenlänge (kleinstes bis längstes) unter allen Genomsegmenten angeordnet. In den meisten Fällen fanden wir heraus, dass über 70 Prozent der Reads von Virusgenomen in diesen Libraries stammten, was eine mindestens 2500-fache Anreicherung gegenüber dem zugefügten Inhalt darstellt.
Ein Vergleich des Prozentsatzes der viralen On-Target-Reads, die durch eine synthetische Viruskontrolle von Twist bei verschiedenen Virustitern erfasst wurden, unter Verwendung einer Multiplex- und Singleplex-Hybridisierungsreaktion. Die erste Grafik zeigt Vergleiche zwischen einer Multiplex- und Singleplex-Hybridisierungsreaktion unter Verwendung von 1.000.000 Kopien pro Library. Die zweite Grafik zeigt einen ähnlichen Vergleich zwischen 100, 10.000 und 1.000.000 Kopien pro Library. Jede Viruskontrolle wurde mit dem Twist Standard Target Enrichment-Workflow und dem Twist Respiratory Virus Research Panel erfasst. Diese Daten zeigen, dass die 8-Plex-Erfassung bei Verwendung des Twist Respiratory Virus Research Panel eine vergleichbar effiziente Anreicherung wie eine Einzelplex-Erfassung ermöglicht.
Das Twist Respiratory Virus Research Panel gewährt Zugriff zum cloudbasierten Tool One Codex. Ideal für den Einsatz in Forschung und Klinik, da es zur Erstellung von publikationsreifen Grafiken und druckfähigen Berichten für Labore verwendet werden kann, die laborentwickelte Tests vorbereiten.
Bei der Anwendung des Twist Respiratory Virus Research Panel können alle Kunden auf die One Codex-Analyse-Plattform zugreifen. Diese Plattform ist ideal für sowohl Forschungs- als auch klinische Anwendungsfälle geeignet, inklusive der Erzeugung von publikationsreifen Grafiken, teilbaren Berichten und anderen Analyse-Tools. Wenn ein Panel gekauft wird, werden Onboarding-Anweisungen und Credits zur Probenanalyse mit der One Codex-Plattform bereitgestellt. Erfahren Sie mehr über die mikrobielle Probenanalyse-Lösung von One Codex.
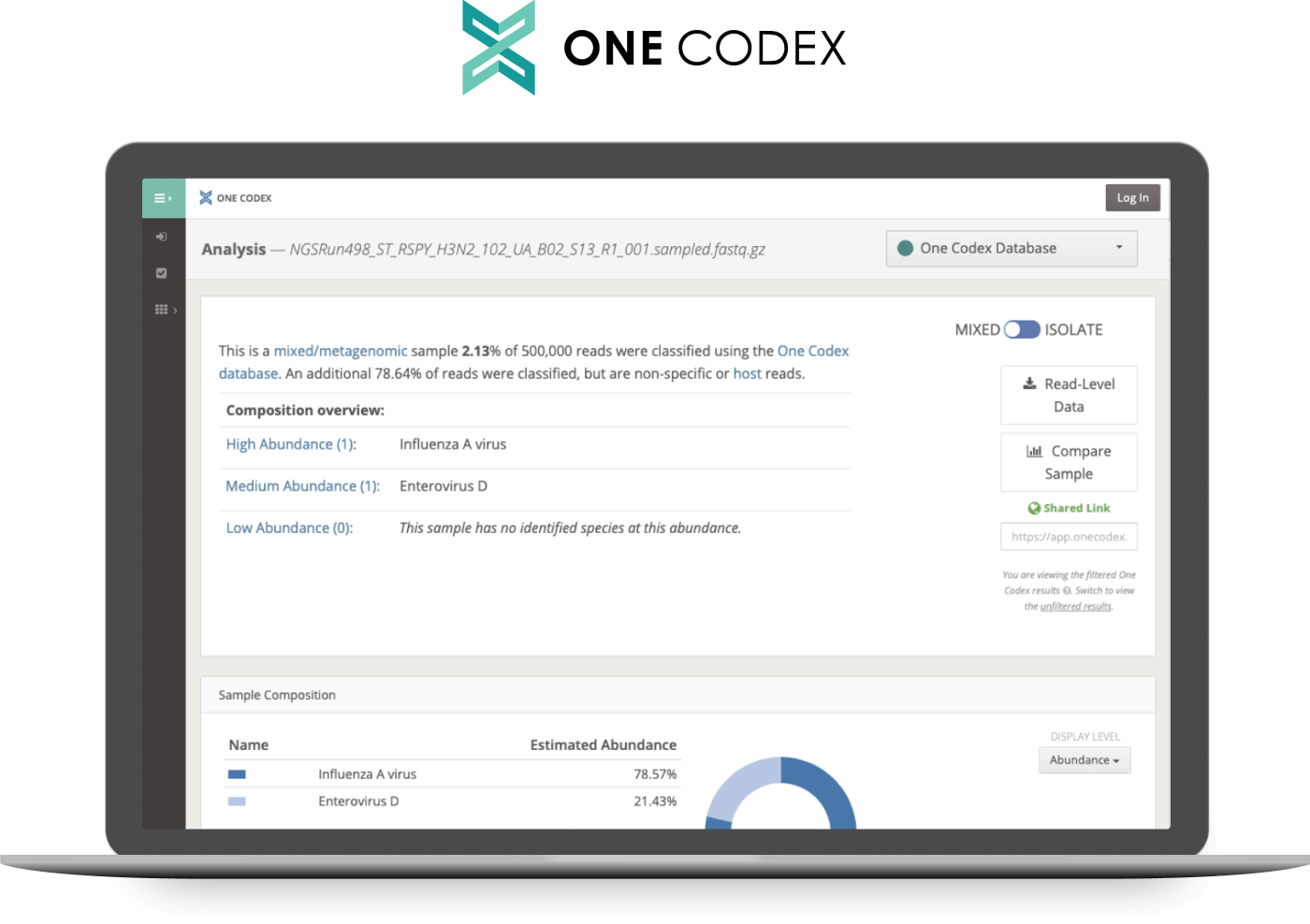
Das Twist Respiratory Virus Research Panel gewährt Zugriff zum cloudbasierten Tool One Codex. Ideal für den Einsatz in Forschung und Klinik, da es zur Erstellung von publikationsreifen Grafiken und druckfähigen Berichten für Labore verwendet werden kann, die laborentwickelte Tests vorbereiten.
Bei der Anwendung des Twist Respiratory Virus Research Panel können alle Kunden auf die One Codex-Analyse-Plattform zugreifen. Diese Plattform ist ideal für sowohl Forschungs- als auch klinische Anwendungsfälle geeignet, inklusive der Erzeugung von publikationsreifen Grafiken, teilbaren Berichten und anderen Analyse-Tools. Wenn ein Panel gekauft wird, werden Onboarding-Anweisungen und Credits zur Probenanalyse mit der One Codex-Plattform bereitgestellt. Erfahren Sie mehr über die mikrobielle Probenanalyse-Lösung von One Codex.

103066
Twist Respiratory Virus Research Panel mit One Codex Software, 2 Reaktionen, Kit103067
Twist Respiratory Virus Research Panel mit One Codex Software, 12 Reaktionen, Kit103068
Twist Respiratory Virus Research Panel mit One Codex Software, 96 Reaktionen, KitFormat
dsDNAPanelgröße
41047 SondenInhalt
Auf Basis von 29 Atemwegsvirus-ReferenzsequenzenDesigndatenbanken
RefSeq & GenBankLagerung bei
-5 bis -30 °CDas Twist Respiratory Virus Research Panel ist nur für Forschungszwecke bestimmt und unterliegt zusätzlichen Nutzungsbeschränkungen, die in den Lieferbedingungen von Twist festgelegt sind.
103066
Twist Respiratory Virus Research Panel mit One Codex Software, 2 Reaktionen, Kit103067
Twist Respiratory Virus Research Panel mit One Codex Software, 12 Reaktionen, Kit103068
Twist Respiratory Virus Research Panel mit One Codex Software, 96 Reaktionen, KitFormat
dsDNAPanelgröße
41047 SondenInhalt
Auf Basis von 29 Atemwegsvirus-ReferenzsequenzenDesigndatenbanken
RefSeq & GenBankLagerung bei
-5 bis -30 °CDas Twist Respiratory Virus Research Panel ist nur für Forschungszwecke bestimmt und unterliegt zusätzlichen Nutzungsbeschränkungen, die in den Lieferbedingungen von Twist festgelegt sind.
