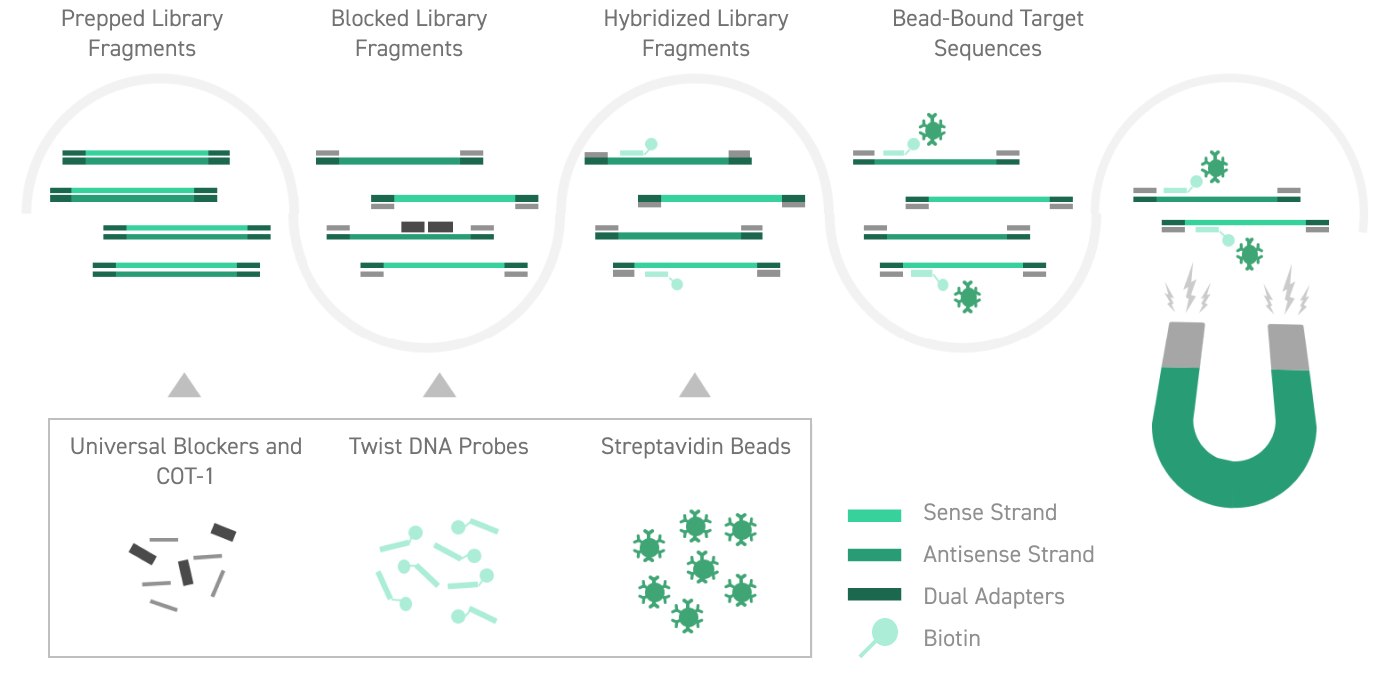
Die Optimierung der Effizienz von NGS beinhaltet oft ein Target Enrichment von genomischen Regionen von Interesse vor der Sequenzierung. Im Arbeitsablauf des Target Enrichment von Twist Bioscience werden biotinylierte synthetische DNA-Sonden so konzipiert, dass sie an Exons oder andere benutzerdefinierte genomische Regionen hybridisieren. Nach der Hybridisierung mit einer genomischen DNA-Probe werden die Sonden purifiziert, um eine Probe zu erzeugen, die für die Zielbereiche angereichert ist.
Die Twist Universal Blockers verbessern die Spezifizität des Target Capture durch die Blockierung der nicht-spezifischen Hybridisierung zwischen Adapter-Sequenzen. Die Blockierung unspezifischer Wechselwirkungen während der Hybridisierung vereinfacht die Versuchsplanung und -durchführung, senkt die Sequenzierungskosten und verbessert die Ergebnisse gezielter Sequenzierungsversuche.
Die Optimierung der Effizienz von NGS beinhaltet oft ein Target Enrichment von genomischen Regionen von Interesse vor der Sequenzierung. Im Arbeitsablauf des Target Enrichment von Twist Bioscience werden biotinylierte synthetische DNA-Sonden so konzipiert, dass sie an Exons oder andere benutzerdefinierte genomische Regionen hybridisieren. Nach der Hybridisierung mit einer genomischen DNA-Probe werden die Sonden purifiziert, um eine Probe zu erzeugen, die für die Zielbereiche angereichert ist.
Die Twist Universal Blockers verbessern die Spezifizität des Target Capture durch die Blockierung der nicht-spezifischen Hybridisierung zwischen Adapter-Sequenzen. Die Blockierung unspezifischer Wechselwirkungen während der Hybridisierung vereinfacht die Versuchsplanung und -durchführung, senkt die Sequenzierungskosten und verbessert die Ergebnisse gezielter Sequenzierungsversuche.
Blockierung der Off-Target-Anbindung zur Verbesserung des On-Target-Capture
Twist Universal Blockers enthalten eine proprietäre Mischung aus universellen Adapter-Sequenzen1. Diese Sequenzen binden genomische Library-Fragmente, um die Cross-Hybridisierung zwischen TruSeq-kompatiblen Adaptern zu blockieren und die On-Target-Capture-Raten zu verbessern. Twist Universal Blockers können die Performance aller Target Enrichment-Workflows verbessern, die TruSeq-kompatible Adapter verwenden.
Das robuste Design der Twist Universal Blockers maximiert die Flexibilität für einen effizienteren Target Enrichment-Workflow. Sie sind mit allen Twist Target Enrichment Panels kompatibel und verbessern die Erfassung von On-Target-Reads unabhängig vom Indexsequenztyp, in Singleplex- und Multiplex-Target Enrichment Workflows und unabhängig von der Panelgröße. Twist Universal Blockers ermöglichen die Flexibilität, das beste Adapter-Indexdesign für Ihr Experiment auszuwählen, ohne die Target Enrichment-Performance zu beeinträchtigen.
Die Leistung hängt nicht vom Indexdesign ab. On-Target-Leistung der Twist Universal Blockers über eine Vielzahl von Single- und Dual-Index-Truseq-kompatiblen Adaptern hinweg. Einzelne Bibliotheken wurden aus einer einzigen genomischen Quelle (NA12878; Coriell) und TruSeq-kompatiblen Adaptern mit Indexlängen von 6 bis 14 bp (eindeutige molekulare Kennung [Unique Molecular Identifier, UMI] nicht eingeschlossen) generiert. Das Hybrid Capture wurde ohne bzw. mit Twist Universal Blockers unter der Verwendung von Exom-Target Enrichment-Panels (33,1 Mb; Twist Bioscience) und 500 ng gDNA pro individueller Library den Empfehlungen des Herstellers entsprechend für 16-stündige Hybridisierungsreaktionen durchgeführt. Cot-DNA war in allen Proben vorhanden. Die Sequenzierung wurde mit einem NextSeq 500/550 High Output v2 Kit durchgeführt, um 2x76 gekoppelte End-Reads zu generieren. Die Daten wurden auf das 150-fache der Zielgröße heruntergerechnet und mit Picard Metrics mit einer Mapping-Qualität von 20 analysiert. Der Prozentsatz der Basen On-Bait umfasste sowohl On-Bait- als auch Near-Bait-Basen und wurde durch die Gleichung 1 - PCT_OFF_BAIT definiert. Die Fehlerbalken stellen eine Standardabweichung oder Beobachtungsbereiche dar; N ≥ 2.
Die Leistung hängt von der Panelgröße ab. On-Target-Leistung der Twist Universal Blockers über eine Reihe von Panelgrößen. Individuelle Libraries wurden aus einer einzelnen Genomquelle (NA12878; Coriell) und 8 bp TruSeq-kompatiblen Adaptern mit Dualindex generiert. Das Hybrid Capture wurde ohne bzw. mit Twist Universal Blockers unter Verwendung von Target Enrichment-Panels verschiedener Größen (0,04 Mb bis 33,1 Mb; Twist Bioscience) und 500 ng gDNA pro individueller Library den Empfehlungen des Herstellers entsprechend für 16-stündige Hybridisierungsreaktionen durchgeführt. Cot-DNA war in allen Proben vorhanden. Die Sequenzierung wurde mit einem NextSeq 500/550 High Output v2 Kit durchgeführt, um 2x76 gekoppelte End-Reads zu generieren. Die Daten wurden auf das 150-fache der Zielgröße heruntergerechnet und mit Picard Metrics mit einer Mapping-Qualität von 20 analysiert. Der Prozentsatz der Basen On-Bait umfasste sowohl On-Bait- als auch Near-Bait-Basen und wurde durch die Gleichung 1 - PCT_OFF_BAIT definiert. Fehlerbalken stellen die Beobachtungsbereiche dar; N = 2.
Blockierung der Off-Target-Anbindung zur Verbesserung des On-Target-Capture
Twist Universal Blockers enthalten eine proprietäre Mischung aus universellen Adapter-Sequenzen1. Diese Sequenzen binden genomische Library-Fragmente, um die Cross-Hybridisierung zwischen TruSeq-kompatiblen Adaptern zu blockieren und die On-Target-Capture-Raten zu verbessern. Twist Universal Blockers können die Performance aller Target Enrichment-Workflows verbessern, die TruSeq-kompatible Adapter verwenden.
Das robuste Design der Twist Universal Blockers maximiert die Flexibilität für einen effizienteren Target Enrichment-Workflow. Sie sind mit allen Twist Target Enrichment Panels kompatibel und verbessern die Erfassung von On-Target-Reads unabhängig vom Indexsequenztyp, in Singleplex- und Multiplex-Target Enrichment Workflows und unabhängig von der Panelgröße. Twist Universal Blockers ermöglichen die Flexibilität, das beste Adapter-Indexdesign für Ihr Experiment auszuwählen, ohne die Target Enrichment-Performance zu beeinträchtigen.

Die Leistung hängt nicht vom Indexdesign ab. On-Target-Leistung der Twist Universal Blockers über eine Vielzahl von Single- und Dual-Index-Truseq-kompatiblen Adaptern hinweg. Einzelne Bibliotheken wurden aus einer einzigen genomischen Quelle (NA12878; Coriell) und TruSeq-kompatiblen Adaptern mit Indexlängen von 6 bis 14 bp (eindeutige molekulare Kennung [Unique Molecular Identifier, UMI] nicht eingeschlossen) generiert. Das Hybrid Capture wurde ohne bzw. mit Twist Universal Blockers unter der Verwendung von Exom-Target Enrichment-Panels (33,1 Mb; Twist Bioscience) und 500 ng gDNA pro individueller Library den Empfehlungen des Herstellers entsprechend für 16-stündige Hybridisierungsreaktionen durchgeführt. Cot-DNA war in allen Proben vorhanden. Die Sequenzierung wurde mit einem NextSeq 500/550 High Output v2 Kit durchgeführt, um 2x76 gekoppelte End-Reads zu generieren. Die Daten wurden auf das 150-fache der Zielgröße heruntergerechnet und mit Picard Metrics mit einer Mapping-Qualität von 20 analysiert. Der Prozentsatz der Basen On-Bait umfasste sowohl On-Bait- als auch Near-Bait-Basen und wurde durch die Gleichung 1 - PCT_OFF_BAIT definiert. Die Fehlerbalken stellen eine Standardabweichung oder Beobachtungsbereiche dar; N ≥ 2.
Die Leistung hängt von der Panelgröße ab. On-Target-Leistung der Twist Universal Blockers über eine Reihe von Panelgrößen. Individuelle Libraries wurden aus einer einzelnen Genomquelle (NA12878; Coriell) und 8 bp TruSeq-kompatiblen Adaptern mit Dualindex generiert. Das Hybrid Capture wurde ohne bzw. mit Twist Universal Blockers unter Verwendung von Target Enrichment-Panels verschiedener Größen (0,04 Mb bis 33,1 Mb; Twist Bioscience) und 500 ng gDNA pro individueller Library den Empfehlungen des Herstellers entsprechend für 16-stündige Hybridisierungsreaktionen durchgeführt. Cot-DNA war in allen Proben vorhanden. Die Sequenzierung wurde mit einem NextSeq 500/550 High Output v2 Kit durchgeführt, um 2x76 gekoppelte End-Reads zu generieren. Die Daten wurden auf das 150-fache der Zielgröße heruntergerechnet und mit Picard Metrics mit einer Mapping-Qualität von 20 analysiert. Der Prozentsatz der Basen On-Bait umfasste sowohl On-Bait- als auch Near-Bait-Basen und wurde durch die Gleichung 1 - PCT_OFF_BAIT definiert. Fehlerbalken stellen die Beobachtungsbereiche dar; N = 2.
Workflow-Schritt
Target EnrichmentLagerung
Bei -5 °C bis -30 °C lagern100856
Twist Universal Blockers, 2 Reaktionen100578
Twist Universal Blockers, 12 Reaktionen100767
Twist Universal Blockers, 96 ReaktionenDie Twist Universal Blockers sind nur für Forschungszwecke bestimmt und unterliegen zusätzlichen Nutzungsbeschränkungen, wie in den Twist Bioscience Lieferbedingungen dargelegt.
Workflow-Schritt
Target EnrichmentLagerung
Bei -5 °C bis -30 °C lagern100856
Twist Universal Blockers, 2 Reaktionen100578
Twist Universal Blockers, 12 Reaktionen100767
Twist Universal Blockers, 96 ReaktionenDie Twist Universal Blockers sind nur für Forschungszwecke bestimmt und unterliegen zusätzlichen Nutzungsbeschränkungen, wie in den Twist Bioscience Lieferbedingungen dargelegt.
