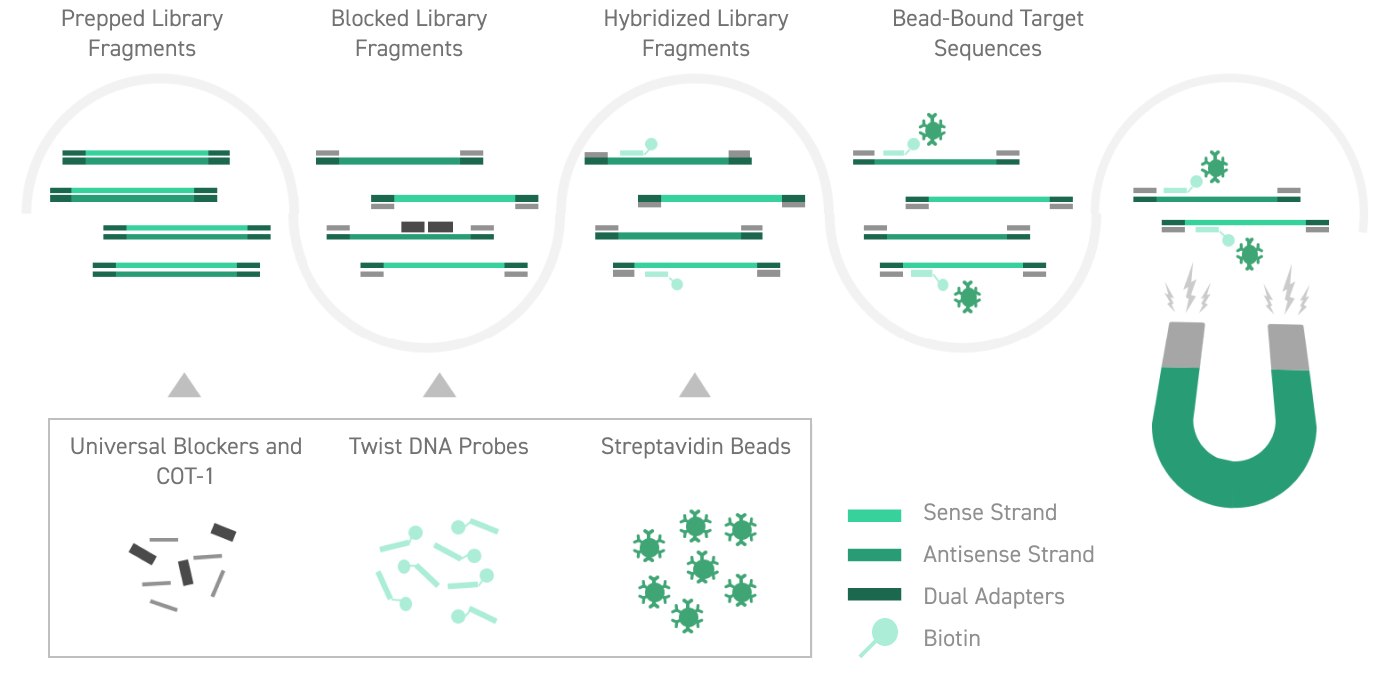
La optimización de la eficiencia de la secuenciación de última generación (NGS) suele conllevar el enriquecimiento dirigido de regiones genómicas de interés antes de la secuenciación. En el flujo de trabajo de enriquecimiento del objetivo Twist Bioscience, las sondas de ADN sintético biotinilado están diseñadas para hibridar a exones u otras regiones genómicas personalizadas. Tras la hibridación con una muestra de ADN genómico, las sondas se purifican para producir una muestra enriquecida para las regiones objetivo.
Los bloqueadores universales Twist mejoran la especificidad de la captura de objetivos bloqueando la hibridación no específica entre secuencias adaptadoras. El bloqueo de interacciones no específicas durante la hibridación simplifica el diseño y la ejecución experimentales, reduce los costes de secuenciación y mejora los resultados de los esfuerzos de secuenciación dirigidos.
La optimización de la eficiencia de la secuenciación de última generación (NGS) suele conllevar el enriquecimiento dirigido de regiones genómicas de interés antes de la secuenciación. En el flujo de trabajo de enriquecimiento del objetivo Twist Bioscience, las sondas de ADN sintético biotinilado están diseñadas para hibridar a exones u otras regiones genómicas personalizadas. Tras la hibridación con una muestra de ADN genómico, las sondas se purifican para producir una muestra enriquecida para las regiones objetivo.
Los bloqueadores universales Twist mejoran la especificidad de la captura de objetivos bloqueando la hibridación no específica entre secuencias adaptadoras. El bloqueo de interacciones no específicas durante la hibridación simplifica el diseño y la ejecución experimentales, reduce los costes de secuenciación y mejora los resultados de los esfuerzos de secuenciación dirigidos.
Bloqueo de las uniones fuera de objetivo para mejorar la captura en el objetivo
Los bloqueadores universales Twist contienen una mezcla patentada de secuencias adaptadoras universales1. Estas secuencias unen fragmentos de bibliotecas genómicas para bloquear la hibridación cruzada entre los adaptadores compatibles con TruSeq y mejoran las tasas de captura en el objetivo. Los bloqueadores universales Twist pueden mejorar el rendimiento de cualquier flujo de trabajo de enriquecimiento del objetivo que utilice adaptadores compatibles con TruSeq.
El diseño sólido de los bloqueadores universales Twist maximiza la flexibilidad para un flujo de trabajo de enriquecimiento del objetivo más eficiente. Compatible con todos los paneles de enriquecimiento del objetivo Twist, mejoran la captura de lecturas en el objetivo con independencia del tipo de secuencia de índice, en flujos de trabajo de enriquecimiento del objetivo multiplexados y monoplexados, y con independencia del tamaño del panel. Los bloqueadores universales Twist le proporcionan la flexibilidad de elegir el mejor diseño de índice de adaptadores para el experimento sin afectación del rendimiento del enriquecimiento del objetivo.
El rendimiento es independiente del diseño de índice. El rendimiento en el objetivo de los bloqueadores universales Twist en diversos adaptadores compatibles con TruSeq de índice sencillo o doble. Las bibliotecas individuales se generaron a partir de una fuente genómica sencilla (NA12878; Coriell) y adaptadores compatibles con TruSeq con longitudes de índice que van desde los 6 hasta los 14 bp (longitud del identificador molecular único [unique molecular identifier, UMI] no incluida). Se realizó la captura híbrida en ausencia o presencia de bloqueadores universales Twist con un panel de enriquecimiento del objetivo del exoma (33,1 Mb; Twist Bioscience) con 500 ng de ADNg por biblioteca individual siguiendo las recomendaciones del fabricante para reacciones de hibridación de 16 horas. El ADN Cot estuvo presente en todas las muestras. La secuenciación se realizó con un kit NextSeq 500/550 v2 de alto rendimiento para generar 2 x 76 lecturas de extremos emparejados. Se submuestrearon los datos a 150x el tamaño del objetivo y se analizaron mediante las métricas de Picard con una calidad de mapeado de 20. El porcentaje de bases en cebo incluye las bases en cebo y cerca del cebo, y se definen con la ecuación 1 - PCT_OFF_BAIT. Las barras de error denotan una desviación estándar o intervalo de observaciones de N ≥ 2.
El rendimiento es independiente del tamaño del panel. Rendimiento en el objetivo de los bloqueadores universales Twist en diversos tamaños de panel. Las bibliotecas individuales se generaron a partir de una fuente genómica única (NA12878; Coriell) y adaptadores compatibles con TruSeq de doble índice de 8 pb. Se realizó la captura híbrida en ausencia o presencia de bloqueadores universales Twist con paneles de enriquecimiento del objetivo de distintos tamaños (de 0,04 Mb a 33,1 Mb; Twist Bioscience) y 500 ng de ADNg por biblioteca individual siguiendo las recomendaciones del fabricante para reacciones de hibridación de 16 horas. El ADN Cot estuvo presente en todas las muestras. La secuenciación se realizó con un kit NextSeq 500/550 v2 de alto rendimiento para generar 2 x 76 lecturas de extremos emparejados. Se submuestrearon los datos a 150x el tamaño del objetivo y se analizaron mediante las métricas de Picard con una calidad de mapeado de 20. El porcentaje de bases en cebo incluye las bases en cebo y cerca del cebo, y se definen con la ecuación 1 - PCT_OFF_BAIT. Las barras de error denotan un intervalo de observaciones N = 2.
Bloqueo de las uniones fuera de objetivo para mejorar la captura en el objetivo
Los bloqueadores universales Twist contienen una mezcla patentada de secuencias adaptadoras universales1. Estas secuencias unen fragmentos de bibliotecas genómicas para bloquear la hibridación cruzada entre los adaptadores compatibles con TruSeq y mejoran las tasas de captura en el objetivo. Los bloqueadores universales Twist pueden mejorar el rendimiento de cualquier flujo de trabajo de enriquecimiento del objetivo que utilice adaptadores compatibles con TruSeq.
El diseño sólido de los bloqueadores universales Twist maximiza la flexibilidad para un flujo de trabajo de enriquecimiento del objetivo más eficiente. Compatible con todos los paneles de enriquecimiento del objetivo Twist, mejoran la captura de lecturas en el objetivo con independencia del tipo de secuencia de índice, en flujos de trabajo de enriquecimiento del objetivo multiplexados y monoplexados, y con independencia del tamaño del panel. Los bloqueadores universales Twist le proporcionan la flexibilidad de elegir el mejor diseño de índice de adaptadores para el experimento sin afectación del rendimiento del enriquecimiento del objetivo.

El rendimiento es independiente del diseño de índice. El rendimiento en el objetivo de los bloqueadores universales Twist en diversos adaptadores compatibles con TruSeq de índice sencillo o doble. Las bibliotecas individuales se generaron a partir de una fuente genómica sencilla (NA12878; Coriell) y adaptadores compatibles con TruSeq con longitudes de índice que van desde los 6 hasta los 14 bp (longitud del identificador molecular único [unique molecular identifier, UMI] no incluida). Se realizó la captura híbrida en ausencia o presencia de bloqueadores universales Twist con un panel de enriquecimiento del objetivo del exoma (33,1 Mb; Twist Bioscience) con 500 ng de ADNg por biblioteca individual siguiendo las recomendaciones del fabricante para reacciones de hibridación de 16 horas. El ADN Cot estuvo presente en todas las muestras. La secuenciación se realizó con un kit NextSeq 500/550 v2 de alto rendimiento para generar 2 x 76 lecturas de extremos emparejados. Se submuestrearon los datos a 150x el tamaño del objetivo y se analizaron mediante las métricas de Picard con una calidad de mapeado de 20. El porcentaje de bases en cebo incluye las bases en cebo y cerca del cebo, y se definen con la ecuación 1 - PCT_OFF_BAIT. Las barras de error denotan una desviación estándar o intervalo de observaciones de N ≥ 2.
El rendimiento es independiente del tamaño del panel. Rendimiento en el objetivo de los bloqueadores universales Twist en diversos tamaños de panel. Las bibliotecas individuales se generaron a partir de una fuente genómica única (NA12878; Coriell) y adaptadores compatibles con TruSeq de doble índice de 8 pb. Se realizó la captura híbrida en ausencia o presencia de bloqueadores universales Twist con paneles de enriquecimiento del objetivo de distintos tamaños (de 0,04 Mb a 33,1 Mb; Twist Bioscience) y 500 ng de ADNg por biblioteca individual siguiendo las recomendaciones del fabricante para reacciones de hibridación de 16 horas. El ADN Cot estuvo presente en todas las muestras. La secuenciación se realizó con un kit NextSeq 500/550 v2 de alto rendimiento para generar 2 x 76 lecturas de extremos emparejados. Se submuestrearon los datos a 150x el tamaño del objetivo y se analizaron mediante las métricas de Picard con una calidad de mapeado de 20. El porcentaje de bases en cebo incluye las bases en cebo y cerca del cebo, y se definen con la ecuación 1 - PCT_OFF_BAIT. Las barras de error denotan un intervalo de observaciones N = 2.
Paso de flujo de trabajo
Enriquecimiento del objetivoAlmacenamiento
Almacenar a de -5 a -30 °C100856
Bloqueadores universales twist, 2 reacciones100578
Bloqueadores universales twist, 12 reacciones100767
Bloqueadores universales twist, 96 reaccionesLos bloqueadores universales Twist están diseñados únicamente para su uso en investigación y están sujetos a restricciones de uso adicionales establecidas en los Términos y condiciones de suministro de Twist.
Paso de flujo de trabajo
Enriquecimiento del objetivoAlmacenamiento
Almacenar a de -5 a -30 °C100856
Bloqueadores universales twist, 2 reacciones100578
Bloqueadores universales twist, 12 reacciones100767
Bloqueadores universales twist, 96 reaccionesLos bloqueadores universales Twist están diseñados únicamente para su uso en investigación y están sujetos a restricciones de uso adicionales establecidas en los Términos y condiciones de suministro de Twist.
