Erschließen Sie sich einen unerkundeten Enzym-Sequenzabschnitt für eine optimale Exploration
Unsere neuartigen Enzymsequenzen wurden aus verschiedenen nicht kultivierten mikrobiellen Einzelzellgenomen identifiziert und mit Hilfe von auf Machine Learning basierenden Modellen, die auf Strukturvorhersage, strukturbasiertem Clustering und Filterung beruhen, nach wünschenswerten Eigenschaften ausgewählt – diese Sequenzen werden Sie wahrscheinlich nirgendwo anders finden.
Transaminasen (TAs) sind Enzyme, die die Übertragung einer Aminogruppe von einem Aminodonor auf ein Keton oder Aldehyd katalysieren. Diese Fähigkeit, die stereoselektive Aminierung prochiraler Ketone zu erleichtern, ist für die pharmazeutische Produktion von großem Wert, da die Nachfrage nach chiralen Aminen als Bausteine für pharmazeutische Wirkstoffe (APIs) hoch ist.
Wir haben eine Untergruppe von TA-Enzymen identifiziert und eingegrenzt, die ihre Aktivität unter einer Vielzahl von extremen Bedingungen und bei vielen verschiedenen Substraten aufrechterhalten, was für den pharmazeutischen Herstellungsprozess nützlich ist.
Unser Screening-Kit ermöglicht die Auswahl funktionell relevanter Sequenzen mit der Möglichkeit, diese in Zukunft mit nachgeschalteten Enzym-Engineering-Ressourcen zu kombinieren, um einen effizienteren Weg für das Protein-Engineering zu schaffen.
So werden Enzyme für das Screening ausgewählt
Die bitBiome bit-GEM Datenbank ist die führende Ressource für durchsuchbare und gut nutzbare Mikroben-Genomsequenzen. Die bit-GEM Datenbank, die sich auf die stärksten mikrobiellen Ressourcen der Natur stützt, darunter Proben aus Umgebungen wie Erde, heißen Quellen, Meerwasser, Bergbaustandorten und anderen extremen Bedingungen, enthält eine Vielzahl von Gensequenzen, die mit der bitBiome Plattform für die Einzelzellsequenzierung erfasst wurden. Diese Sequenzierungsmethode gewährleistet eine tiefere Genomabdeckung und ermöglicht die Entdeckung der mikrobiellen „dunklen Materie“. Diese Sequenzen haben eine geringe Ähnlichkeit mit denen im öffentlichen Raum, so dass der Kunde die Freiheit hat, in diesem Sequenzabschnitt zu agieren.



Erschließen Sie sich einen unerkundeten Enzym-Sequenzabschnitt für eine optimale Exploration
Unsere neuartigen Enzymsequenzen wurden aus verschiedenen nicht kultivierten mikrobiellen Einzelzellgenomen identifiziert und mit Hilfe von auf Machine Learning basierenden Modellen, die auf Strukturvorhersage, strukturbasiertem Clustering und Filterung beruhen, nach wünschenswerten Eigenschaften ausgewählt – diese Sequenzen werden Sie wahrscheinlich nirgendwo anders finden.
Transaminasen (TAs) sind Enzyme, die die Übertragung einer Aminogruppe von einem Aminodonor auf ein Keton oder Aldehyd katalysieren. Diese Fähigkeit, die stereoselektive Aminierung prochiraler Ketone zu erleichtern, ist für die pharmazeutische Produktion von großem Wert, da die Nachfrage nach chiralen Aminen als Bausteine für pharmazeutische Wirkstoffe (APIs) hoch ist.
Wir haben eine Untergruppe von TA-Enzymen identifiziert und eingegrenzt, die ihre Aktivität unter einer Vielzahl von extremen Bedingungen und bei vielen verschiedenen Substraten aufrechterhalten, was für den pharmazeutischen Herstellungsprozess nützlich ist.
Unser Screening-Kit ermöglicht die Auswahl funktionell relevanter Sequenzen mit der Möglichkeit, diese in Zukunft mit nachgeschalteten Enzym-Engineering-Ressourcen zu kombinieren, um einen effizienteren Weg für das Protein-Engineering zu schaffen.
So werden Enzyme für das Screening ausgewählt
Die bitBiome bit-GEM Datenbank ist die führende Ressource für durchsuchbare und gut nutzbare Mikroben-Genomsequenzen. Die bit-GEM Datenbank, die sich auf die stärksten mikrobiellen Ressourcen der Natur stützt, darunter Proben aus Umgebungen wie Erde, heißen Quellen, Meerwasser, Bergbaustandorten und anderen extremen Bedingungen, enthält eine Vielzahl von Gensequenzen, die mit der bitBiome Plattform für die Einzelzellsequenzierung erfasst wurden. Diese Sequenzierungsmethode gewährleistet eine tiefere Genomabdeckung und ermöglicht die Entdeckung der mikrobiellen „dunklen Materie“. Diese Sequenzen haben eine geringe Ähnlichkeit mit denen im öffentlichen Raum, so dass der Kunde die Freiheit hat, in diesem Sequenzabschnitt zu agieren.



So werden Enzyme für das Screening ausgewählt
Die bit-GEM Datenbank, die sich auf starke mikrobielle Ressourcen der Natur stützt, darunter Proben aus Umgebungen wie Erde, heißen Quellen, Meerwasser, Bergbaustandorten und anderen extremen Bedingungen, enthält eine Vielzahl von Gensequenzen, die mit der bitBiome Plattform für die Einzelzellsequenzierung erfasst wurden. Diese Sequenzierungsmethode gewährleistet eine tiefere Genomabdeckung und ermöglicht die Entdeckung der mikrobiellen „dunklen Materie“. Diese Sequenzen haben eine geringe Ähnlichkeit mit denen im öffentlichen Raum, was eine größere Handlungsfreiheit innerhalb dieses Sequenzabschnitts ermöglicht. Die Auswahl der 48 Enzyme, die in diesem Kit enthalten sind, wurde nicht nur durch die Möglichkeiten der Twist-DNA-Synthese, sondern auch durch eine gemeinsame bioinformatische Arbeit zur Verfeinerung des funktionalen Sequenzabschnitts unterstützt.
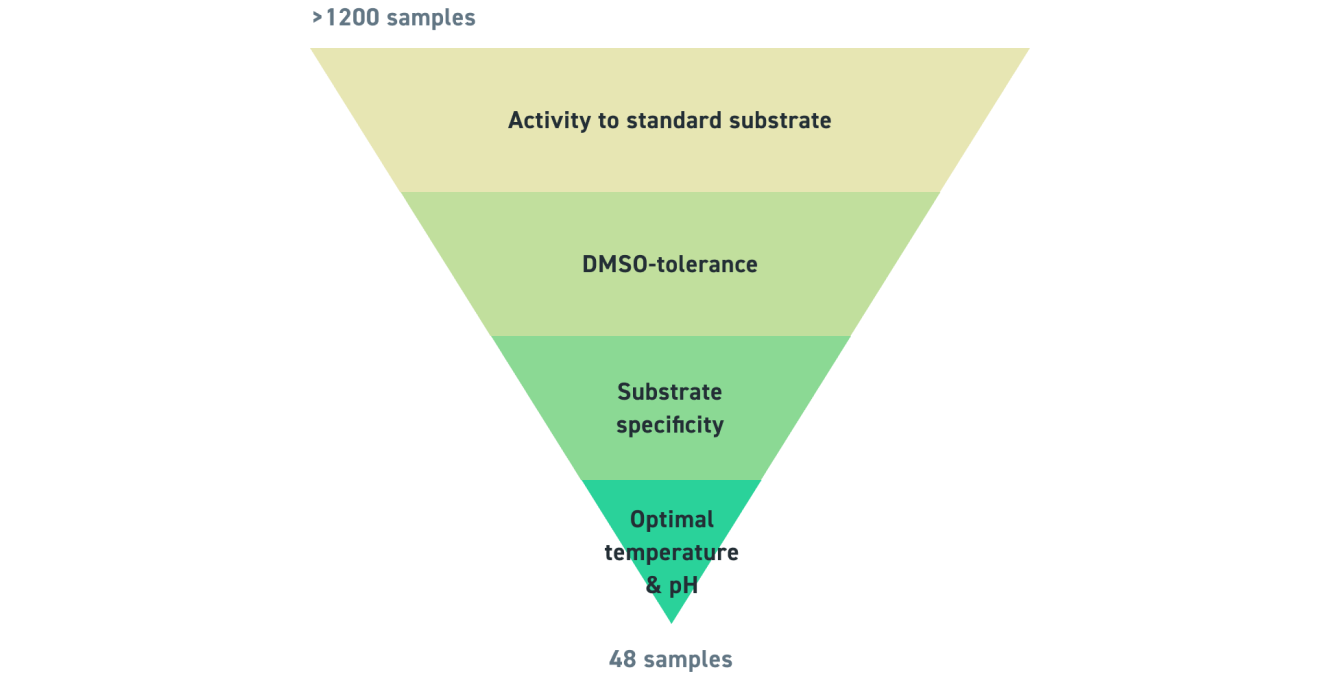
So wurden die Enzyme für unser Kit ausgewählt
Insgesamt wurden 369 aktive Transaminasen mit 19 Substraten identifiziert. 273 der Enzyme erwiesen sich in 10 % DMSO als aktiv mit 30 aktiv bei 60 °C. Schließlich wurden die Enzyme auf pH-Empfindlichkeit und Selektivität getestet.
Kriterien für das Transaminasen-Screening
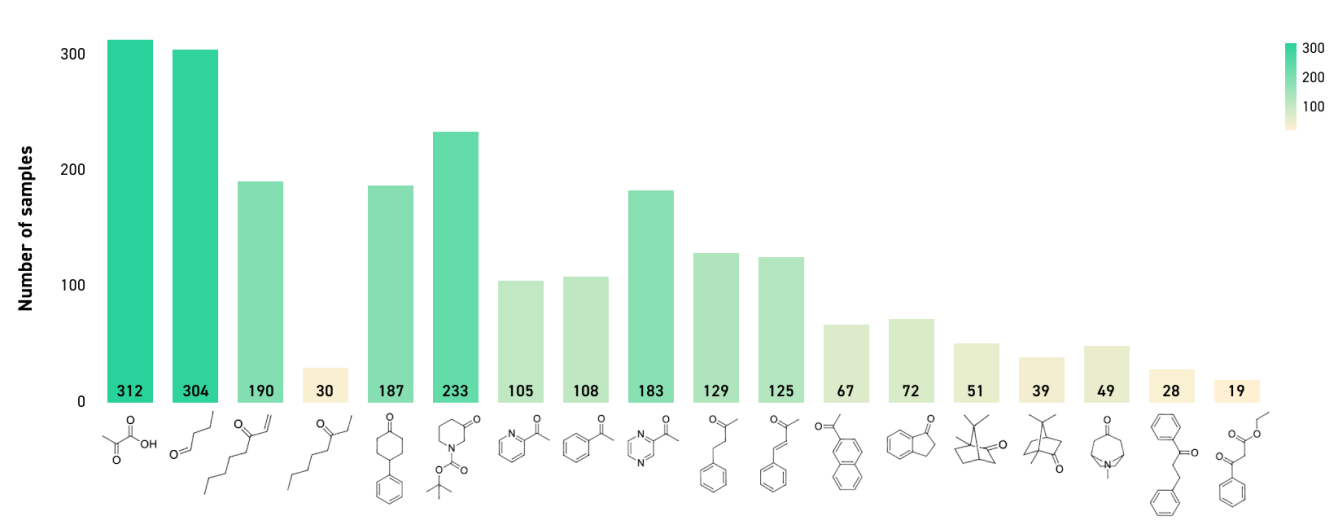
Im Folgenden werden die Enzyme aufgeschlüsselt, die mit verschiedenen Substraten unter den zuvor beschriebenen unterschiedlichen Bedingungen (verschiedene DMSO-Anteile, organische Lösungsmittel, Temperaturen und pH-Werte) getestet wurden.
So werden Enzyme für das Screening ausgewählt
Die bit-GEM Datenbank, die sich auf starke mikrobielle Ressourcen der Natur stützt, darunter Proben aus Umgebungen wie Erde, heißen Quellen, Meerwasser, Bergbaustandorten und anderen extremen Bedingungen, enthält eine Vielzahl von Gensequenzen, die mit der bitBiome Plattform für die Einzelzellsequenzierung erfasst wurden. Diese Sequenzierungsmethode gewährleistet eine tiefere Genomabdeckung und ermöglicht die Entdeckung der mikrobiellen „dunklen Materie“. Diese Sequenzen haben eine geringe Ähnlichkeit mit denen im öffentlichen Raum, was eine größere Handlungsfreiheit innerhalb dieses Sequenzabschnitts ermöglicht. Die Auswahl der 48 Enzyme, die in diesem Kit enthalten sind, wurde nicht nur durch die Möglichkeiten der Twist-DNA-Synthese, sondern auch durch eine gemeinsame bioinformatische Arbeit zur Verfeinerung des funktionalen Sequenzabschnitts unterstützt.
So wurden die Enzyme für unser Kit ausgewählt
Insgesamt wurden 369 aktive Transaminasen mit 19 Substraten identifiziert. 273 der Enzyme erwiesen sich in 10 % DMSO als aktiv mit 30 aktiv bei 60 °C. Schließlich wurden die Enzyme auf pH-Empfindlichkeit und Selektivität getestet.
Kriterien für das Transaminasen-Screening

Im Folgenden werden die Enzyme aufgeschlüsselt, die mit verschiedenen Substraten unter den zuvor beschriebenen unterschiedlichen Bedingungen (verschiedene DMSO-Anteile, organische Lösungsmittel, Temperaturen und pH-Werte) getestet wurden.

