Leistungssprung mit KI-Unterstützung
Bei der Twist AI Hypermutated scFv Library kommt künstliche Intelligenz zur Designoptimierung synthetischer Antikörperlibrarys mit vollhumanen Antikörpersequenzen zum Einsatz. Ein neurales Netzwerk bildet die Rekombination und Hypermutation von B-Zell-Rezeptoren nach und erzeugt auf diese Weise in der Entwicklung einsetzbare Antikörper.
Mit dieser einzigartigen Library können Sie die Identifizierung und Entwicklung therapeutischer Antikörper unabhängig von der Indikation weiter vorantreiben.
Leistungssprung mit KI-Unterstützung
Bei der Twist AI Hypermutated scFv Library kommt künstliche Intelligenz zur Designoptimierung synthetischer Antikörperlibrarys mit vollhumanen Antikörpersequenzen zum Einsatz. Ein neurales Netzwerk bildet die Rekombination und Hypermutation von B-Zell-Rezeptoren nach und erzeugt auf diese Weise in der Entwicklung einsetzbare Antikörper.
Mit dieser einzigartigen Library können Sie die Identifizierung und Entwicklung therapeutischer Antikörper unabhängig von der Indikation weiter vorantreiben.
Mit Deep Learning als Basis stellt die Twist AI Hypermutated scFv Library eine vielseitige Plattform für die Antikörperidentifizierung dar. Das für die Library verwendete neurale Netzwerk wurde mit Millionen Antikörpersequenzen gefüttert und erzeugte so diverse Antikörperrepertoires. Dieser Prozess ähnelt der natürlichen Hypermutation und Rekombination von B-Zell-Rezeptoren.
Die Library begrenzt die Diversität in der Complementary-Determining Region 1 (CDR1) und der Complementary-Determining Region 2 (CDR2) und erhöht zugleich die Diversität in CDR3. Die vier Schwerketten-Leichtketten-Kombinationen (VH3-23/VK1-39, VH3-23/VK3-20, VH1-69/ VK1-39 und VH1-69/VK3-20) umfassen jeweils 200 verknüpfte HCDR1-HCDR2-Sequenzen mit 100.000 HCDR3s sowie 100 verknüpfte LCDR1-LCDR2-Sequenzen mit 10.000 LCDR3s. Die kombinatorische Assemblierung ergibt eine vollhumane scFv-Library mit 400.000 HCDR3s, 40.000 LCDR3s sowie einer Diversität von 1 x 109.
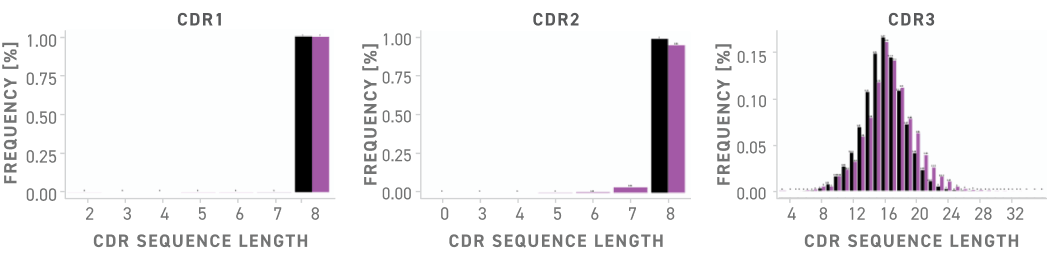
Die Sequenzen der AI Hypermutated scFv Library werden computergestützt abgeleitet. Hierzu wird auf einen sorgfältig ausgewählten Teildatensatz einer vollständigen Datenbank natürlich vorkommender Humanantikörper zugegriffen. Bei CDR1, CDR2 und CDR3 von IGHV3-23 entsprechen die Längenverteilungen der ausgewählten Antikörpersequenzen (violett) sehr genau jenen des Repertoires natürlicher Humanantikörper (schwarz).
Mit Deep Learning als Basis stellt die Twist AI Hypermutated scFv Library eine vielseitige Plattform für die Antikörperidentifizierung dar. Das für die Library verwendete neurale Netzwerk wurde mit Millionen Antikörpersequenzen gefüttert und erzeugte so diverse Antikörperrepertoires. Dieser Prozess ähnelt der natürlichen Hypermutation und Rekombination von B-Zell-Rezeptoren.
Die Library begrenzt die Diversität in der Complementary-Determining Region 1 (CDR1) und der Complementary-Determining Region 2 (CDR2) und erhöht zugleich die Diversität in CDR3. Die vier Schwerketten-Leichtketten-Kombinationen (VH3-23/VK1-39, VH3-23/VK3-20, VH1-69/ VK1-39 und VH1-69/VK3-20) umfassen jeweils 200 verknüpfte HCDR1-HCDR2-Sequenzen mit 100.000 HCDR3s sowie 100 verknüpfte LCDR1-LCDR2-Sequenzen mit 10.000 LCDR3s. Die kombinatorische Assemblierung ergibt eine vollhumane scFv-Library mit 400.000 HCDR3s, 40.000 LCDR3s sowie einer Diversität von 1 x 109.
Die Sequenzen der AI Hypermutated scFv Library werden computergestützt abgeleitet. Hierzu wird auf einen sorgfältig ausgewählten Teildatensatz einer vollständigen Datenbank natürlich vorkommender Humanantikörper zugegriffen. Bei CDR1, CDR2 und CDR3 von IGHV3-23 entsprechen die Längenverteilungen der ausgewählten Antikörpersequenzen (violett) sehr genau jenen des Repertoires natürlicher Humanantikörper (schwarz).

Vom Panning zum funktionellen Assay in 10–12 Wochen. Am Prozessbeginn steht ein Phagenscreening der diversen Twist AI Hypermutated scFv Library gegen die Zielantigene, am Prozessende schließlich die Reformatierung infrage kommender Antikörperfragmente in ein komplettes IgG.
Mit einer Lizenz der Twist AI Hypermutated scFv Library haben Sie auch die Möglichkeit, eigene Identifizierungsprojekte auf den Weg zu bringen. Weitere Informationen finden Sie unter biopharma@twistbioscience.com.
Vom Panning zum funktionellen Assay in 10–12 Wochen. Am Prozessbeginn steht ein Phagenscreening der diversen Twist AI Hypermutated scFv Library gegen die Zielantigene, am Prozessende schließlich die Reformatierung infrage kommender Antikörperfragmente in ein komplettes IgG.
Mit einer Lizenz der Twist AI Hypermutated scFv Library haben Sie auch die Möglichkeit, eigene Identifizierungsprojekte auf den Weg zu bringen. Weitere Informationen finden Sie unter biopharma@twistbioscience.com.
Proof-of-Concept-Daten
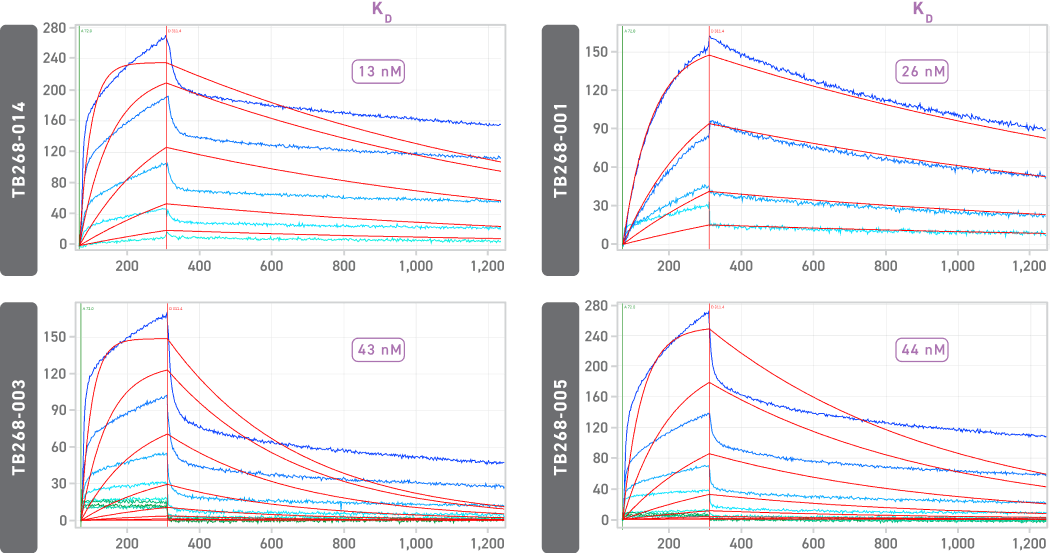
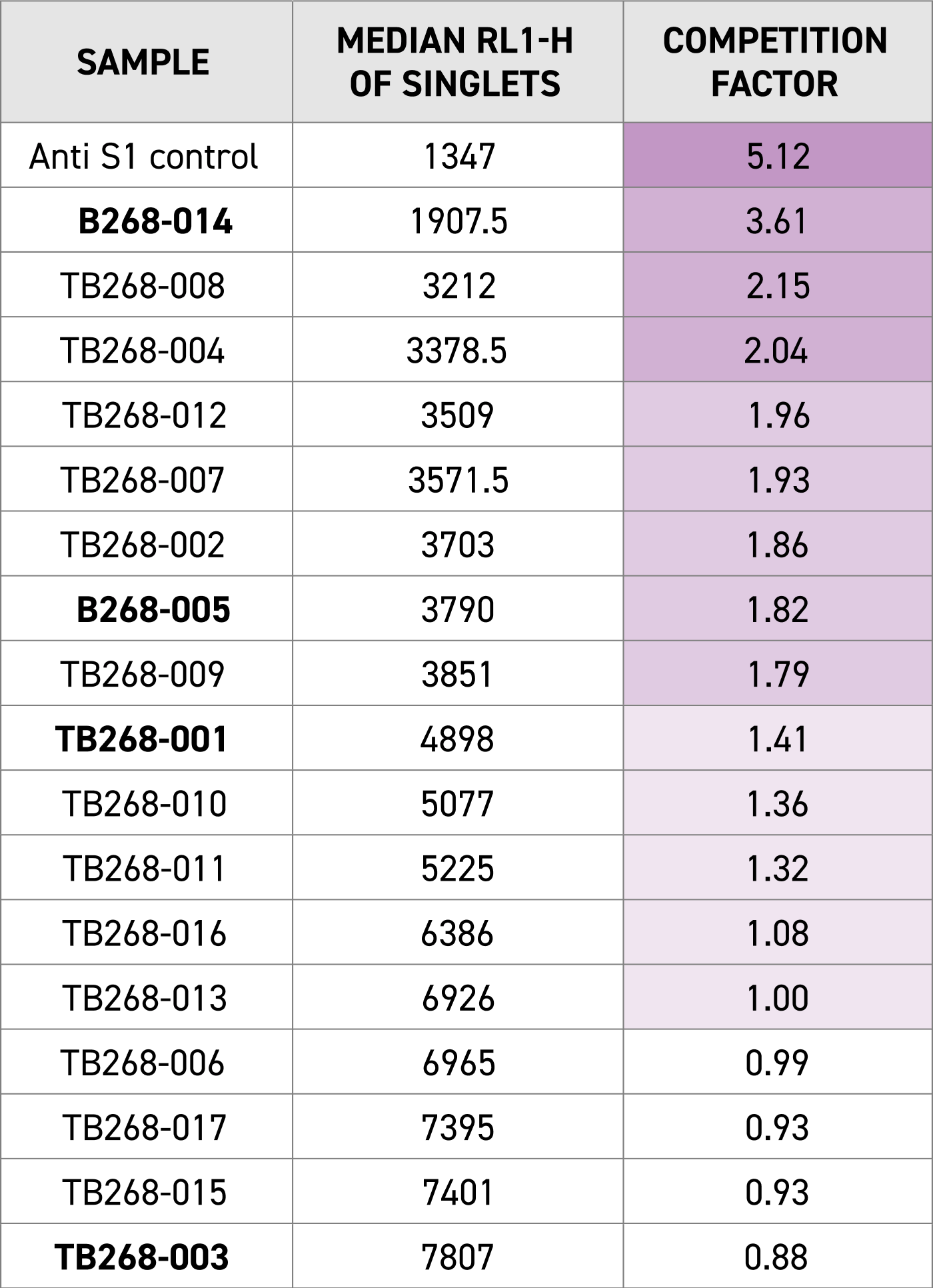
Die Twist AI Hypermutated scFv Library wurde erfolgreich einem Panning gegen das auf der Oberfläche des Coronavirus vorliegende Schlüsselprotein SARS-CoV-2-Spike-Protein S1 unterzogen, um einzigartige Klone mit wünschenswerten Eigenschaften zu identifizieren. Die Affinität wurde anhand der Oberflächenplasmonresonanz ermittelt und die Aktivität mittels kompetitiver Assays nachgewiesen.
Mit der AI Hypermutated scFv Library lassen sich eine hohe Bindungsaffinität aufweisende Kandidatenantikörper gegen das S1-Protein von SARS-CoV-2 effektiv ermitteln.
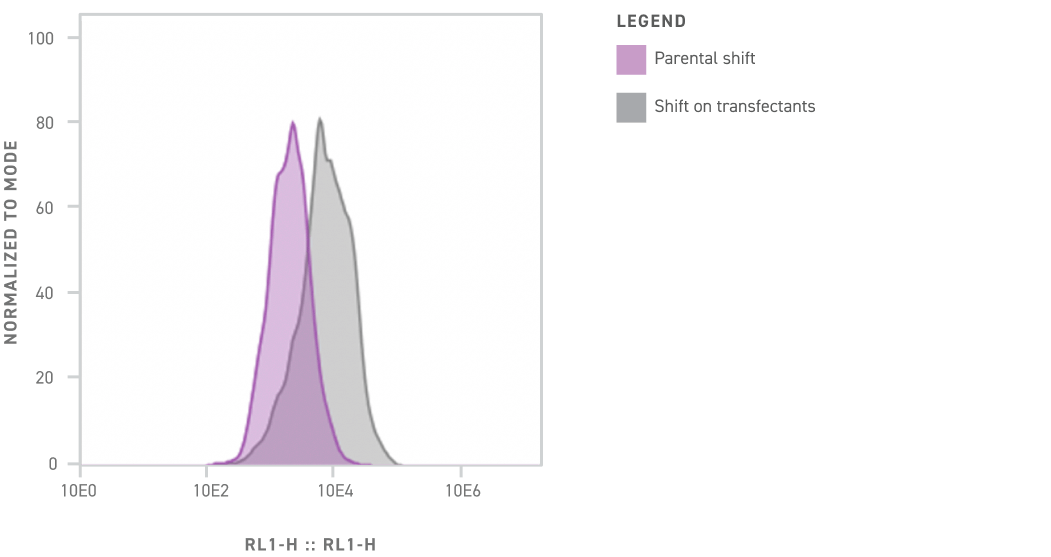
Mehrere Anti-S1-Antikörper der AI Hypermutated scFv Library hemmen nachweislich die S1-Bindung ACE2-exprimierender VERO-E6-Zellen. Das Diagramm zu den Ergebnissen der durchflusszytometrischen Analyse des repräsentativen Klons TB268-14 zeigt die Verschiebung der Transfektantenpopulation gegenüber der Elternpopulation.
Proof-of-Concept-Daten
Die Twist AI Hypermutated scFv Library wurde erfolgreich einem Panning gegen das auf der Oberfläche des Coronavirus vorliegende Schlüsselprotein SARS-CoV-2-Spike-Protein S1 unterzogen, um einzigartige Klone mit wünschenswerten Eigenschaften zu identifizieren. Die Affinität wurde anhand der Oberflächenplasmonresonanz ermittelt und die Aktivität mittels kompetitiver Assays nachgewiesen.
Mit der AI Hypermutated scFv Library lassen sich eine hohe Bindungsaffinität aufweisende Kandidatenantikörper gegen das S1-Protein von SARS-CoV-2 effektiv ermitteln.

Mehrere Anti-S1-Antikörper der AI Hypermutated scFv Library hemmen nachweislich die S1-Bindung ACE2-exprimierender VERO-E6-Zellen. Das Diagramm zu den Ergebnissen der durchflusszytometrischen Analyse des repräsentativen Klons TB268-14 zeigt die Verschiebung der Transfektantenpopulation gegenüber der Elternpopulation.


Webinar
Entwicklung von Therapeutika gegen praktisch jedes Ziel mit neuen Antikörper-Libraries
