Cas9-Alternativen ermöglichen CRISPR-Anwendungen der nächsten Generation
Verbesserung des Ernteertrags. Ausrottung von Malaria bei Mücken. Heilung bisher unheilbarer genetischer Erkrankungen. Sieben Jahre nach dem Debüt von CRISPR-Cas9 als Genschere ist die Frage: „Was kann Cas9 bewirken?“ schwer zu beantworten. Oder, was kann Cas9 nicht? Wird seine Nukleaseaktivität entfernt, wird das Cas9 unter anderem zu einer modularen Plattform für die Transkriptionskontrolle, epigenetische Modifikation, genomische Markierung und Basenmodifizierung. Der enorme biotechnologische Erfolg von Cas9 hat die Bemühungen zur Erweiterung der CRISPR-Toolbox um andere CRISPR-Nukleasen vorangetrieben und die nächste Generation von CRISPR-Anwendungen ermöglicht.
CRISPR-assoziierte (Cas) Enzyme existieren in den meisten Archaeen und vielen Bakterien als Bestandteil ihrer adaptiven Immunität. Im Jahr 2015 – nur ein paar Jahre nach der ersten Umnutzung von Cas9 als Genmodifikator – wurden Cas-Systeme anhand ihrer Unterscheidungsmerkmale in 2 Klassen, 5 Typen und 16 Subtypen unterteilt. Heute umfasst das Klassifizierungssystem 2 Klassen, 6 Typen und 33 Subtypen (Makarova et al., 2020). Diese Änderung ist teilweise auf die Suche nach neuen Klasse-2-Systemen zurückzuführen, die für das Genom-Engineering im Stil von Cas9 verwendet werden könnten. Dazu gehören der DNA-Editor Cas12a (Cpf1) und Cas13-basierte RNA-Editoren.
Argonautenproteine, eine Familie von Nukleinsäure-gesteuerten Proteinen, die sich größtenteils von CRISPR-Cas-Systemen unterscheiden, erhielten 2016 schlechte Presse, als ein hochkarätiger Artikel zurückgezogen wurde, nachdem mehrere unabhängige Gruppen die Behauptungen der sogenannten Genmodifizierungsfähigkeiten nicht replizieren konnten (Gao et al., 2016). Dennoch sollten Argonautenproteine hier erwähnt werden, wenn auch nur, um auf eine Anwendung hinzuweisen, für die sie besser geeignet sind als CRISPR-Systeme.
Im Folgenden werden einige der transformativsten Entwicklungen in der CRISPR- (und Argonautenprotein-)Technologie hervorgehoben, die von Experten von Twist Bioscience zusammengestellt wurden.
Cas12 vereinfacht kombinatorische Analysen
Viele biologische Prozesse wie die Tumormetastasierung beinhalten komplexe genetische Interaktionen, die mit einem Single-Knockout-Ansatz nicht vollständig gelöst werden können. Obwohl Cas9 der Goldstandard für Single-Knockout-Analysen ist, wurde seine Anwendung in kombinatorischen Analysen durch seine suboptimalen Multiplexing-Funktionen behindert. Mehrere unabhängige Gruppen haben kürzlich das CRISPR-System Cas12a (Cpf1) vom Typ V optimiert, um eine verbesserte Alternative zu Cas9 für die kombinatorische Genmodifizierung im großen Maßstab bereitzustellen (DeWeirdt et al., 2020; Gier et al., 2020).
Was macht Cas12a besser für kombinatorische Analysen als Cas9? Cas12a kann Arrays von CRISPR-RNA (crRNA) ohne RNase III und eine transaktivierende RNA (tracrRNA) verarbeiten – Komponenten, die Cas9 benötigt, um dieselbe Aufgabe zu erfüllen. Da Cas9 crRNA-Arrays nicht selbst verarbeiten kann, müssen Cas9-Guides normalerweise in separaten Expressionskassetten, eine pro Guide, exprimiert werden. Die repetitiven Elemente in Cas9-Multiplex-Guides machen diese anfällig für Rekombination und Entkopplung während der lentiviralen Transduktion, PCR und Tiefensequenzierung. Diese Faktoren können das Library-Design, die Klonierung und die Analyse erschweren.
Mit Cas12a können Oligonukleotide, die mehrere crRNAs enthalten, synthetisiert und für kombinatorische Analysen leicht in lentivirale Vektoren kloniert werden. Darüber hinaus sind die repetitiven Elemente in Cas12a-Multiplex-Guide-Vektoren vergleichsweise kurz und toleranter gegenüber Sequenzmodifikationen, wodurch Cas12a-Multiplex-Guides während der gesamten Analyse weniger anfällig für Rekombination und Entkopplung sind (DeWeirdt et al., 2020; Gier et al., 2020). Ein 300mer Oligonukleotid von Twist Bioscience kann 3 bis 4 Guides gleichzeitig codieren.
Während Cas12a ein ausgezeichneter Multiplexer ist, umfassen einige seiner Einschränkungen eine restriktivere PAM-Spezifität (5'-TTTV) und eine geringere Mutationseffizienz im Vergleich zu Cas9. Diese Parameter wurden durch strukturgesteuertes Protein Engineering von Cas12a aus Acidaminococcus (AsCas12a) verbessert (Kleinstiver et al., 2019). Erzielt wurde ein modifiziertes Enzym namens Enhanced AsCas12a (enAsCas12a), das Mehrpunktmutationen in für die PAM-Erkennung wichtigen Rückständen enthält. Zusätzlich zur Erweiterung der PAM-Spezifität (jetzt unter anderem mit TTYN, VTTV und TRTV) wurde zufällig festgestellt, dass enAsCas12a die Modifizierungseffizienz im Vergleich zu AsCas12a im Durchschnitt um das Doppelte verbessert. Die Effizienz von AsCas12a wurde weiter verbessert, indem mehrere Kernlokalisierungssequenzen hinzugefügt und die crRNA-Regeln innerhalb und außerhalb des Ziels geklärt wurden (DeWeirdt et al., 2020; Gier et al., 2020).
CRISPR-Systeme vom Typ VI haben RNA im Visier
Die RNA-Interferenz (RNAi) unter Verwendung von Small hairpin RNA (shRNA) ist eine etablierte Gen-Knockdown-Strategie, bei der ein endogener RNA-Verarbeitungsweg zum Abbau zellulärer mRNA kooptiert wird. RNAi leidet jedoch unter Effekten außerhalb des Ziels und einer inkonsistenten Knockdown-Effizienz. Das Spezifitätsproblem wurde mit CRISPR-Interferenz gelöst, einem Knockdown-Ansatz, bei dem eine inaktive Cas9-Nuklease verwendet wird, die an die Transkriptionsrepressor-Domäne KRAB fusioniert ist. Trotzdem können sowohl CRISPR-Interferenz als auch RNAi gelegentlich ihre Ziele bei hoher basaler Genexpression bzw. schlechter shRNA-hairpin-Verarbeitung nicht robust blockieren.
Betrachten Sie RfxCas13d (CasRx), eine RNA-gesteuerte, RNA-zielgerichtete CRISPR-Nuklease. CasRx war möglicherweise nicht der erste entdeckte RNA-Modifikator, aber Seite-an-Seite-Vergleiche mit anderen Cas13-Systemen (LwaCas13a und PspCas13b) und vorhandenen Methoden zeigen, dass CasRx über viele Gene hinweg den größten Knockdown induziert – und dies mit hoher Spezifität. Darüber hinaus kann eine inaktive Variante der dCasRx-Nuklease auf cis-Elemente von prä-mRNA abzielen, um deren Spleißen zu regulieren. Um dieses Merkmal hervorzuheben, zielten Konermann et al. (2018) dCasRx auf einen intronischen Spleißschalldämpfer ab, um das pathologische Spleißen von Tau-codierender mRNA in einem neuronalen Modell der Frontotemporalen Demenz zu korrigieren.
Aufgrund ihrer Präferenz für einzelsträngige RNA kann das Targeting von Cas13-Systemen im Allgemeinen durch die RNA-Sekundärstruktur behindert werden (Abudayyeh et al., 2017). Ihre Fähigkeit, vorübergehende Veränderungen durch gezieltes Targeting von RNA über das Genom zu verursachen, und ihr Mangel an Sequenzbeschränkungen machen sie jedoch zu attraktiven Transkriptom-Modifizierungswerkzeugen für experimentelle und therapeutische Anwendungen.
CRISPR-Cas-Systeme der Klasse 1 diversifizieren das CRISPR-Toolkit
Da CRISPR-Cas-Systeme der Klasse 2 wie Cas9, Cas12s und Cas13s weiterhin den Bereich der Genomtechnik dominieren, gewinnen CRISPR-Cas-Systeme der Klasse 1 aufgrund ihrer einzigartigen Spaltungseigenschaften und ihrer Multiproteinkomplexe zunehmend an Aufmerksamkeit. Am häufigsten sind Typ-I-Systeme, die die Cas3-Helikase mit einem Komplex von akzessorischen Cas-Proteinen kombinieren, der als CRISPR-assoziierter Komplex zur Virenabwehr oder „Cascade“ bezeichnet wird.
Wenn Cas9 und Cas12a Genscheren sind, dann ist Cas3 ein Gen-Schredder. Anstatt kleine Insertions-/Deletionsmutanten wie Cas9 zu produzieren, verursacht Cas3 ein Spektrum großer genomischer Deletionen mit einer Länge von 0,5 bis 100 kb, die Zielregionen des Genoms vorgelagert sind (Dolan et al., 2019). Dieses Merkmal verspricht neuartige Hochdurchsatz-Analysen, bei denen Libraries von Deletionsmutanten mit vergleichsweise weniger Guides als CRISPR-Systeme der Klasse 2 erzeugt werden können.
The Cascade complex alone also provides a modular platform for RNA-guided transcriptional activation and repression. Site-directed repression can be achieved using crRNAs in cas3-deleted E. coli (Luo et al., 2015; Rath et al., 2015) or in mammalian cells expressing exogenous Cascade proteins (Pickar-Oliver et al., 2019; Chen et al., 2020).
For gene activation, transcriptional activator domains need to be fused to one or more members of Cascade. Chen et al. (2020) obtained the best results when the synthetic VPR (VP64-p65-Rta) activator domain was fused to Csy3, the backbone component of Pseudomonas aeruginosa Cascade (PaeCascade) that interacts with the crRNA. Because multiple Csy3 proteins participate in the formation of the PaeCascade complex, multiple transcriptional effector domains can be recruited to a single Cascade complex. This strategy does not necessarily work for other class 1 systems, however, as the fusion of transcriptional effectors to Cas7 of EcoCascade (its Csy3 equivalent) failed to elevate transcription at targeted loci (Pickar-Oliver et al., 2019).
Das Multiprotein-Wesen von Cascade ist praktisch für den Aufbau modularer Geräte zur Transkriptionskontrolle, bedeutet aber auch, dass mehrere Effektorproteine coexprimiert werden müssen. Dies könnte die Verwendung von Klasse-1-Systemen für einige Anwendungen behindern.
Seltsame CRISPR-Loci enthüllen CRISPR-assoziierte Transposasen
Most CRISPR loci contain genes for site-directed nucleases. For example, CRISPR loci encoding type I systems typically contain cas3, which encodes the endonuclease effector of these Cas systems. Recent bioinformatic analyses, however, revealed CRISPR loci containing Tn7-like transposon genes instead of Cas endonuclease genes. From these loci, two CRISPR-associated transposon systems have been characterized: the class 2 type V-K effector Cas12k (Strecker et al., 2019) and the class 1 type I-F Cascade complex (Halpin-Healy et al., 2020). Instead of cleaving DNA, these Cas systems can insert large segments of DNA into the genome in a site-directed and nuclease-independent fashion.
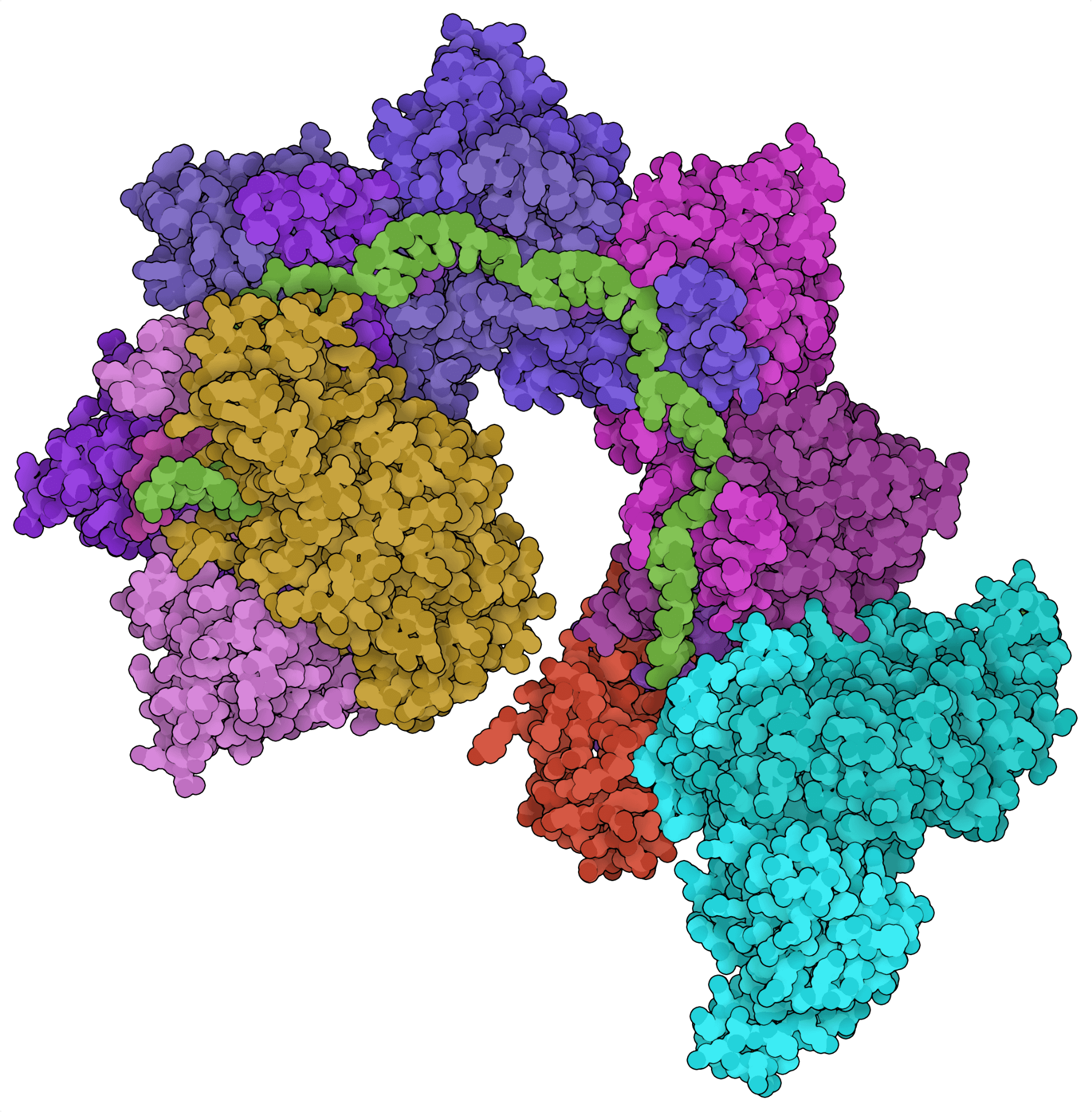
Herkömmliche CRISPR-basierte Gen-Knock-in-Ansätze sind abhängig von einer homologiegesteuerten Reparatur nach einem zielgerichteten DNA-Bruch – ein sehr ineffizienter Prozess. Die neu identifizierten CRISPR-assoziierten Transposons bieten eine potenziell effizientere Problemumgehung, indem sie die Transposition großer DNA-Fragmente ermöglichen. CRISPR-assoziierte Transposasen könnten verwendet werden, um fehlerhafte Exons zu ersetzen oder chimäre Antigenrezeptor-(CAR-)T-Zellen zu erzeugen. Dies klingt vielversprechend, jedoch wurden die Transposaseaktivitäten dieser Systeme in eukaryotischen Zellen noch nicht nachgewiesen.
Argonauten ermöglichen DNA-Datenspeicherung
Sometimes the best alternative to Cas9 is not a CRISPR system at all. This is true for nick-based DNA data storage, which requires a nicking enzyme to encode information in native DNA. DNA has been proposed as an alternative to current silicon-based storage media because of its relatively high durability and enormous storage density. To further push the limits of the information storage density of DNA, researchers have recently developed a nick-based approach (Tabatabaei et al., 2020).
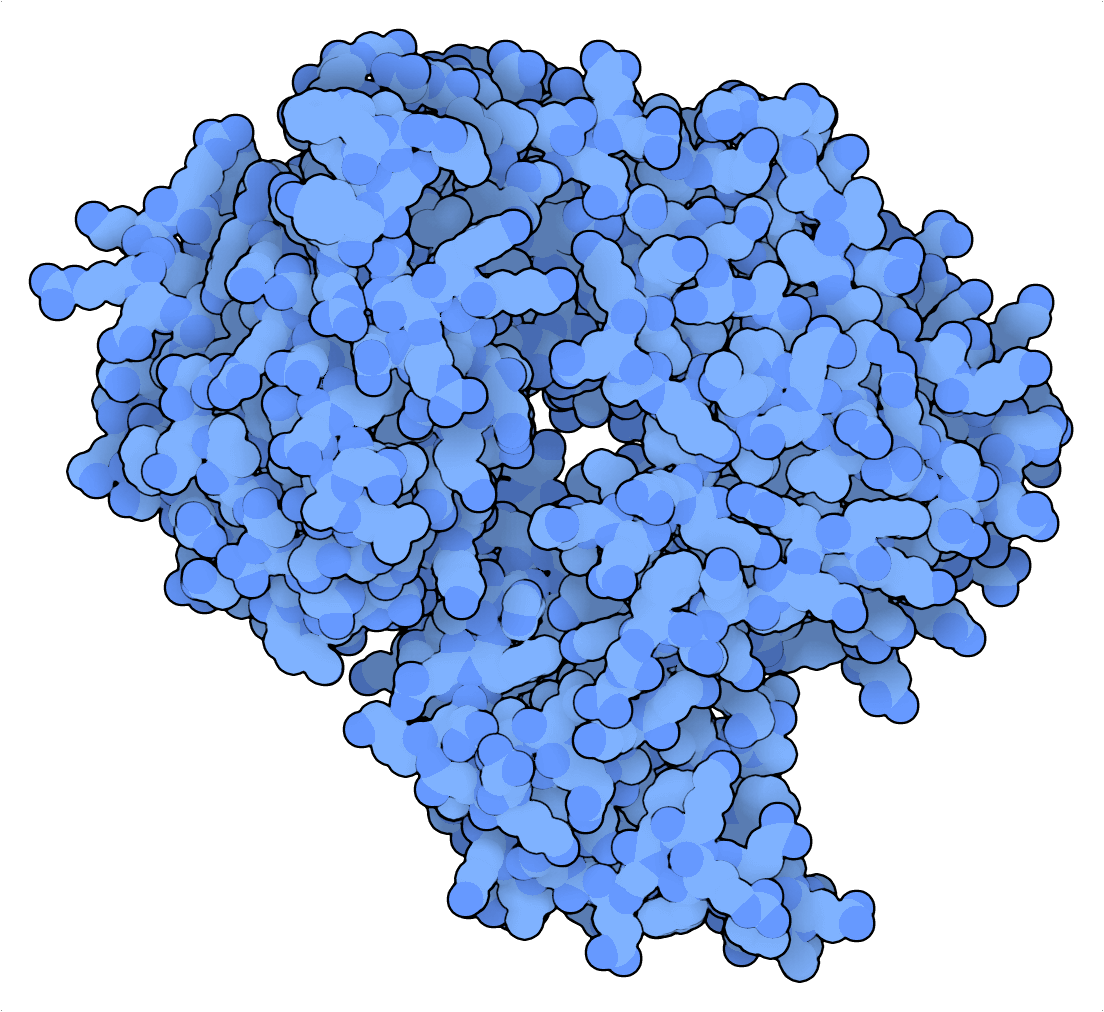
Tabatabaei et al. (2020) nutzten die günstigen Nick-Eigenschaften von PfArgo, um ein DNA-Schreibgerät zu entwickeln, das einen dsDNA-Strang in eine „Lochkarte“ verwandelt. Hier werden binäre Informationen in einem Muster von Kerben (Nicks) positionell codiert, die durch Denaturieren der DNA-Einzelstrangfragmente gelesen werden können. Diese Fragmente können dann durch NGS oder Nanopore-Sequenzierung sequenziert und auf die Referenz-Lochkarten abgebildet werden, um die codierten Informationen abzurufen. Tabatabaei et al. (2020) komprimierten eine 329-KB-JPEG-Datei des Lincoln Memorial in eine 14-KB-DNA-Datei, was die hervorragende Speicherdichte dieser Strategie hervorhebt!
Bei der Nick-basierten DNA-Datenspeicherung verblassen die Kerbeneigenschaften der Cas9-Nickase im Vergleich zu denen von Pyrococcus furiosus Argonaute (PfAgo). Während Cas9-Nickase ein Enzym mit einer Umsatzaktivität von 1 ist, das auf instabilen und PAM-beschränkten RNA-DNA-Wechselwirkungen beruht, kann PfAgo Hunderte von Kerben pro Enzym mithilfe kurzer DNA-Guides induzieren, die nicht durch eine bestimmte Zielsequenz begrenzt sind.
Dies sind nur einige der Anwendungen, die durch nukleinsäuregesteuerte Proteine in den Familien CRISPR und Argonaute ermöglicht werden. Um mehr darüber zu erfahren, wie Sie Ihre CRISPR-Toolbox erweitern können, sprechen Sie mit einem Teammitglied.
Protein illustrations were generated from PDB structures using Illustrate, a non-photorealistic biomolecular illustration tool (Goodsell et al., 2019).
Literatur
Abudayyeh OO, Gootenberg JS, Essletzbichler P, Han S, Joung J, Belanto JJ, Verdine V, Cox DBT, Kellner MJ, Regev A, Lander ES, Voytas DF, Ting AY, Zhang F (2017) RNA targeting with CRISPR-Cas13. Nature 550:280-284.
Chen Y, Liu J, Zhi S, Zheng Q, Ma W, Huang J, Liu Y, Liu D, Liang P, Songyang Z (2020) Repurposing type I-F CRISPR-Cas system as a transcriptional activation tool in human cells. Nat Commun 11:3136.
DeWeirdt, PC, Sangree, AK, Hanna, RE et al. (2020) Genetic screens in isogenic mammalian cell lines without single cell cloning. Nat Commun 11, 752.
Dolan AE, Hou Z, Xiao Y, Gramelspacher MJ, Heo J, Howden SE, Freddolino PL, Ke A, Zhang Y (2019) Introducing a Spectrum of Long-Range Genomic Deletions in Human Embryonic Stem Cells Using Type I CRISPR-Cas. Mol Cell 74:936-950 e935.
Gao F, Shen XZ, Jiang F, Wu Y, Han C (2016) DNA-guided genome editing using the Natronobacterium gregoryi Argonaute. Nat Biotechnol 34:768-773.
Gier, RA, Budinich, KA, Evitt, NH et al. (2020) High-performance CRISPR-Cas12a genome editing for combinatorial genetic screening. Nat Commun 11, 3455
Goodsell, DS, Autin, L, Olson, AJ (2019) Illustrate: Software for Biomolecular Illustration. Structure 27:1716-1720.e1
Halpin-Healy TS, Klompe SE, Sternberg SH, Fernandez IS (2020) Structural basis of DNA targeting by a transposon-encoded CRISPR-Cas system. Nature 577:271-274.
Kleinstiver BP, Sousa AA, Walton RT, Tak YE, Hsu JY, Clement K, Welch MM, Horng JE, Malagon-Lopez J, Scarfo I, Maus MV, Pinello L, Aryee MJ, Joung JK (2019) Engineered CRISPR-Cas12a variants with increased activities and improved targeting ranges for gene, epigenetic and base editing. Nat Biotechnol 37:276-282.
Konermann S, Lotfy P, Brideau NJ, Oki J, Shokhirev MN, Hsu PD (2018) Transcriptome Engineering with RNA-Targeting Type VI-D CRISPR Effectors. Cell 173:665-676 e614.
Luo ML, Mullis AS, Leenay RT, Beisel CL (2015) Repurposing endogenous type I CRISPR-Cas systems for programmable gene repression. Nucleic Acids Res 43:674-681.
Makarova KS et al. (2020) Evolutionary classification of CRISPR-Cas systems: a burst of class 2 and derived variants. Nat Rev Microbiol 18:67-83.
Pickar-Oliver A, Black JB, Lewis MM, Mutchnick KJ, Klann TS, Gilcrest KA, Sitton MJ, Nelson CE, Barrera A, Bartelt LC, Reddy TE, Beisel CL, Barrangou R, Gersbach CA (2019) Targeted transcriptional modulation with type I CRISPR-Cas systems in human cells. Nat Biotechnol 37:1493-1501.
Rath D, Amlinger L, Hoekzema M, Devulapally PR, Lundgren M (2015) Efficient programmable gene silencing by Cascade. Nucleic Acids Res 43:237-246.
Strecker J, Ladha A, Gardner Z, Schmid-Burgk JL, Makarova KS, Koonin EV, Zhang F (2019) RNA-guided DNA insertion with CRISPR-associated transposases. Science 365:48-53.
Tabatabaei SK, Wang B, Athreya NBM, Enghiad B, Hernandez AG, Fields CJ, Leburton JP, Soloveichik D, Zhao H, Milenkovic O (2020) DNA punch cards for storing data on native DNA sequences via enzymatic nicking. Nat Commun 11:1742.
Was denken Sie?
Gefällt mir
Gefällt mir nicht
Gefällt mir sehr
Überraschend
Interessant
