Détection de la méthylation améliorée pour les flux de travail de biopsie liquide
Les technologies de biopsie liquide transforment rapidement le paysage de la détection et de la surveillance précoces du cancer. En offrant une approche aussi peu invasive que possible, les biopsies liquides permettent d’analyser des biomarqueurs circulants tels que l’ADNcf (ADN acellulaire), qui apportent des informations précieuses sur les altérations génétiques et épigénétiques associées au cancer. Parmi ces biomarqueurs, les changements de méthylation de l’ADN se distinguent comme une solution prometteuse pour l’identification et le suivi de divers cancers avec une spécificité et une sensibilité élevées.
En dépit de ce potentiel, le travail avec l’ADNcf présente des défis uniques en raison de son abondance limitée et de ses modèles de fragmentation distincts. La plupart des fragments d’ADNcf ont une longueur d’environ 167 pb, ce qui reflète leur origine liée aux nucléosomes. Cette fragmentation se traduit par :
- Une faible diversité stop-start, ce qui permet d’obtenir davantage de molécules uniques appelées lectures de séquençage en double
- Une moindre complexité de la banque, ce qui peut freiner la sensibilité de l’analyse
Ces propriétés inhérentes rendent les flux de travail traditionnels de détection de la méthylation moins efficaces pour l’analyse de l’ADNcf ; des outils et des techniques avancés adaptés sont donc nécessaires pour répondre à ces limitations.
Les adaptateurs UMI méthylés Twist améliorent les flux de travail de méthylation de l’ADNcf, en permettant une déduplication précise grâce à des identifiants moléculaires uniques (UMI), ce qui réduit les identifications de fausses duplications dans les échantillons à faible diversité. Leur conception garantit la compatibilité avec les protocoles EM-Seq, en préservant la longueur des fragments, en maximisant les données utilisables et en améliorant la couverture et la reproductibilité des cibles, ce qui en fait un outil essentiel pour assurer des études de méthylation avec un niveau de confiance élevé.
Détection de la méthylation améliorée pour les flux de travail de biopsie liquide
Les technologies de biopsie liquide transforment rapidement le paysage de la détection et de la surveillance précoces du cancer. En offrant une approche aussi peu invasive que possible, les biopsies liquides permettent d’analyser des biomarqueurs circulants tels que l’ADNcf (ADN acellulaire), qui apportent des informations précieuses sur les altérations génétiques et épigénétiques associées au cancer. Parmi ces biomarqueurs, les changements de méthylation de l’ADN se distinguent comme une solution prometteuse pour l’identification et le suivi de divers cancers avec une spécificité et une sensibilité élevées.
En dépit de ce potentiel, le travail avec l’ADNcf présente des défis uniques en raison de son abondance limitée et de ses modèles de fragmentation distincts. La plupart des fragments d’ADNcf ont une longueur d’environ 167 pb, ce qui reflète leur origine liée aux nucléosomes. Cette fragmentation se traduit par :
- Une faible diversité stop-start, ce qui permet d’obtenir davantage de molécules uniques appelées lectures de séquençage en double
- Une moindre complexité de la banque, ce qui peut freiner la sensibilité de l’analyse
Ces propriétés inhérentes rendent les flux de travail traditionnels de détection de la méthylation moins efficaces pour l’analyse de l’ADNcf ; des outils et des techniques avancés adaptés sont donc nécessaires pour répondre à ces limitations.
Les adaptateurs UMI méthylés Twist améliorent les flux de travail de méthylation de l’ADNcf, en permettant une déduplication précise grâce à des identifiants moléculaires uniques (UMI), ce qui réduit les identifications de fausses duplications dans les échantillons à faible diversité. Leur conception garantit la compatibilité avec les protocoles EM-Seq, en préservant la longueur des fragments, en maximisant les données utilisables et en améliorant la couverture et la reproductibilité des cibles, ce qui en fait un outil essentiel pour assurer des études de méthylation avec un niveau de confiance élevé.
Intégration des UMI dans l’analyse pour une meilleure résolution des doublons
L’identification éclairée des doublons par UMI réduit les doublons dans la banque de plus de 15 %, permettant une augmentation de la complexité de la banque (25 à 35 %) et de la couverture cible moyenne (6 à 8 %) par rapport à l’identification de doublons standard basée sur la position. (Figure 1)
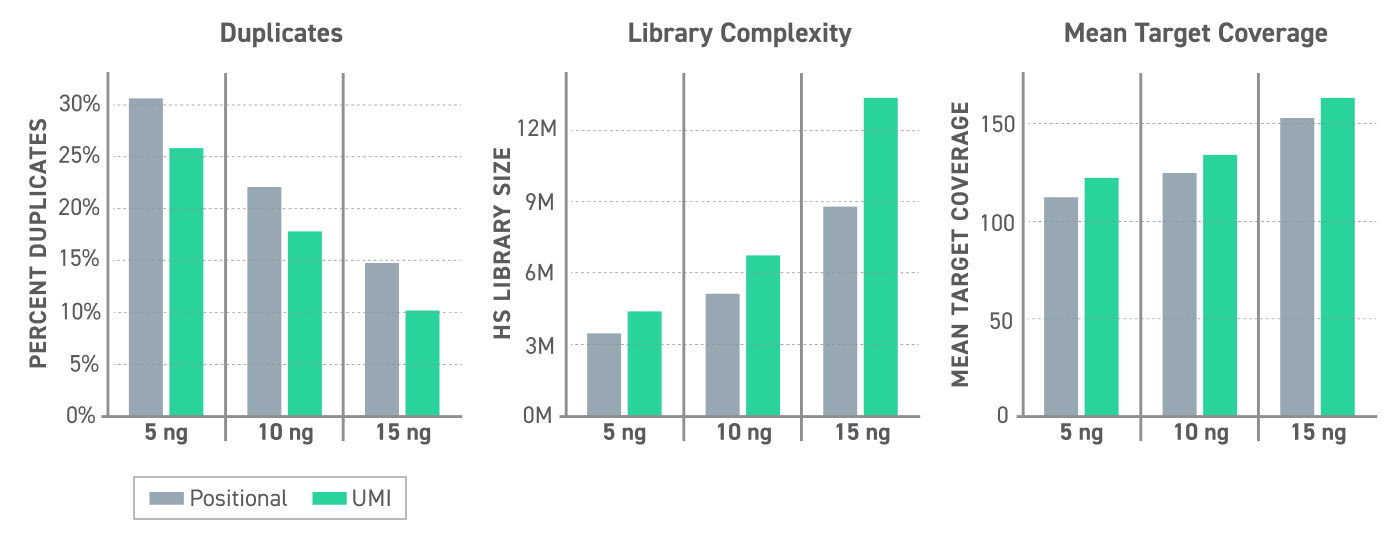
Figure 1. Performances des UMI avec différentes entrées de masse d’ADNcf 5, 10 ou 15 ng d’ADNcf ont été utilisés pour générer des banques converties en EM-seq avec des adaptateurs UMI méthylés Twist après la préparation de banque NEBNext EM-Seq. Les banques ont été capturées avec le Panel de méthylation pan-cancéreux Twist Alliance - 1,5 Mb à l’aide du protocole d’analyse de méthylation par séquençage ciblée, et séquencées sur le système Illumina Nextseq 550 avec une couverture brute de 1000X. Les doublons ont été identifiés à l’aide de l’outil GATK MarkDuplicates (méthode positionnelle standard) ou à l’aide des UMI avec l’outil GATK UmiAwareMarkDuplicatesWithMateCigar.
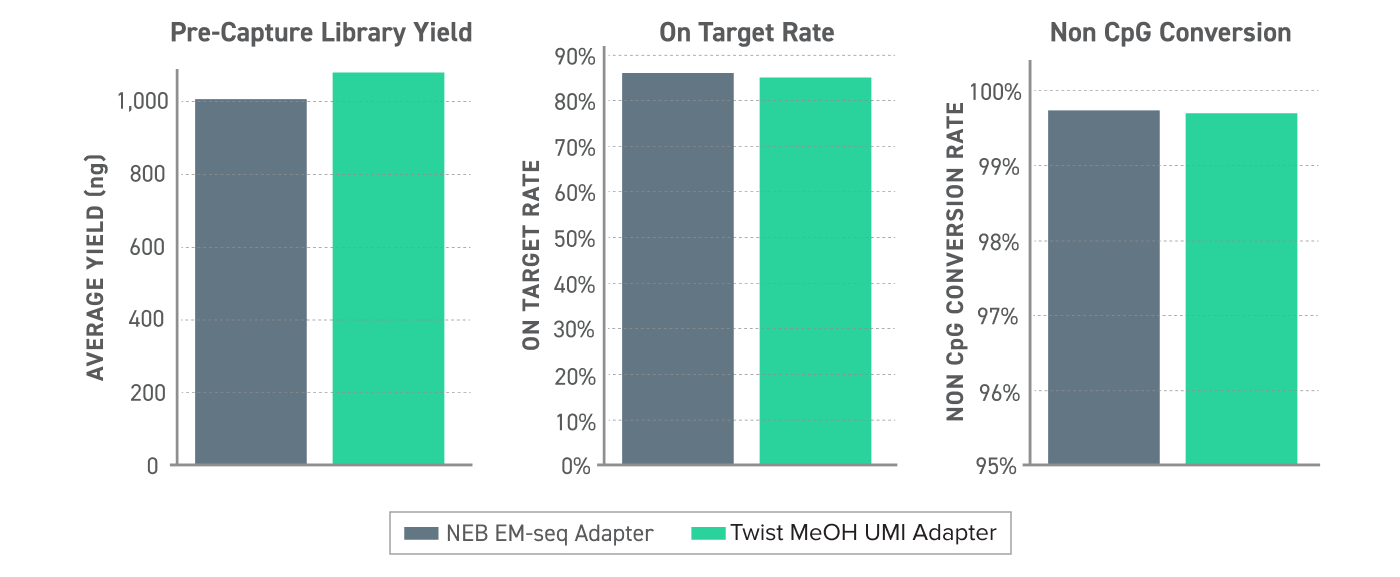
Figure 2. Performances de l’UMI méthylé dans le flux de travail de détection de méthylation Twist 10 ng d’ADNcf humain ont été utilisés pour générer des banques converties en EM-seq avec les adaptateurs NEB EMseq ou les nouveaux adaptateurs UMI méthylés Twist. Les banques ont été capturées à l’aide du panel de méthylation pan-cancéreux Twist Alliance -1,5 Mb, en utilisant le protocole d’analyse de méthylation par séquençage ciblée, et séquencées sur le système Illumina Nextseq 550 avec une couverture brute de 1000X.
Exemple de flux de travail bioinformatique
Les informations UMI peuvent être intégrées dans un pipeline bioinformatique pour éliminer les doublons avant l’analyse en aval.

Figure 3. Résumé du flux de travail d’analyse visant à traiter les UMI pour marquer les doublons
Les lectures brutes sont traitées pour marquer les séquences d’adaptateur et extraire les informations UMI. Les lectures sont ensuite alignées sur un génome de référence à l’aide de BWA-meth et marquées pour les doublons à l’aide d’informations UMI. Après le marquage des doublons, les mesures Picard peuvent être collectées à l’aide de GATK et une analyse complémentaire peut être effectuée.
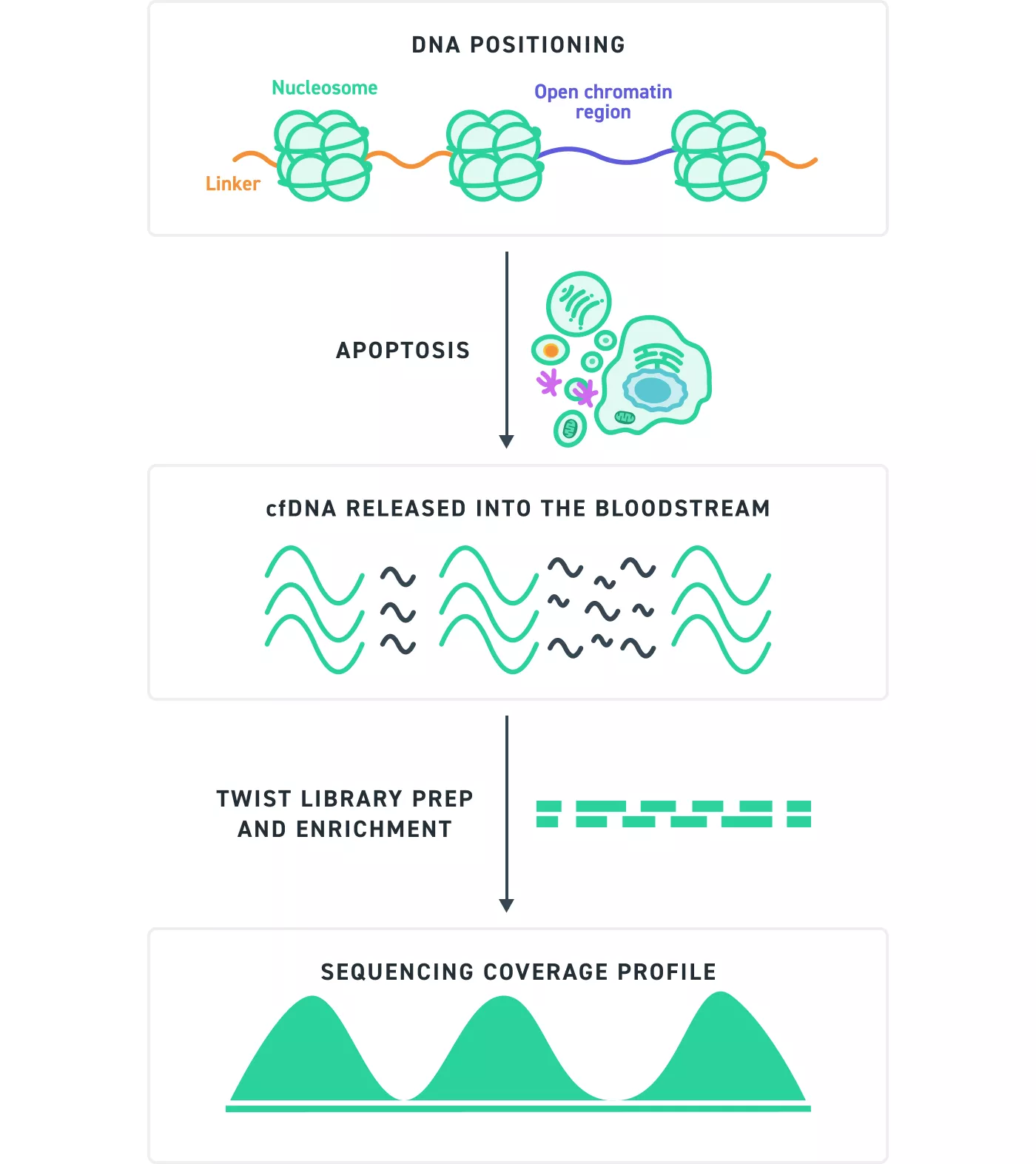
Figure 4. Schéma de la fragmentation apoptotique de l’ADNlc jusqu’à la couverture du séquençage :
Lors de l’apoptose, l’ADN libre circulant (ADNlc) est libéré dans la circulation sanguine. Alors que les endonucléases fragmentent l’ADN de manière aléatoire, la manière dont l’ADN s’enroule autour des nucléosomes introduit un biais de position, qui limite la diversité des sites de démarrage et d’arrêt des fragments. Par conséquent, des molécules distinctes peuvent apparaître comme des doublons dans les données de séquençage. Des identifiants moléculaires uniques (Unique Molecular Identifier, UMI) sont ajoutés lors de la préparation de banque pour étiqueter les molécules individuelles, permettant une différenciation précise entre les vrais doublons et les molécules uniques distinctes.
Intégration des UMI dans l’analyse pour une meilleure résolution des doublons
L’identification éclairée des doublons par UMI réduit les doublons dans la banque de plus de 15 %, permettant une augmentation de la complexité de la banque (25 à 35 %) et de la couverture cible moyenne (6 à 8 %) par rapport à l’identification de doublons standard basée sur la position. (Figure 1)

Figure 1. Performances des UMI avec différentes entrées de masse d’ADNcf 5, 10 ou 15 ng d’ADNcf ont été utilisés pour générer des banques converties en EM-seq avec des adaptateurs UMI méthylés Twist après la préparation de banque NEBNext EM-Seq. Les banques ont été capturées avec le Panel de méthylation pan-cancéreux Twist Alliance - 1,5 Mb à l’aide du protocole d’analyse de méthylation par séquençage ciblée, et séquencées sur le système Illumina Nextseq 550 avec une couverture brute de 1000X. Les doublons ont été identifiés à l’aide de l’outil GATK MarkDuplicates (méthode positionnelle standard) ou à l’aide des UMI avec l’outil GATK UmiAwareMarkDuplicatesWithMateCigar.

Figure 2. Performances de l’UMI méthylé dans le flux de travail de détection de méthylation Twist 10 ng d’ADNcf humain ont été utilisés pour générer des banques converties en EM-seq avec les adaptateurs NEB EMseq ou les nouveaux adaptateurs UMI méthylés Twist. Les banques ont été capturées à l’aide du panel de méthylation pan-cancéreux Twist Alliance -1,5 Mb, en utilisant le protocole d’analyse de méthylation par séquençage ciblée, et séquencées sur le système Illumina Nextseq 550 avec une couverture brute de 1000X.
Exemple de flux de travail bioinformatique
Les informations UMI peuvent être intégrées dans un pipeline bioinformatique pour éliminer les doublons avant l’analyse en aval.

Figure 3. Résumé du flux de travail d’analyse visant à traiter les UMI pour marquer les doublons
Les lectures brutes sont traitées pour marquer les séquences d’adaptateur et extraire les informations UMI. Les lectures sont ensuite alignées sur un génome de référence à l’aide de BWA-meth et marquées pour les doublons à l’aide d’informations UMI. Après le marquage des doublons, les mesures Picard peuvent être collectées à l’aide de GATK et une analyse complémentaire peut être effectuée.
Figure 4. Schéma de la fragmentation apoptotique de l’ADNlc jusqu’à la couverture du séquençage :
Lors de l’apoptose, l’ADN libre circulant (ADNlc) est libéré dans la circulation sanguine. Alors que les endonucléases fragmentent l’ADN de manière aléatoire, la manière dont l’ADN s’enroule autour des nucléosomes introduit un biais de position, qui limite la diversité des sites de démarrage et d’arrêt des fragments. Par conséquent, des molécules distinctes peuvent apparaître comme des doublons dans les données de séquençage. Des identifiants moléculaires uniques (Unique Molecular Identifier, UMI) sont ajoutés lors de la préparation de banque pour étiqueter les molécules individuelles, permettant une différenciation précise entre les vrais doublons et les molécules uniques distinctes.

110830
Produit : Adaptateurs UMI méthylés Twist – Compatibles avec TruSeq, 96 échantillon110830
Produit : Adaptateurs UMI méthylés Twist – Compatibles avec TruSeq, 96 échantillon