Accédez à des régions de séquence enzymatiques inexploitées pour permettre une exploration optimale
Nos nouvelles séquences enzymatiques ont été identifiées à partir de divers génomes unicellulaires microbiens non cultivés et ont été sélectionnées pour leurs propriétés intéressantes à l’aide de modèles assistés par apprentissage automatique en fonction de la prédiction de la structure, du clustering basé sur la structure et du filtrage - vous ne trouverez probablement ces séquences nulle part ailleurs.
Les transaminases (TA) sont des enzymes qui catalysent le transfert d’un groupe aminé d’un donneur d’amines à une cétone ou à un aldéhyde. Cette capacité à faciliter l’amination stéréosélective des cétones prochirales est précieuse dans la fabrication pharmaceutique, où la demande d’amines chirales comme éléments constitutifs d’ingrédients pharmaceutiques actifs (API) est élevée.
Nous avons identifié et réduit un sous-ensemble d’enzymes TA qui maintiennent leur activité dans une gamme de conditions extrêmes sur de nombreux substrats divers, utiles pour le processus de fabrication pharmaceutique.
Notre kit de criblage permet de sélectionner des séquences pertinentes sur le plan fonctionnel, qui pourront à l’avenir être combinées avec des ressources d’ingénierie enzymatique en aval afin de fournir une voie plus efficace pour l’ingénierie des protéines.
Comment les enzymes sont sélectionnées pour le criblage
La base de données bitBiome bit-GEM est la principale ressource de séquences génomiques de microbes consultables et hautement utilisables. Exploitant les ressources microbiennes les plus puissantes de la nature, notamment des échantillons provenant d’environnements tels que le sol, les sources thermales, l’eau de mer, les sites miniers et d’autres conditions extrêmes, la base de données bit-GEM contient un vaste éventail de séquences de gènes acquises par la plateforme de séquençage unicellulaire bitBiome. Cette méthode de séquençage assure une couverture plus profonde du génome, ce qui permet de découvrir la « matière noire » microbienne. Ces séquences ont peu de similitudes avec celles du domaine public, ce qui donne au client la liberté d’opérer dans ces régions de séquence.



Accédez à des régions de séquence enzymatiques inexploitées pour permettre une exploration optimale
Nos nouvelles séquences enzymatiques ont été identifiées à partir de divers génomes unicellulaires microbiens non cultivés et ont été sélectionnées pour leurs propriétés intéressantes à l’aide de modèles assistés par apprentissage automatique en fonction de la prédiction de la structure, du clustering basé sur la structure et du filtrage - vous ne trouverez probablement ces séquences nulle part ailleurs.
Les transaminases (TA) sont des enzymes qui catalysent le transfert d’un groupe aminé d’un donneur d’amines à une cétone ou à un aldéhyde. Cette capacité à faciliter l’amination stéréosélective des cétones prochirales est précieuse dans la fabrication pharmaceutique, où la demande d’amines chirales comme éléments constitutifs d’ingrédients pharmaceutiques actifs (API) est élevée.
Nous avons identifié et réduit un sous-ensemble d’enzymes TA qui maintiennent leur activité dans une gamme de conditions extrêmes sur de nombreux substrats divers, utiles pour le processus de fabrication pharmaceutique.
Notre kit de criblage permet de sélectionner des séquences pertinentes sur le plan fonctionnel, qui pourront à l’avenir être combinées avec des ressources d’ingénierie enzymatique en aval afin de fournir une voie plus efficace pour l’ingénierie des protéines.
Comment les enzymes sont sélectionnées pour le criblage
La base de données bitBiome bit-GEM est la principale ressource de séquences génomiques de microbes consultables et hautement utilisables. Exploitant les ressources microbiennes les plus puissantes de la nature, notamment des échantillons provenant d’environnements tels que le sol, les sources thermales, l’eau de mer, les sites miniers et d’autres conditions extrêmes, la base de données bit-GEM contient un vaste éventail de séquences de gènes acquises par la plateforme de séquençage unicellulaire bitBiome. Cette méthode de séquençage assure une couverture plus profonde du génome, ce qui permet de découvrir la « matière noire » microbienne. Ces séquences ont peu de similitudes avec celles du domaine public, ce qui donne au client la liberté d’opérer dans ces régions de séquence.



Comment les enzymes sont sélectionnées pour le criblage
S’appuyant sur les puissantes ressources microbiennes de la nature, notamment des échantillons provenant d’environnements tels que le sol, les sources thermales, l’eau de mer, les sites miniers et d’autres conditions extrêmes, la base de données bit-GEM contient un vaste éventail de séquences génétiques acquises par la plateforme de séquençage unicellulaire bitBiome. Cette méthode de séquençage assure une couverture plus profonde du génome, ce qui permet de découvrir la « matière noire » microbienne. Ces séquences ont peu de similitudes avec celles du domaine public, ce qui donne au client la liberté d’opérer dans ces régions de séquence. La sélection des 48 enzymes fournies dans ce kit s’est appuyée non seulement sur les capacités de synthèse d’ADN de Twist, mais aussi sur un effort bioinformatique collaboratif afin d’affiner les régions de séquence fonctionnelles.
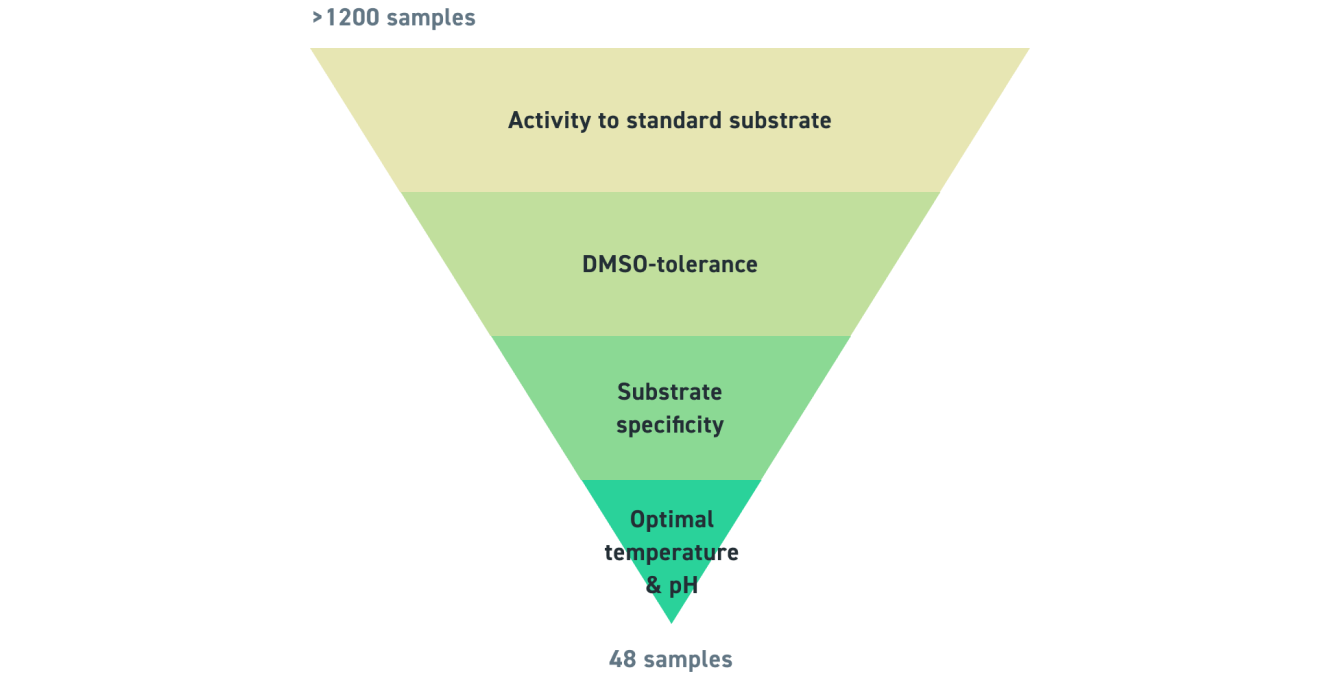
Comment les enzymes ont été sélectionnées pour notre kit
Un total de 369 transaminases actives a été identifié avec 19 substrats. 273 des enzymes se sont révélées actives dans 10 % de DMSO, avec 30 actives à 60 °C. Enfin, les enzymes ont été testées pour leur sensibilité de pH et leur sélectivité.
Critères de criblage des transaminases
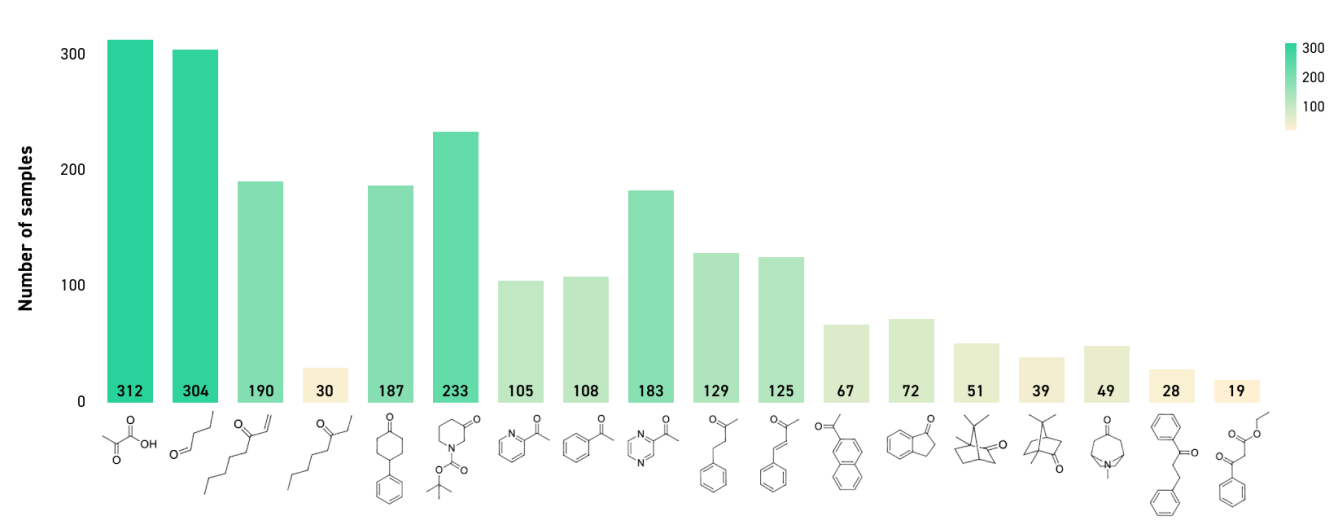
Vous trouverez ci-dessous une répartition des enzymes testées contre divers substrats dans les différentes conditions décrites précédemment (divers % de DMSO, solvants organiques, températures et niveaux de pH).
Comment les enzymes sont sélectionnées pour le criblage
S’appuyant sur les puissantes ressources microbiennes de la nature, notamment des échantillons provenant d’environnements tels que le sol, les sources thermales, l’eau de mer, les sites miniers et d’autres conditions extrêmes, la base de données bit-GEM contient un vaste éventail de séquences génétiques acquises par la plateforme de séquençage unicellulaire bitBiome. Cette méthode de séquençage assure une couverture plus profonde du génome, ce qui permet de découvrir la « matière noire » microbienne. Ces séquences ont peu de similitudes avec celles du domaine public, ce qui donne au client la liberté d’opérer dans ces régions de séquence. La sélection des 48 enzymes fournies dans ce kit s’est appuyée non seulement sur les capacités de synthèse d’ADN de Twist, mais aussi sur un effort bioinformatique collaboratif afin d’affiner les régions de séquence fonctionnelles.
Comment les enzymes ont été sélectionnées pour notre kit
Un total de 369 transaminases actives a été identifié avec 19 substrats. 273 des enzymes se sont révélées actives dans 10 % de DMSO, avec 30 actives à 60 °C. Enfin, les enzymes ont été testées pour leur sensibilité de pH et leur sélectivité.
Critères de criblage des transaminases

Vous trouverez ci-dessous une répartition des enzymes testées contre divers substrats dans les différentes conditions décrites précédemment (divers % de DMSO, solvants organiques, températures et niveaux de pH).

