Présentation des fragments de gènes multiplex Twist
Les fragments de gènes multiplex Twist sont des séquences de fragments de gènes à double brin d’une longueur maximale de 500 paires de bases qui sont livrées sous forme de pool au lieu d’un fragment par matrice de puits. Ce format groupé de fragments permet des applications de criblage à haut débit telles que l’édition primaire, le criblage fonctionnel extrêmement complexe basé sur CRISPR, l’ingénierie des peptides et des protéines, la recherche d’anticorps et les tests de rapporteurs massivement parallèles (MPRA), où le coût par fragment peut être un obstacle au nombre désiré de variants.
Les fragments de gènes multiplex sont synthétisés à partir de séquences définies par l’utilisateur dans des longueurs allant jusqu’à 500 paires de bases par gène. Ces séquences définies par l’utilisateur sont initialement synthétisées dans des pools d’oligonucléotides d’une longueur maximale de 500 nucléotides, puis amplifiées dans de l’ADN double brin et livrées sous forme de fragments de gènes multiplex qui ont été soigneusement analysés pour s’assurer que >90 % des gènes du pool sont de la bonne longueur. Ce produit est optimisé pour les applications de criblage rapide à haut débit.


Témoignages
Présentation des fragments de gènes multiplex Twist
Les fragments de gènes multiplex Twist sont des séquences de fragments de gènes à double brin d’une longueur maximale de 500 paires de bases qui sont livrées sous forme de pool au lieu d’un fragment par matrice de puits. Ce format groupé de fragments permet des applications de criblage à haut débit telles que l’édition primaire, le criblage fonctionnel extrêmement complexe basé sur CRISPR, l’ingénierie des peptides et des protéines, la recherche d’anticorps et les tests de rapporteurs massivement parallèles (MPRA), où le coût par fragment peut être un obstacle au nombre désiré de variants.
Les fragments de gènes multiplex sont synthétisés à partir de séquences définies par l’utilisateur dans des longueurs allant jusqu’à 500 paires de bases par gène. Ces séquences définies par l’utilisateur sont initialement synthétisées dans des pools d’oligonucléotides d’une longueur maximale de 500 nucléotides, puis amplifiées dans de l’ADN double brin et livrées sous forme de fragments de gènes multiplex qui ont été soigneusement analysés pour s’assurer que >90 % des gènes du pool sont de la bonne longueur. Ce produit est optimisé pour les applications de criblage rapide à haut débit.


Nous expédions en seulement 4 jours*
En savoir plus

Témoignages
Les pools de fragments de gènes multiplex Twist permettent une représentation complète de chaque séquence commandée, afin d’assurer un contrôle précis de la construction des variants pour un criblage plus ciblé et plus rationnel.
Un pool de fragments de gènes multiplex de variants à double ARNg de 500 pb montre une représentation complète des fragments avec des absences d’amplification minimales (Fig. 1), tout comme un pool de variants à triple ARNg de 450 pb (Fig. 2). Enfin, un pool de fragments de gènes multiplex avec des variants contenant des GC sur une large gamme, y compris des fragments atteignant 80% de GC, montre une forte représentation et un biais minimal (Fig. 3).
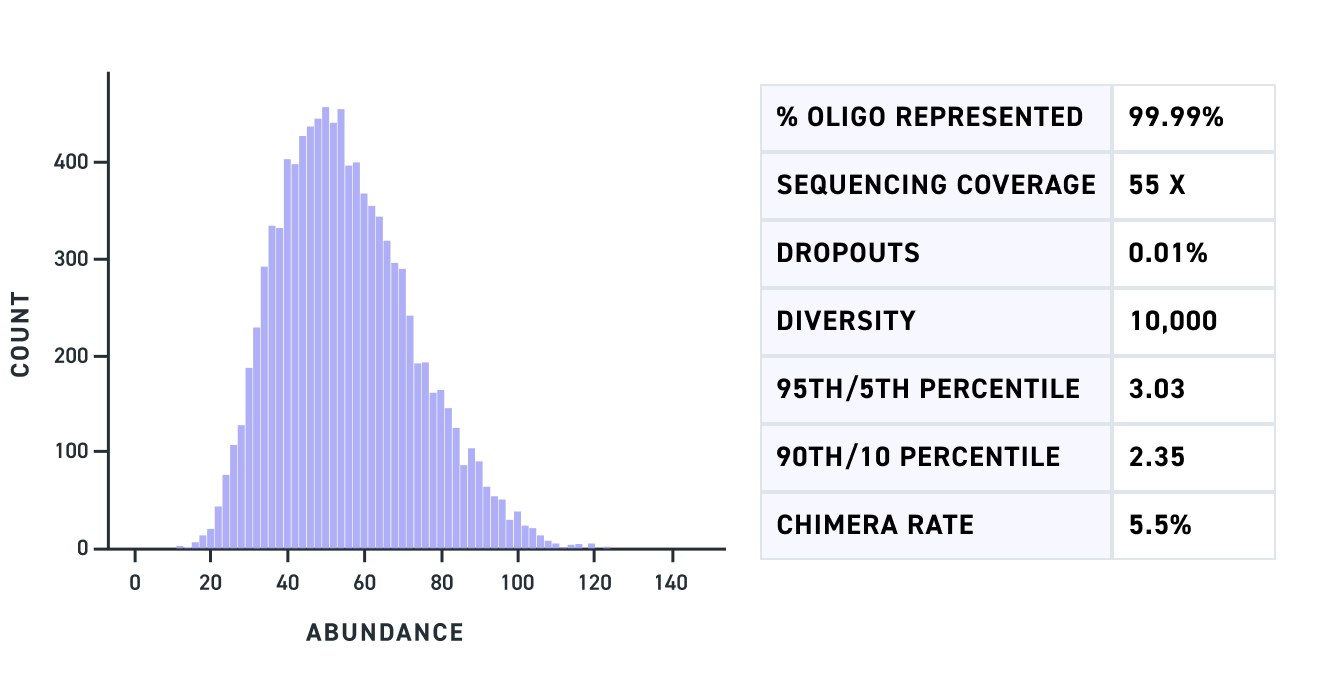
Figure 1. Graphique du nombre de lectures de chacun des variants à double ARNg cartographiés (axe des abscisses) pour un pool amplifié de fragments de gènes multiplex de 500 pb.
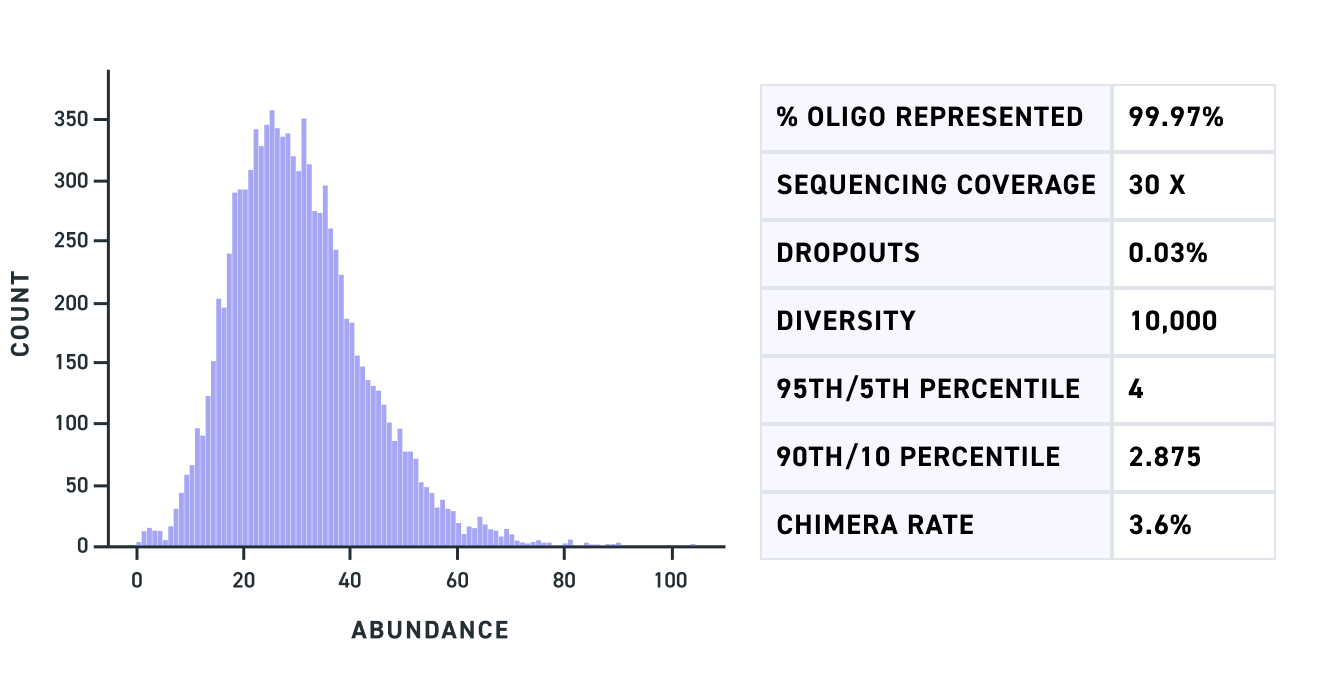
Figure 2. Graphique du nombre de lectures de chacun des variants à triple ARNg cartographiés (axe des abscisses) pour un pool amplifié de fragments de gènes multiplex de 450 pb.
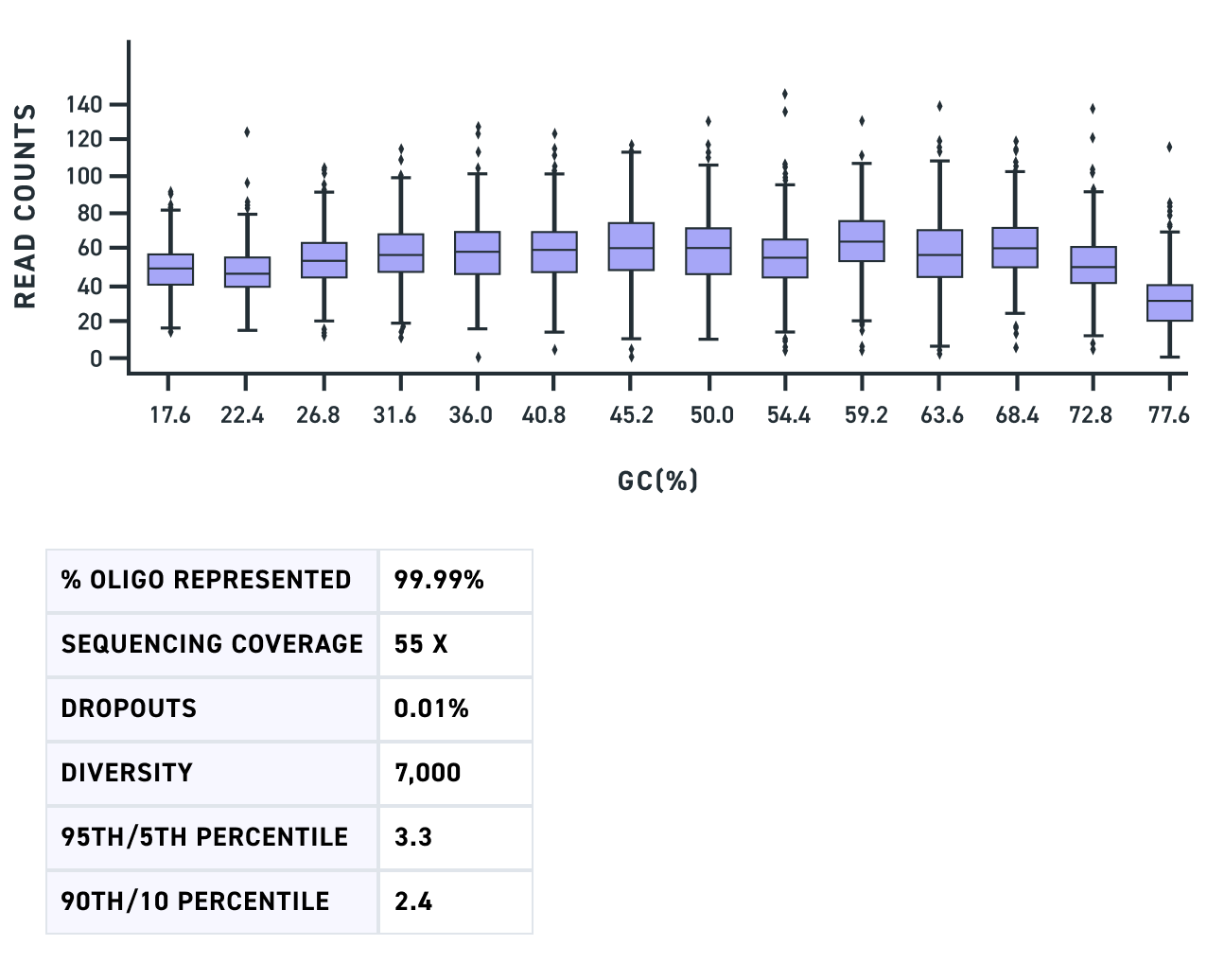
Figure 3. Création de diagrammes GC d’un pool amplifié de fragments de gènes multiplex d’une longueur de 500 pb, montrant un biais minimal parmi une large gamme de teneur en GC allant jusqu’à 80 %. L’axe des abscisses représente les catégories de 14 groupes de séquences ayant des teneurs en GC différentes. L’axe des ordonnées représente le nombre de lectures.
Les pools de fragments de gènes multiplex Twist permettent une représentation complète de chaque séquence commandée, afin d’assurer un contrôle précis de la construction des variants pour un criblage plus ciblé et plus rationnel.
Un pool de fragments de gènes multiplex de variants à double ARNg de 500 pb montre une représentation complète des fragments avec des absences d’amplification minimales (Fig. 1), tout comme un pool de variants à triple ARNg de 450 pb (Fig. 2). Enfin, un pool de fragments de gènes multiplex avec des variants contenant des GC sur une large gamme, y compris des fragments atteignant 80% de GC, montre une forte représentation et un biais minimal (Fig. 3).

Figure 1. Graphique du nombre de lectures de chacun des variants à double ARNg cartographiés (axe des abscisses) pour un pool amplifié de fragments de gènes multiplex de 500 pb.

Figure 2. Graphique du nombre de lectures de chacun des variants à triple ARNg cartographiés (axe des abscisses) pour un pool amplifié de fragments de gènes multiplex de 450 pb.

Figure 3. Création de diagrammes GC d’un pool amplifié de fragments de gènes multiplex d’une longueur de 500 pb, montrant un biais minimal parmi une large gamme de teneur en GC allant jusqu’à 80 %. L’axe des abscisses représente les catégories de 14 groupes de séquences ayant des teneurs en GC différentes. L’axe des ordonnées représente le nombre de lectures.
Rendement
Minimum de 200 ng d’ADNdb amplifiéDélai de traitement
8 à 12 jours ouvrablesFormat de livraison
ADNdb déshydraté, regroupé dans un tube de 2 mlLongueur
301 à 500 pbTaille du pool
Pas de minimum, pas de maximumUniformité
90 % des séquences se situent à ~3x de la moyenne*Contrôle de la qualité (CQ)
Analyse des fragments pour s’assurer que >90 % des gènes d’un pool ont la bonne longueur*Taux d’erreur
1:2000 nt (à partir de la synthèse du pool original, avant amplification)*Conditions générales : Pour les fragments de gènes multiplex, le délai de traitement standard est de 8 à 12 jours ouvrables. Cela varie selon la complexité et la longueur de la séquence. La longueur des fragments de gènes multiplex peut atteindre 500 paires de bases, avec jusqu’à 460 pb allouées pour la région variable et un minimum de 40 pb conservées pour la région amorce. Les tests de contrôle de la qualité garantissent que >90 % des séquences de gènes d’un pool seront de la longueur définie par l’utilisateur.
Réservé à l’utilisation dans le cadre de la recherche. Non destiné à être utilisé dans des procédures de diagnostic.
Rendement
Minimum de 200 ng d’ADNdb amplifiéDélai de traitement
8 à 12 jours ouvrablesFormat de livraison
ADNdb déshydraté, regroupé dans un tube de 2 mlLongueur
301 à 500 pbTaille du pool
Pas de minimum, pas de maximumUniformité
90 % des séquences se situent à ~3x de la moyenne*Contrôle de la qualité (CQ)
Analyse des fragments pour s’assurer que >90 % des gènes d’un pool ont la bonne longueur*Taux d’erreur
1:2000 nt (à partir de la synthèse du pool original, avant amplification)*Conditions générales : Pour les fragments de gènes multiplex, le délai de traitement standard est de 8 à 12 jours ouvrables. Cela varie selon la complexité et la longueur de la séquence. La longueur des fragments de gènes multiplex peut atteindre 500 paires de bases, avec jusqu’à 460 pb allouées pour la région variable et un minimum de 40 pb conservées pour la région amorce. Les tests de contrôle de la qualité garantissent que >90 % des séquences de gènes d’un pool seront de la longueur définie par l’utilisateur.
Réservé à l’utilisation dans le cadre de la recherche. Non destiné à être utilisé dans des procédures de diagnostic.
Commençons
- Étape 1 : Remplir le formulaire de contact sur cette page
- Étape 2 : Télécharger le formulaire de demande de FGM et remplir tous les champs du formulaire pour la conception de vos FGM.
- Étape 3 : Chargez le formulaire de demande complété dans l’onglet « Envoyer fichier » de cette page.
En cas de questions, ne pas hésiter à nous envoyer un e-mail à customersupport@twistbioscience.com.
Prêt à envoyer ?
- Étape 1 : Charger le formulaire de demande complété
- Étape 2 : Les experts de l’équipe chargée des FGM examinent votre projet.
- Étape 3 : L’équipe chargée des FGM valide le projet et vous contacte pour vous proposer un devis.
- Étape 4 : Une fois le devis accepté par le client, l’équipe chargée des FGM envoie le projet à l’équipe de production.
En cas de questions, ne pas hésiter à nous envoyer un e-mail à customersupport@twistbioscience.com.
Commençons
- Étape 1 : Remplir le formulaire de contact sur cette page
- Étape 2 : Télécharger le formulaire de demande de FGM et remplir tous les champs du formulaire pour la conception de vos FGM.
- Étape 3 : Chargez le formulaire de demande complété dans l’onglet « Envoyer fichier » de cette page.
En cas de questions, ne pas hésiter à nous envoyer un e-mail à customersupport@twistbioscience.com.
Prêt à envoyer ?
- Étape 1 : Charger le formulaire de demande complété
- Étape 2 : Les experts de l’équipe chargée des FGM examinent votre projet.
- Étape 3 : L’équipe chargée des FGM valide le projet et vous contacte pour vous proposer un devis.
- Étape 4 : Une fois le devis accepté par le client, l’équipe chargée des FGM envoie le projet à l’équipe de production.
En cas de questions, ne pas hésiter à nous envoyer un e-mail à customersupport@twistbioscience.com.
Guide/conseils
Lignes directrices pour la conception de fragments de gènes multiplex Twist.
Guide/conseils
Lignes directrices pour la conception de fragments de gènes multiplex Twist.