Fragmentación enzimática mejorada
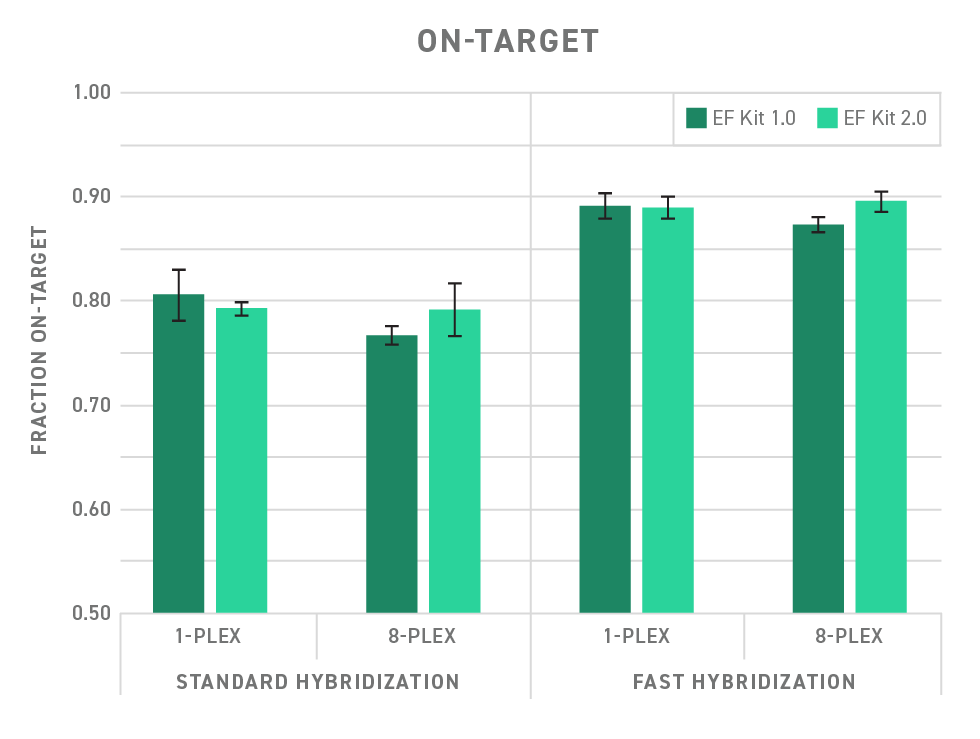
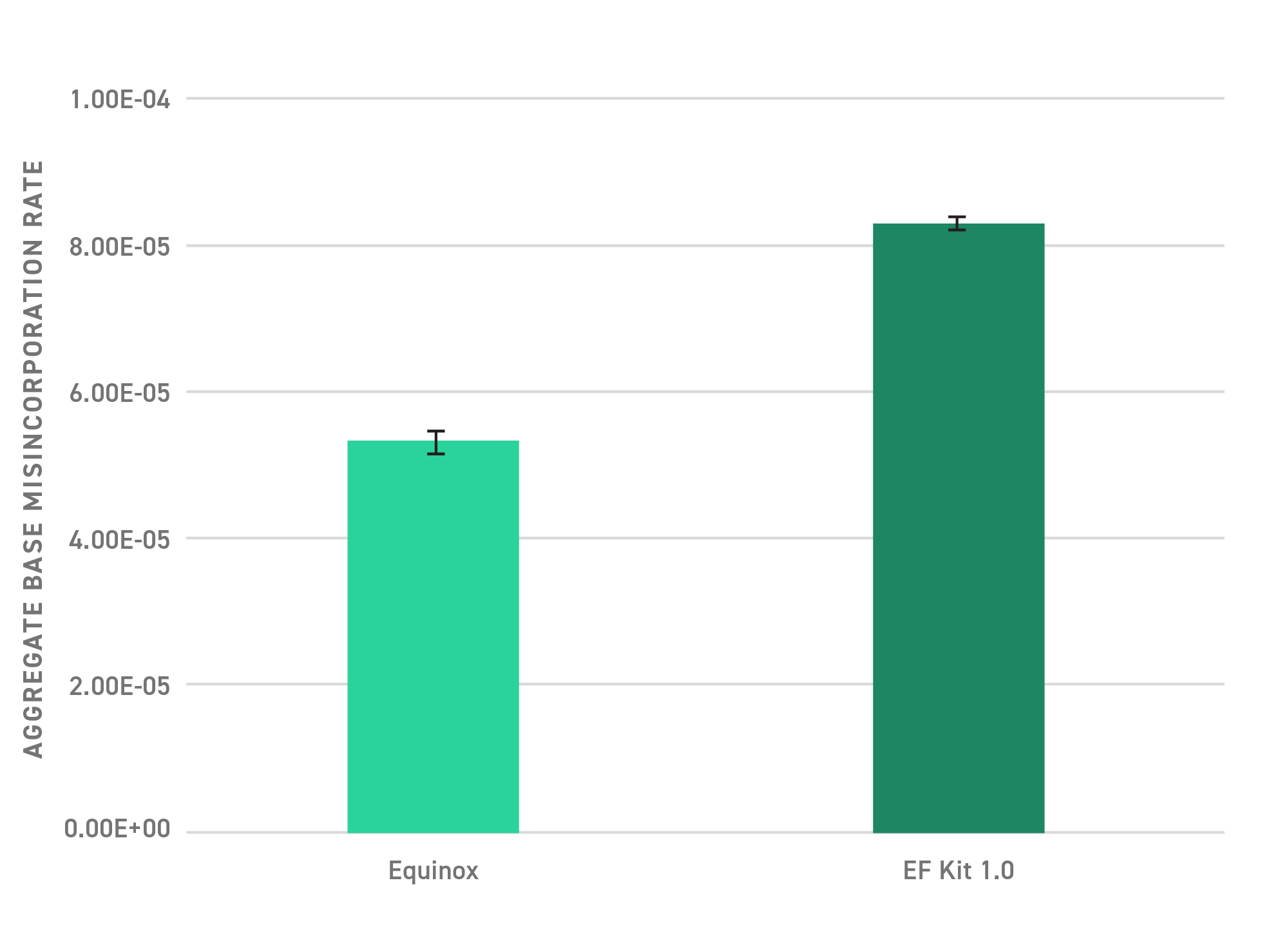
El kit de preparación de bibliotecas de fragmentación enzimática (EF) Twist 2.0 está diseñado para ayudarle a conseguir una secuenciación más eficiente y unos resultados de la secuenciación de última generación (NGS) más precisos. En comparación con el kit de preparación de bibliotecas de fragmentación enzimática Twist 1.0, este kit actualizado presenta varias ventajas. Además de los nuevos módulos de fragmentación y unión, el kit actualizado ahora incluye la mezcla de amplificación de preparación de bibliotecas Equinox. Esta formulación enzimática de arranque en caliente presenta una menor tasa de error* y una mayor eficiencia a volúmenes de entrada bajos, lo que le hace ideal para muestras como las de tejidos fijados en formol e incluidos en parafina, en las que se necesitan una eficiencia y un rendimiento altos.
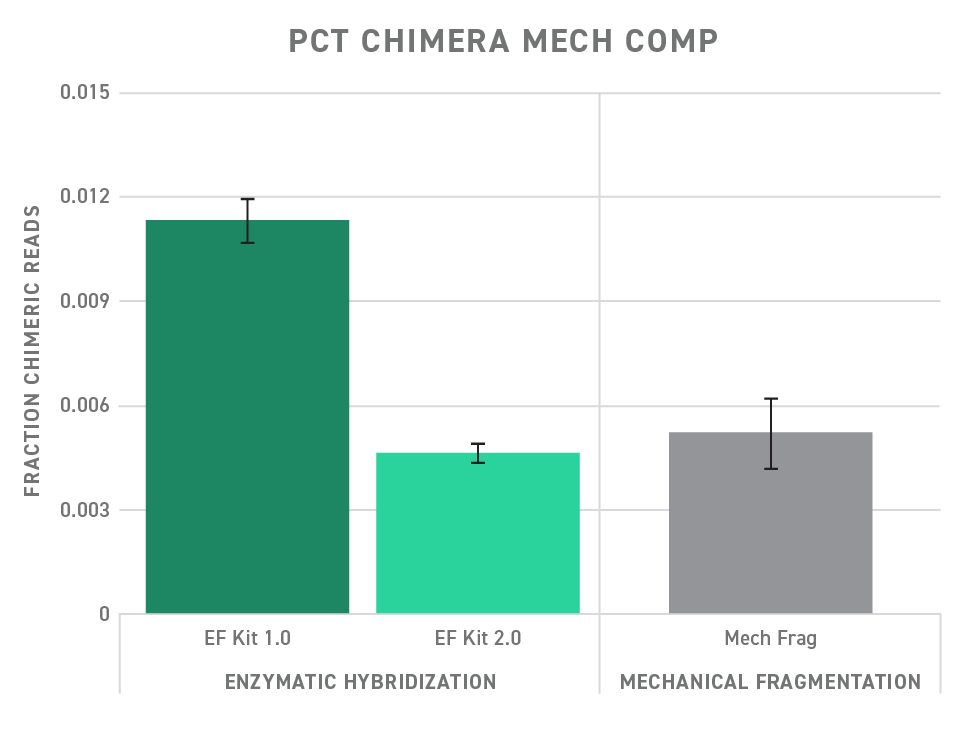
El flujo de trabajo optimizado del kit combina los pasos de preparación de bibliotecas en una reacción en un solo tubo que, junto con una tasa de quimeras baja y el flujo de trabajo de enriquecimiento del objetivo altamente uniforme de Twist, permite una preparación de bibliotecas y un resultado de secuenciación de mejor calidad.
* Con respecto a la fragmentación enzimática Twist 1.0
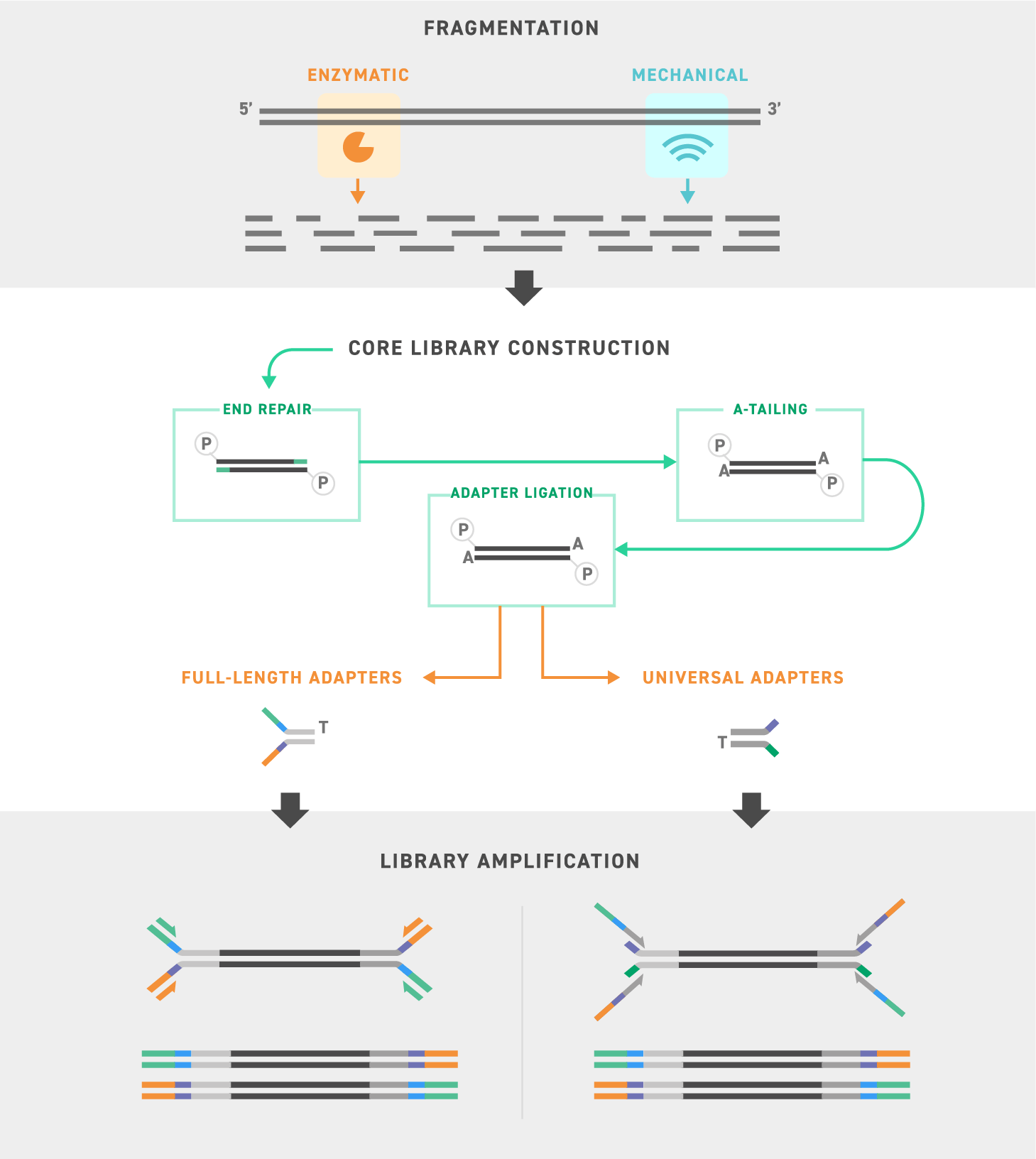
Flujo de trabajo de preparación de bibliotecas
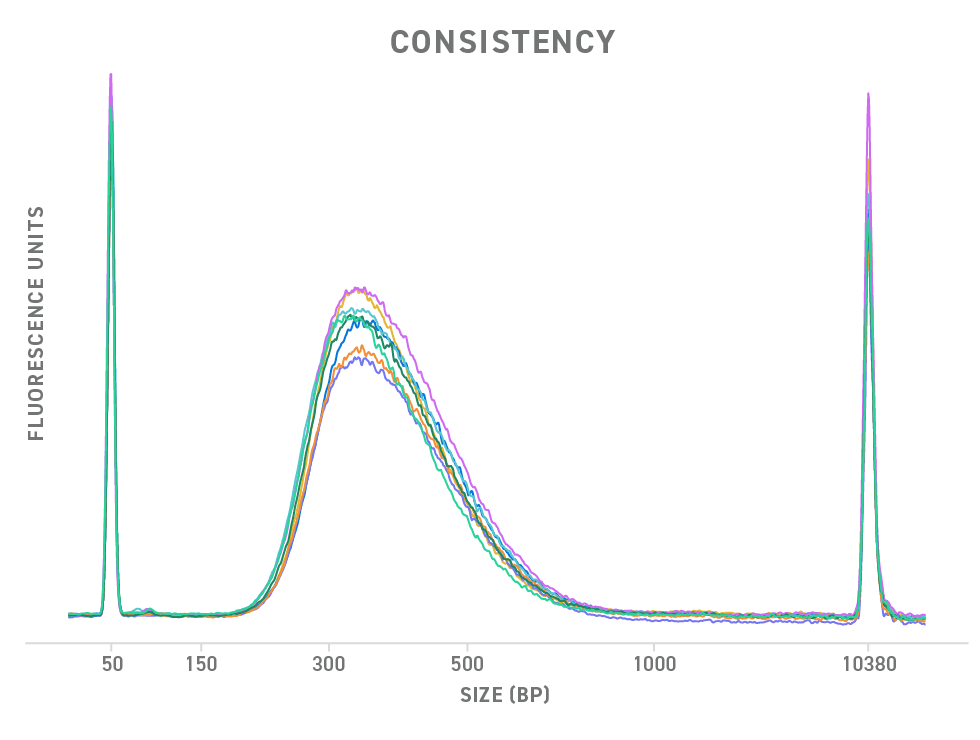
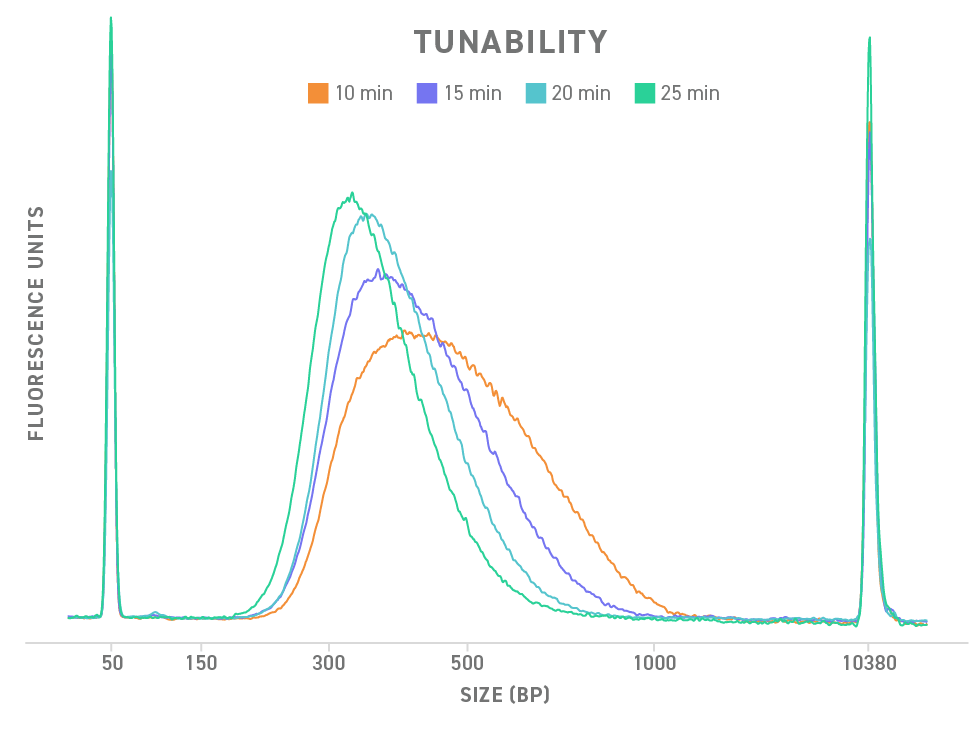
El kit de preparación de bibliotecas de fragmentación enzimática Twist 2.0 incluye los reactivos necesarios para la reparación de extremos, la adición de AMP-d al extremo 3’, la unión del adaptador y la amplificación de la biblioteca. Este kit también incorpora las enzimas para la fragmentación de muestras de ADNg y permite tamaños de inserción ajustables. Después de la construcción de la biblioteca principal, se pueden utilizar adaptadores universales o de longitud completa para satisfacer las necesidades de su aplicación.
Garantice la precisión de la secuenciación de exomas completos mediante el seguimiento de las muestras desde la sangre completa. El ID de muestras humanas Twist utiliza la preparación de bibliotecas Twist y el PCR multiplexado establecidos para identificar y realizar un seguimiento de las muestras con el fin de evitar la mezcla de las muestras y datos incorrectos.
Obtener unos buenos datos de secuenciación de última generación (NGS) de incluso las muestras más complejas requiere los mejores reactivos durante toda la secuenciación.
Lea nuestro blog para descubrir cómo el kit de preparación de bibliotecas de fragmentación enzimática Twist 2.0 está formulado para garantizar una calidad excepcional de las bibliotecas y así obtener unos datos de NGS excepcionales.

Cuando se trabaja con muestras degradadas difíciles, como las de tejidos fijados en formol e incluidos en parafina, la fragmentación mecánica puede ser una opción viable. Y, al igual que la fragmentación enzimática, los reactivos de calidad son clave para obtener unos resultados de calidad. Obtenga más información sobre nuestro kit de preparación de bibliotecas de fragmentación mecánica.