Identification des facteurs de virulence du coronavirus pour le développement de vaccins à ARN avec la méthode fate-seq basée sur le pool d’oligonucléotides de Twist Bioscience.
Le monde continue de subir l’impact de la pandémie mondiale causée par le SARS-CoV-2, le virus à l’origine du COVID-19. Elle a incontestablement bouleversé la vie de la planète ; mais à mesure que nous continuons à nous battre, de nouveaux signes de progrès nous donnent de l’espoir. La communauté des chercheurs a été particulièrement touchée et les scientifiques se sont rapidement mobilisés pour aborder et répondre aux questions relatives à la manière dont ce virus infecte, transmet et mute. L’un de ces groupes, présenté ici, montre comment les pool d’oligonucléotides de Twist Bioscience peuvent permettre des innovations passionnantes ainsi qu’une meilleure compréhension de la façon dont le SARS-CoV-2 déjoue notre système immunitaire.
To cause infection, COVID-19 must enter a host cell and release its RNA genome. Virulence is established via replication of this genome, and production of new viral particles through the host cell’s expression of viral genes (Boopathi et al., 2020). Our cells have a number of innate mechanisms that restrict viral replication including RNA degrading proteins and genetic checkpoints alerting the immune system that “this cell has been infected.” These systems work in tandem with the adaptive immune system to halt virulence.
Pourtant, dans cette bataille pour la survie, certains virus ont développé une résistance à nos systèmes immunitaires innés. Dans le cas de certains virus à ARN, la stabilité de leur génome les protège des enzymes de dégradation de l’ARN. La stabilité de l’ARN est accrue lorsqu’il forme des structures secondaires complexes par appariement de bases avec lui-même. Ces structures secondaires peuvent être insensibles à la défense antivirale de la cellule hôte, voire la bloquer activement, ce qui permet au virus de se répliquer sans être détecté.
Les traitements qui ciblent la stabilité de l’ARN viral à évolution rapide peuvent permettre au système immunitaire de l’hôte de dégrader et d’éliminer efficacement l’infection virale. Dans un article récemment publié dans Biochemical and Biophysical Research Communications, Wakida et al. ont développé une méthode pour explorer de manière exhaustive les séquences d’ARN dérivées de virus à ARN et à ADN qui contribuent à la stabilité de l’ARN dans les cellules hôtes. L’auteur principal, le professeur Nobuyoshi Akimitsu, décrit cette étude comme une étape vers l’amélioration de la médecine fondée sur l’ARN.
L’ARNm est instable et sa traduction dans la cellule est souvent médiocre. En étudiant les éléments naturels qui renforcent la stabilité et la structure de l’ARNm, nous espérons améliorer la fonctionnalité de l’ARNm en tant que médicament », explique le professeur Akimitsu.
La méthode développée dans cette étude est appelée « Fate-seq ». La méthode Fate-seq repose sur le principe que les séquences d’ARN stables sont dégradées dans une cellule de mammifère beaucoup plus lentement que les séquences instables. En attachant des séquences d’ARN d’intérêt à une molécule signal comme la protéine fluorescente verte et en les introduisant dans les cellules, la dégradation peut être directement mesurée. Les cellules qui présentent le signal fluorescent le plus fort au fil du temps contiennent des marqueurs attachés aux structures d’ARN les plus stables. Le séquençage des cellules présentant le signal le plus fort permet ensuite de cartographier avec précision les séquences d’ARN les plus stables.
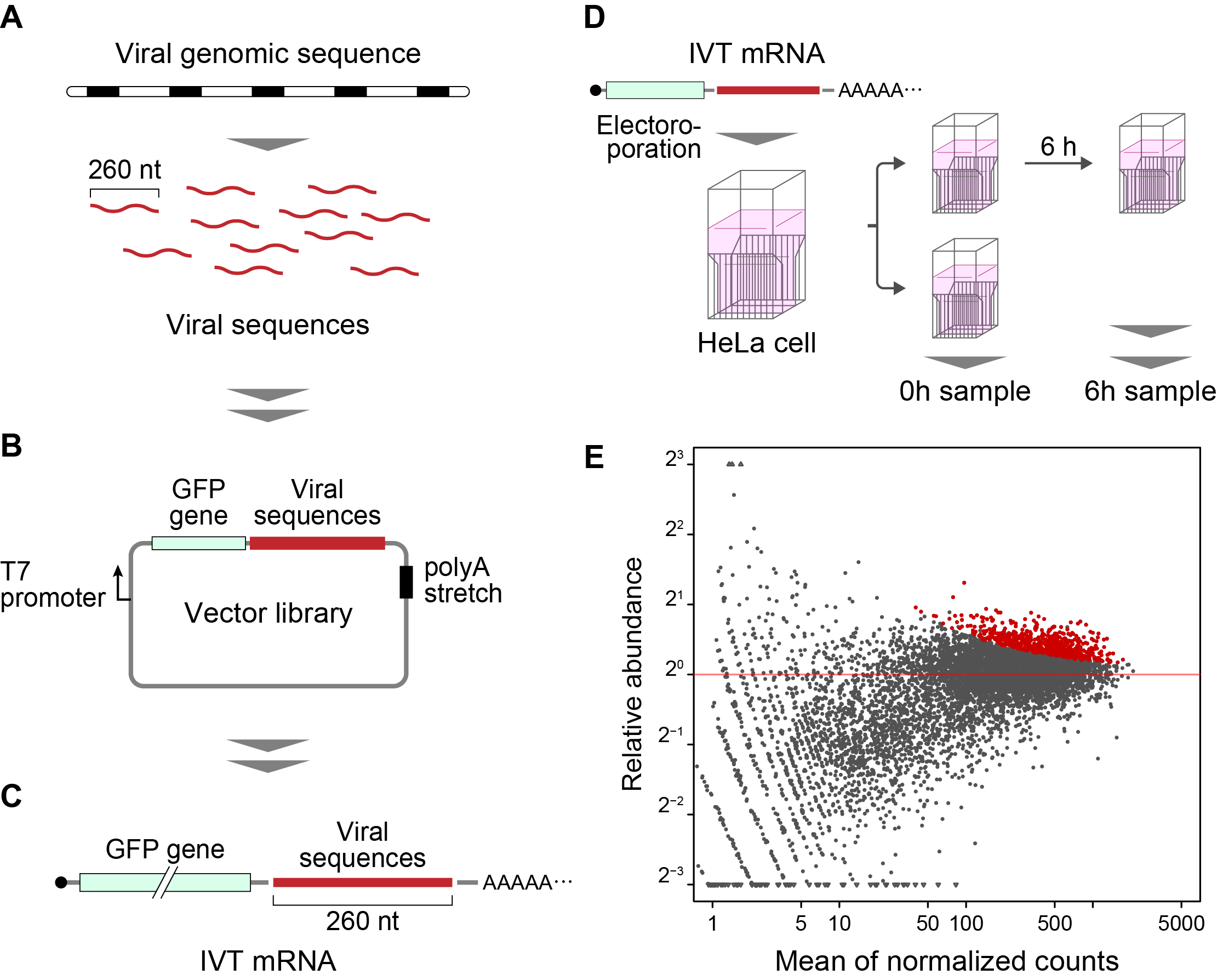
Wakida et al. ont utilisé les pools d’oligonucléotide de Twist Bioscience pour analyser le génome de 26 virus différents. Chaque génome viral a été divisé en fragments de 260 nucléotides (nt), avec un chevauchement de 200 nt. Le professeur Akimitsu a souligné l’importance des oligonucléotides de haute qualité dans cette étude.
“When doing a genome scan like Fate-seq, you need to be sure you are covering as much of the genome as possible, as evenly as possible. Because of the good uniformity and low error of Twist Oligo Pools, we can get even coverage across the viral genomes, and know we do not miss any potentially critical data.” Twist Oligo Pools can be synthesized up to 300 nucleotides, with an industry leading error rate of 1:2000.
Dans cette étude, Fate-seq a mis en évidence deux régions hautement conservées de stabilisation de l’ARN (COV001 et COV002) dans le coronavirus lié au syndrome respiratoire aigu sévère (SARS-CoV) et le SARS-CoV-2. Les résultats de Wakida et al. ont identifié un changement dans la structure secondaire de COV001 dans SARS-CoV-2 qui n’a pas été détecté pour SARS-CoV. Les auteurs interprètent que « cette différence potentielle dans la structure secondaire peut contribuer à la virulence du SARS-CoV-2 Les séquences identifiées, qui pourraient affecter la stabilité de l’ARN viral et les protéines de liaison à l’ARN se liant à ces séquences, peuvent constituer des cibles possibles pour le traitement des maladies infectieuses virales, dont le COVID-19. »
Development of COVID-19 vaccines are currently in high-demand, many consisting of messenger RNA to activate the host immune system (Callaway, 2020). New technologies like Fate-seq to identify stabilizing molecules will offer significant benefits to the RNA vaccine development field, allowing more stable and efficacious vaccines to be produced. Moreover, Prof. Akimitsu reiterated “RNA stability is important for the viral life cycle. In theory, by inhibiting stability, we can reduce viral proliferation and virulence.” Fate-seq is therefore an expeditious and promising approach to spotlight critical genomic regions for further characterization as targets for antiviral therapeutics.
Photo de couverture par Fusion Medical Animation sur Unsplash
Qu’en pensez-vous ?
J’aime
bien
Je n’aime pas
J’aime beaucoup
Je suis surpris(e)
C’est intéressant
