Identificación de los factores de virulencia del coronavirus para el desarrollo de la vacuna de ARN con los grupos de oligonucleótidos Twist
El mundo sigue notando el impacto de la pandemia global provocada por el SARS-CoV-2, el virus que provoca la COVID-19. No hay duda de que esta ha cambiado la vida en el planeta, pero, a medida que seguimos luchando, las nuevas señales de progreso nos dan esperanzas. Esto ha afectado especialmente a la comunidad investigadora y los científicos se han movilizado rápidamente para resolver esta situación y responder a preguntas relacionadas con la forma en la que el virus infecta, se transmite y muta. Uno de estos grupos, perfilado aquí, muestra cómo pueden usarse los grupos de oligonucleótidos Twist para conseguir innovaciones emocionantes, lo que nos permite entender mejor como el SARS-CoV-2 secuestra nuestro sistema inmunológico.
To cause infection, COVID-19 must enter a host cell and release its RNA genome. Virulence is established via replication of this genome, and production of new viral particles through the host cell’s expression of viral genes (Boopathi et al., 2020). Our cells have a number of innate mechanisms that restrict viral replication including RNA degrading proteins and genetic checkpoints alerting the immune system that “this cell has been infected.” These systems work in tandem with the adaptive immune system to halt virulence.
Aun así, en la carrera armamentista por la supervivencia, algunos virus han desarrollado resistencia a nuestros sistemas inmunológicos innatos. En algunos virus de ARN, la estabilidad de su genoma les proporciona protección frente a las enzimas degradadoras de ARN. La estabilidad del ARN aumenta cuando forma estructuras secundarias complejas a través del emparejamiento de pares consigo mismo. Estas estructuras secundarias pueden ser no susceptibles a la orden antiviral de la célula huésped o incluso bloquearla activamente, lo que permite que el virus se replique de forma desapercibida.
Los tratamientos terapéuticos que tienen como objetivo la estabilidad del ARN viral de evolución rápida pueden permitir que el sistema inmunológico huésped degrade y elimine la infección viral de forma efectiva. En un artículo de publicación reciente, Biochemical and Biophysical Research Communications, Wakida et al. desarrollaron un método para interrogar de forma expansiva las secuencias de ARN derivadas de virus de ARN y ARN que contribuyen a la estabilidad del ARN en las células huésped. El autor experimentado, el profesor Nobuyoshi Akimitsu, describe este estudio como un paso hacia delante en la mejora de la medicina del ARN.
“El ARN mensajero es inestable y la traducción en la célula suele ser mala. Esperamos mejorar la funcionalidad del ARN mensajero como una medicina a través del estudio de los elementos naturales que mejoran la estabilidad y la estructura del ARN mensajero”, explicó el profesor Akimitsu.
El método desarrollado en este estudio se denomina “Fate-seq”. La Fate-seq trabaja sobre el principio de que las secuencias estables de ARN se degradarán en una célula de mamífero mucho más despacio que las secuencias inestables. Acoplando secuencias de ARN de interés a una molécula de señalización como una proteína fluorescente verde e introduciéndolas en células, se puede medir la degradación directamente. Las células que muestren la señal fluorescente más fuerte con el paso del tiempo contendrán indicadores acoplados a las estructuras de ARN más estables. Entonces, la secuenciación de las células con la señal más fuerte permite el mapeado preciso de secuencias de ARN altamente estables.
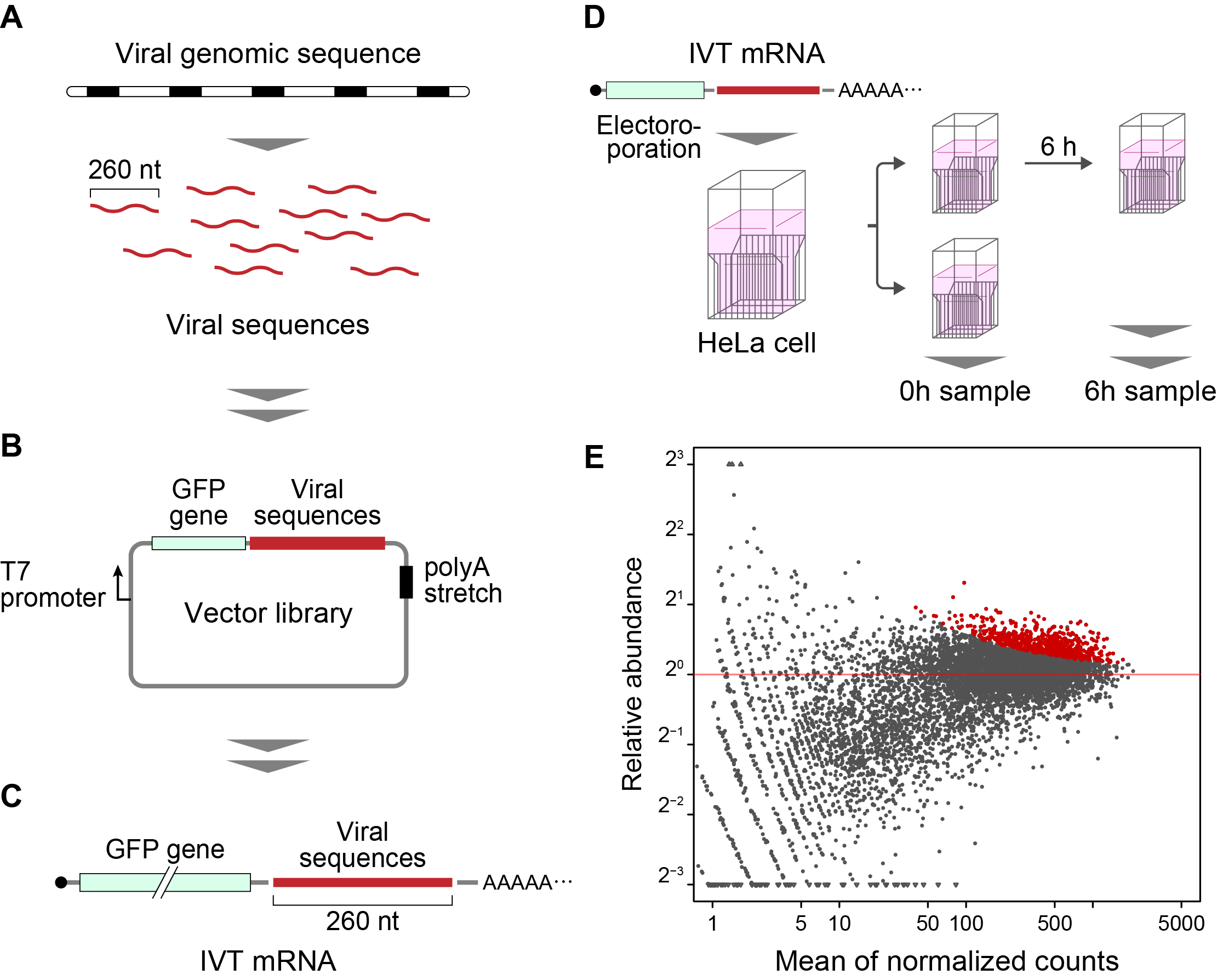
Wakida et al. utilizaron los grupos de oligonucleótidos Twist Bioscience para escanear el genoma de 26 virus diferentes. Cada genoma viral se dividió en trozos de 260 nucleótidos (nt) con un solapamiento de 200 nt. El profesor Akimitsu describió la importancia de utilizar oligonucleótidos de alta calidad en el estudio.
“When doing a genome scan like Fate-seq, you need to be sure you are covering as much of the genome as possible, as evenly as possible. Because of the good uniformity and low error of Twist Oligo Pools, we can get even coverage across the viral genomes, and know we do not miss any potentially critical data.” Twist Oligo Pools can be synthesized up to 300 nucleotides, with an industry leading error rate of 1:2000.
En el estudio, Fate-seq descubrió dos regiones estabilizadoras de ARN altamente conservado (COV001 y COV002) en el coronavirus (SARS-CoV) relacionado con el síndrome respiratorio agudo grave y el SARS-CoV-2. Los resultados de Wakida et al. determinaron un cambio en la estructura secundaria de la COV001 en el SARS-CoV-2 que no se infirió en el SARS-CoV. Los autores interpretan que “esta diferencia potencial en la estructura secundaria puede contribuir a la virulencia del SARS-CoV-2. Las secuencias identificadas, que podrían afectar a la estabilidad del ARN viral y las proteínas de unión con el ARN que se unen a estas secuencias, pueden ofrecer objetivos para el tratamiento de enfermedades infecciosas virales, incluida la COVID-19”.
Development of COVID-19 vaccines are currently in high-demand, many consisting of messenger RNA to activate the host immune system (Callaway, 2020). New technologies like Fate-seq to identify stabilizing molecules will offer significant benefits to the RNA vaccine development field, allowing more stable and efficacious vaccines to be produced. Moreover, Prof. Akimitsu reiterated “RNA stability is important for the viral life cycle. In theory, by inhibiting stability, we can reduce viral proliferation and virulence.” Fate-seq is therefore an expeditious and promising approach to spotlight critical genomic regions for further characterization as targets for antiviral therapeutics.
Foto de cubierta de Fusion Medical Animation en Unsplash
¿Qué piensa?
Me gusta
No me gusta
Me encanta
Me asombra
Me interesa
